11.4: Síntese de Proteínas (Tradução)
- Page ID
- 181328
Objetivos de
- Descreva o código genético e explique por que ele é considerado quase universal
- Explicar o processo de tradução e as funções da maquinaria molecular da tradução
- Compare a tradução em eucariotos e procariontes
A síntese de proteínas consome mais energia de uma célula do que qualquer outro processo metabólico. Por sua vez, as proteínas representam mais massa do que qualquer outra macromolécula de organismos vivos. Eles realizam praticamente todas as funções de uma célula, servindo como elementos funcionais (por exemplo, enzimas) e estruturais. O processo de tradução, ou síntese de proteínas, a segunda parte da expressão gênica, envolve a decodificação por um ribossomo de uma mensagem de mRNA em um produto polipeptídico.
O Código Genético
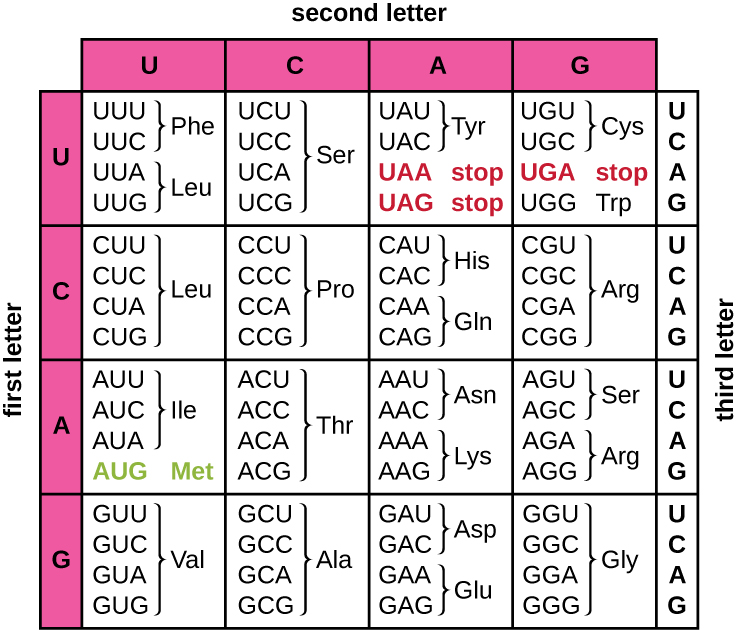
A tradução do modelo de mRNA converte informações genéticas baseadas em nucleotídeos na “linguagem” dos aminoácidos para criar um produto proteico. Uma sequência proteica consiste em 20 aminoácidos de ocorrência comum. Cada aminoácido é definido dentro do mRNA por um trigêmeo de nucleotídeos chamado códon. A relação entre um códon de mRNA e seu aminoácido correspondente é chamada de código genético.
O código de três nucleotídeos significa que há um total de 64 combinações possíveis (4 3, com quatro nucleotídeos diferentes possíveis em cada uma das três posições diferentes dentro do códon). Esse número é maior do que o número de aminoácidos e um determinado aminoácido é codificado por mais de um códon (Figura\(\PageIndex{1}\)). Essa redundância no código genético é chamada de degeneração. Normalmente, enquanto as duas primeiras posições em um códon são importantes para determinar qual aminoácido será incorporado a um polipeptídeo em crescimento, a terceira posição, chamada de posição de oscilação, é menos crítica. Em alguns casos, se o nucleotídeo na terceira posição for alterado, o mesmo aminoácido ainda será incorporado.
Enquanto 61 dos 64 trigêmeos possíveis codificam aminoácidos, três dos 64 códons não codificam para um aminoácido; eles terminam a síntese protéica, liberando o polipeptídeo da máquina de tradução. Eles são chamados de códons de parada ou códons sem sentido. Outro códon, o AUG, também tem uma função especial. Além de especificar o aminoácido metionina, ele também normalmente serve como códon inicial para iniciar a tradução. O quadro de leitura, a forma como os nucleotídeos no mRNA são agrupados em códons, para tradução é definido pelo códon inicial AUG próximo à extremidade 5' do mRNA. Cada conjunto de três nucleotídeos após esse códon inicial é um códon na mensagem de mRNA.
O código genético é quase universal. Com algumas exceções, praticamente todas as espécies usam o mesmo código genético para a síntese de proteínas, o que é uma evidência poderosa de que toda a vida existente na Terra compartilha uma origem comum. No entanto, aminoácidos incomuns, como selenocisteína e pirrolisina, foram observados em arquéias e bactérias. No caso da selenocisteína, o códon usado é o UGA (normalmente um códon de parada). No entanto, o UGA pode codificar a selenocisteína usando uma estrutura de alça estaminal (conhecida como sequência de inserção de selenocisteína, ou elemento SECIS), que é encontrada na região não traduzida de 3' do mRNA. A pirrolisina usa um códon de parada diferente, o UAG. A incorporação da pirrolisina requer o gene pyLS e um RNA de transferência exclusivo (tRNA) com um anticódon CUA.
Exercício\(\PageIndex{1}\)
- Quantas bases existem em cada códon?
- Qual aminoácido é codificado pelo códon AAU?
- O que acontece quando um códon de parada é alcançado?
A maquinaria de síntese prote
Além do modelo de mRNA, muitas moléculas e macromoléculas contribuem para o processo de tradução. A composição de cada componente varia entre os táxons; por exemplo, os ribossomos podem consistir em diferentes números de RNAs ribossômicos (rRNAs) e polipeptídeos, dependendo do organismo. No entanto, as estruturas e funções gerais do maquinário de síntese de proteínas são comparáveis das bactérias às células humanas. A tradução requer a entrada de um modelo de mRNA, ribossomos, tRNAs e vários fatores enzimáticos.
Ribossomos
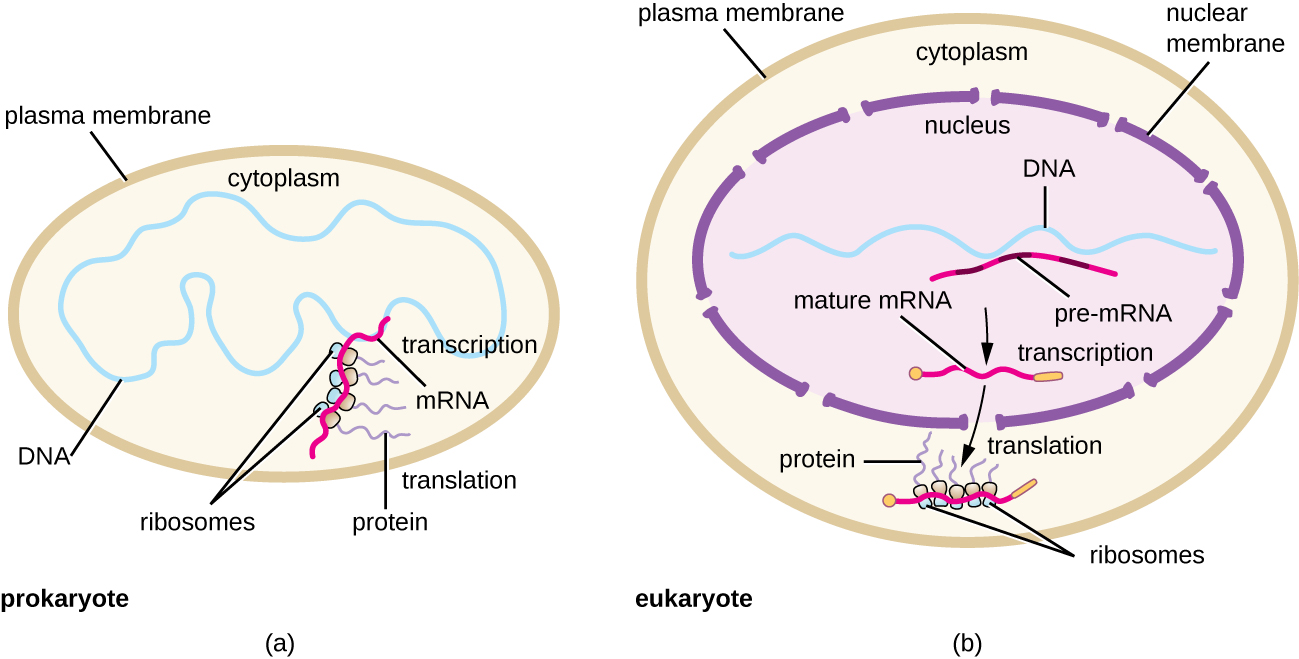
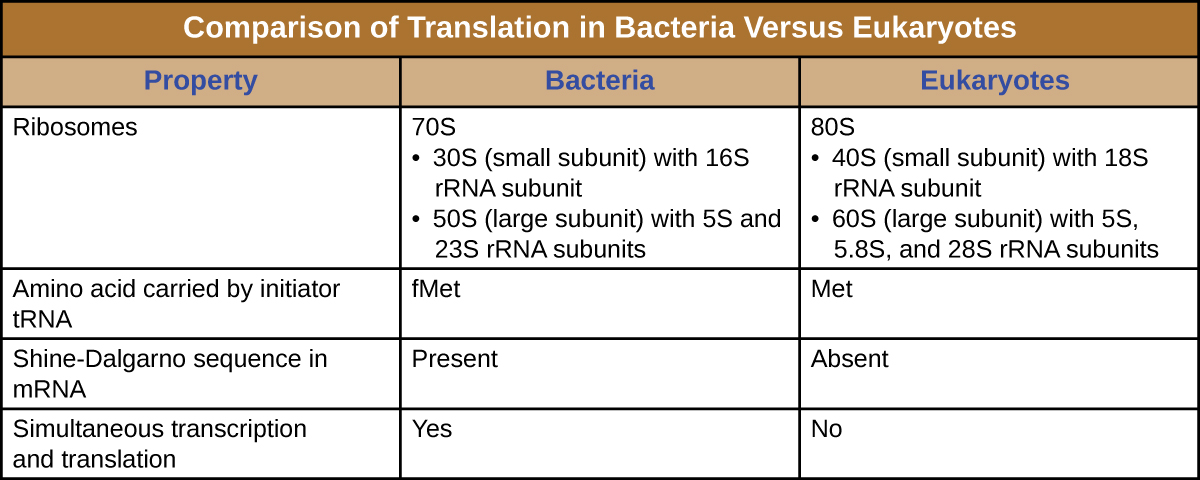
Um ribossomo é uma macromolécula complexa composta por rRNAs catalíticos (chamados ribozimas) e rRNAs estruturais, bem como muitos polipeptídeos distintos. Os rRNAs maduros compõem aproximadamente 50% de cada ribossomo. Os procariontes têm ribossomos 70S, enquanto os eucariotas têm ribossomos 80S no citoplasma e retículo endoplasmático rugoso e ribossomos 70S em mitocôndrias e cloroplastos. Os ribossomos se dissociam em subunidades grandes e pequenas quando não estão sintetizando proteínas e se reassociam durante o início da tradução. Em E. coli, a subunidade pequena é descrita como 30S (que contém a subunidade 16S rRNA), e a subunidade grande é 50S (que contém as subunidades de rRNA 5S e 23S), totalizando 70S (as unidades Svedberg não são aditivas). Os ribossomos eucariotas têm uma pequena subunidade 40S (que contém a subunidade 18S rRNA) e uma grande subunidade 60S (que contém as subunidades 5S, 5.8S e 28S rRNA), totalizando 80S. A subunidade pequena é responsável pela ligação ao modelo de mRNA, enquanto a subunidade grande se liga aos tRNAs (discutida na próxima subseção).
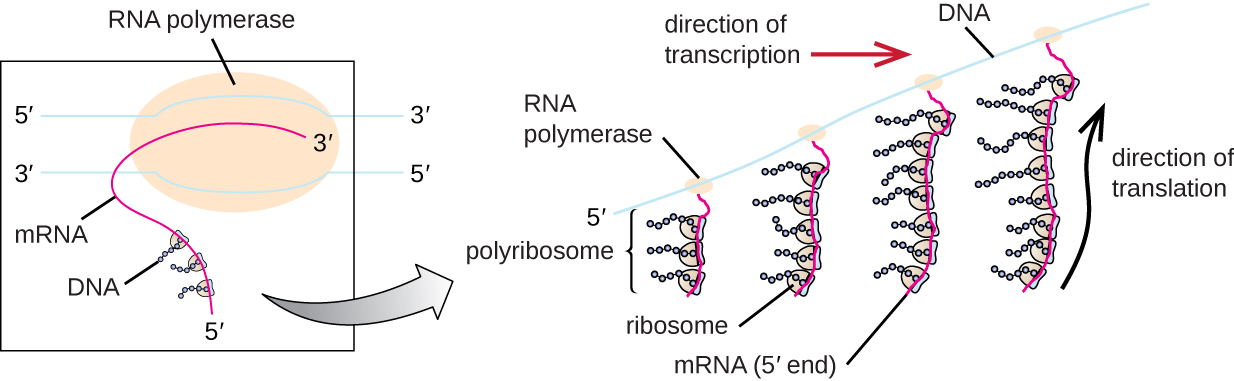
Cada molécula de mRNA é traduzida simultaneamente por muitos ribossomos, todos sintetizando proteínas na mesma direção: lendo o mRNA de 5' a 3' e sintetizando o polipeptídeo do terminal N para o terminal C. A estrutura completa contendo um mRNA com vários ribossomos associados é chamada de poliribossomo (ou polissomo). Tanto nas bactérias quanto nas arquéias, antes que ocorra o término da transcrição, cada transcrição codificadora de proteína já está sendo usada para iniciar a síntese de várias cópias do (s) polipeptídeo (s) codificado (s) porque os processos de transcrição e tradução podem ocorrer simultaneamente, formando poliribossomos (Figura \(\PageIndex{2}\)). A razão pela qual a transcrição e a tradução podem ocorrer simultaneamente é porque esses dois processos ocorrem na mesma direção de 5' a 3', ambos ocorrem no citoplasma da célula e porque o transcrito do RNA não é processado depois de transcrito. Isso permite que uma célula procariótica responda a um sinal ambiental que requer novas proteínas muito rapidamente. Em contraste, nas células eucarióticas, a transcrição e a tradução simultâneas não são possíveis. Embora os poliribossomos também se formem em eucariotos, eles não podem fazer isso até que a síntese de RNA esteja completa e a molécula de RNA tenha sido modificada e transportada para fora do núcleo.
RNAs de transferência
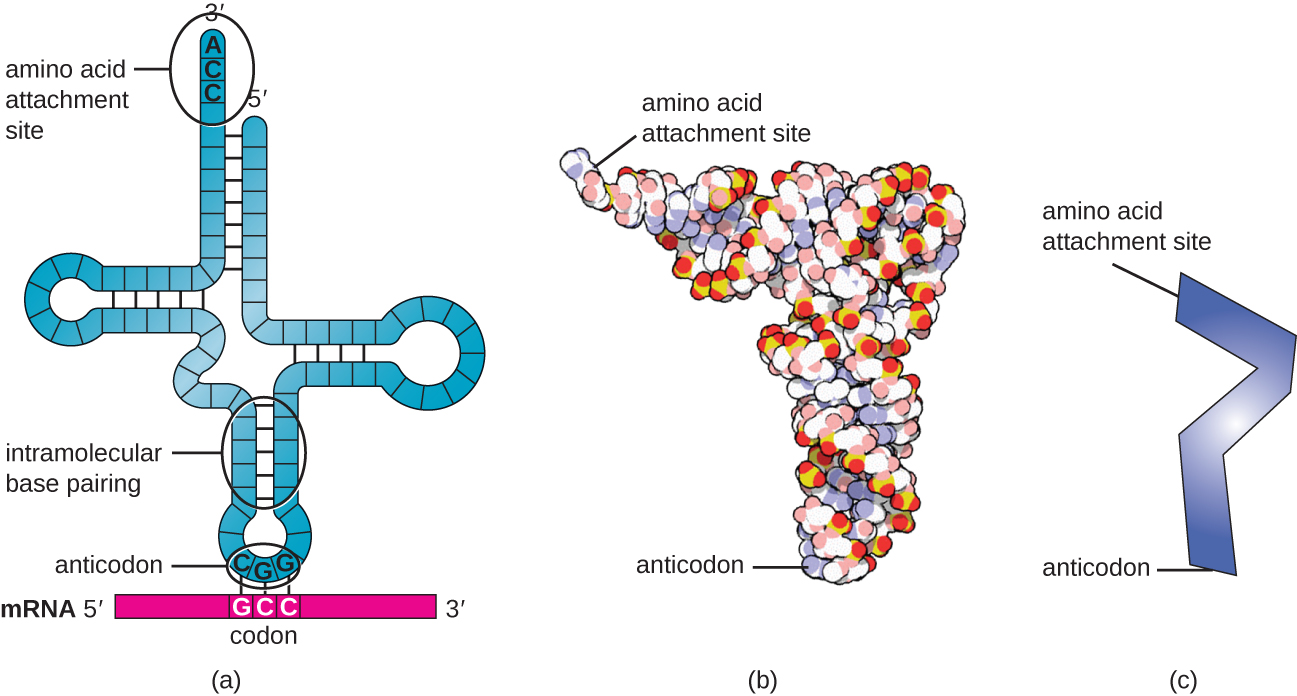
RNAs de transferência (tRNAs) são moléculas de RNA estrutural e, dependendo da espécie, existem muitos tipos diferentes de tRNAs no citoplasma. As espécies bacterianas normalmente têm entre 60 e 90 tipos. Servindo como adaptadores, cada tipo de tRNA se liga a um códon específico no modelo de mRNA e adiciona o aminoácido correspondente à cadeia polipeptídica. Portanto, os tRNAs são as moléculas que realmente “traduzem” a linguagem do RNA para a linguagem das proteínas. Como moléculas adaptadoras da tradução, é surpreendente que os tRNAs possam encaixar tanta especificidade em um pacote tão pequeno. A molécula de tRNA interage com três fatores: aminoacil tRNA sintetase, ribossomos e mRNA.
Os tRNAs maduros assumem uma estrutura tridimensional quando bases complementares expostas na molécula de RNA de fita simples se ligam entre si (Figura\(\PageIndex{3}\)). Essa forma posiciona o local de ligação de aminoácidos, chamado de extremidade de ligação de aminoácidos CCA, que é uma sequência de citosina-citosina-adenina na extremidade 3' do tRNA, e o anticodonato na outra extremidade. O anticódon é uma sequência de três nucleotídeos que se liga a um códon de mRNA por meio de emparelhamento de bases complementares.
Um aminoácido é adicionado ao final de uma molécula de tRNA por meio do processo de “carga” de tRNA, durante o qual cada molécula de tRNA é ligada ao seu aminoácido correto ou cognato por um grupo de enzimas chamadas aminoacil tRNA sintetases. Existe pelo menos um tipo de aminoacil tRNA sintetase para cada um dos 20 aminoácidos. Durante esse processo, o aminoácido é primeiro ativado pela adição de monofosfato de adenosina (AMP) e depois transferido para o tRNA, tornando-o um tRNA carregado, e o AMP é liberado.
Exercício\(\PageIndex{2}\)
- Descreva a estrutura e a composição do ribossomo procariótico.
- Em que direção o modelo de mRNA é lido?
- Descreva a estrutura e a função de um tRNA.
O mecanismo da síntese protéica
A tradução é semelhante em procariontes e eucariotas. Aqui, exploraremos como a tradução ocorre em E. coli, um procariota representativo, e especificaremos quaisquer diferenças entre a tradução bacteriana e eucariótica.
Iniciação
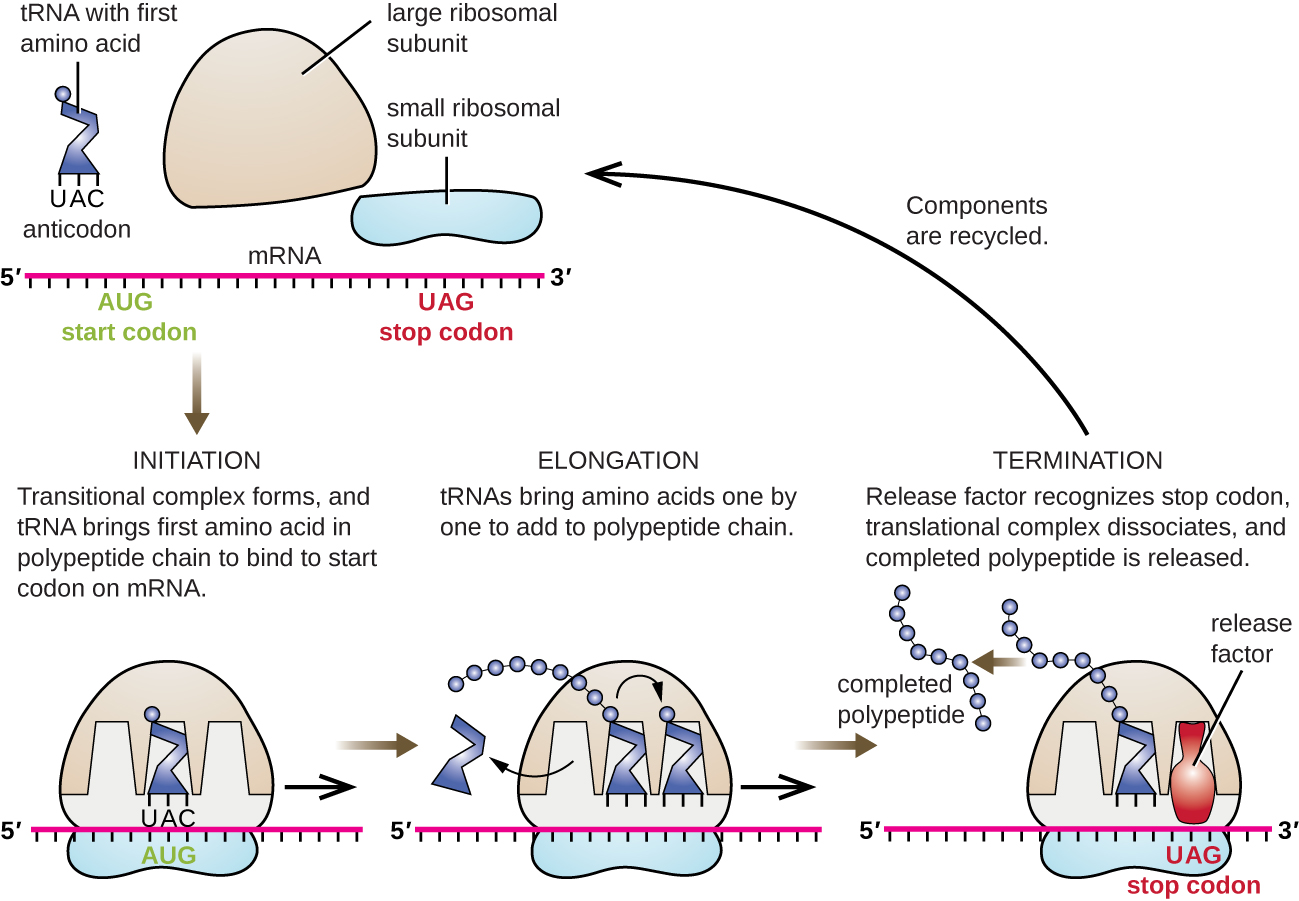
O início da síntese protéica começa com a formação de um complexo de iniciação. Em E. coli, esse complexo envolve o pequeno ribossomo 30S, o modelo de mRNA, três fatores de iniciação que ajudam o ribossomo a se reunir corretamente, o trifosfato de guanosina (GTP) que atua como uma fonte de energia e um iniciador especial (tRNA) transportando N -formil-metionina (FMET-tRNA fMET). ) (Figura\(\PageIndex{4}\)). O tRNA iniciador interage com o códon inicial AUG do mRNA e carrega uma metionina formilada (fMET). Devido ao seu envolvimento na iniciação, o FMet é inserido no início (terminal N) de cada cadeia polipeptídica sintetizada por E. coli. Em E. coli, o mRNA, uma sequência líder a montante do primeiro códon AUG, chamada sequência Shine-Dalgarno (também conhecida como sítio de ligação ribossômico AGGAGG), interage por meio de emparelhamento de bases complementares com as moléculas de rRNA que compõem o ribossomo. Essa interação ancora a subunidade ribossômica 30S na localização correta no modelo de mRNA. Nesse ponto, a subunidade ribossômica 50S então se liga ao complexo de iniciação, formando um ribossomo intacto.
Nos eucariotos, a formação do complexo de iniciação é semelhante, com as seguintes diferenças:
- O tRNA iniciador é um tRNA especializado diferente que transporta metionina, chamado met-tRNAi
- Em vez de se ligar ao mRNA na sequência Shine-Dalgarno, o complexo de iniciação eucariótica reconhece a capa de 5' do mRNA eucariótico e, em seguida, rastreia ao longo do mRNA na direção 5' a 3' até que o códon inicial do AUG seja reconhecido. Nesse ponto, a subunidade 60S se liga ao complexo de met-tRNAi, mRNA e subunidade 40S.
Alongamento
Em procariontes e eucariotos, os princípios básicos do alongamento da tradução são os mesmos. Em E. coli, a ligação da subunidade ribossômica 50S para produzir o ribossomo intacto forma três sítios ribossômicos funcionalmente importantes: O sítio A (aminoacil) se liga aos tRNAs de aminoacil carregados de entrada. O sítio P (peptidil) se liga aos tRNAs carregados que transportam aminoácidos que formaram ligações peptídicas com a cadeia polipeptídica em crescimento, mas ainda não se dissociaram do tRNA correspondente. O site E (saída) libera tRNAs dissociados para que eles possam ser recarregados com aminoácidos livres. Há uma exceção notável a esta linha de montagem de tRNAs: Durante a formação do complexo de iniciação, o FMEt-tRNA bacteriano fMET ou o met-tRNAi eucariótico entram diretamente no sítio P sem primeiro entrar no sítio A, fornecendo um local A gratuito pronto para aceitar o tRNA correspondente ao primeiro códon após o AUG.
O alongamento ocorre com movimentos de códon único do ribossomo, cada um chamado de evento de translocação. Durante cada evento de translocação, os tRNAs carregados entram no local A, depois mudam para o local P e, finalmente, para o local E para remoção. Os movimentos ribossômicos, ou etapas, são induzidos por mudanças conformacionais que avançam o ribossomo em três bases na direção 3'. As ligações peptídicas se formam entre o grupo amino do aminoácido ligado ao tRNA do sítio A e o grupo carboxila do aminoácido ligado ao tRNA do sítio P. A formação de cada ligação peptídica é catalisada pela peptidil transferase, uma ribozima à base de RNA que é integrada à subunidade ribossômica 50S. O aminoácido ligado ao tRNA do sítio P também está ligado à crescente cadeia polipeptídica. À medida que o ribossomo atravessa o mRNA, o antigo tRNA do sítio P entra no sítio E, se desprende do aminoácido e é expelido. Várias das etapas durante o alongamento, incluindo a ligação de um aminoacil tRNA carregado ao sítio A e a translocação, requerem energia derivada da hidrólise do GTP, que é catalisada por fatores específicos de alongamento. Surpreendentemente, o aparelho de tradução de E. coli leva apenas 0,05 segundos para adicionar cada aminoácido, o que significa que uma proteína de 200 aminoácidos pode ser traduzida em apenas 10 segundos.
Rescisão
O término da tradução ocorre quando um códon sem sentido (UAA, UAG ou UGA) é encontrado para o qual não há tRNA complementar. Ao se alinharem com o local A, esses códons sem sentido são reconhecidos por fatores de liberação em procariontes e eucariotos que resultam na separação do aminoácido do sítio P de seu tRNA, liberando o polipeptídeo recém-fabricado. As subunidades ribossômicas pequenas e grandes se dissociam do mRNA e umas das outras; elas são recrutadas quase imediatamente para outro complexo de iniciação de tradução.
Em resumo, existem várias características principais que distinguem a expressão gênica procariótica daquela observada em eucariotos. Eles são ilustrados na Figura\(\PageIndex{5}\) e listados na Figura\(\PageIndex{6}\).
Direcionamento, dobramento e modificação de proteínas
Durante e após a tradução, os polipeptídeos podem precisar ser modificados antes de serem biologicamente ativos. As modificações pós-traducionais incluem:
- remoção de sequências de sinais traduzidos — caudas curtas de aminoácidos que ajudam a direcionar uma proteína para um compartimento celular específico
- “dobramento” adequado do polipeptídeo e associação de múltiplas subunidades polipeptídicas, muitas vezes facilitadas pelas proteínas chaperonas, em uma estrutura tridimensional distinta
- processamento proteolítico de um polipeptídeo inativo para liberar um componente proteico ativo, e
- várias modificações químicas (por exemplo, fosforilação, metilação ou glicosilação) de aminoácidos individuais.
Exercício\(\PageIndex{3}\)
- Quais são os componentes do complexo de iniciação para tradução em procariontes?
- Quais são as duas diferenças entre o início da tradução procariótica e eucariótica?
- O que ocorre em cada um dos três sítios ativos do ribossomo?
- O que causa o término da tradução?
Conceitos principais e resumo
- Na tradução, os polipeptídeos são sintetizados usando sequências de mRNA e maquinaria celular, incluindo tRNAs que combinam códons de mRNA a aminoácidos e ribossomos específicos compostos de RNA e proteínas que catalisam a reação.
- O código genético é degenerado, pois vários códons de mRNA codificam para os mesmos aminoácidos. O código genético é quase universal entre os organismos vivos.
- Os ribossomos procarióticos (70S) e eucarióticos citoplasmáticos (80S) são compostos por uma grande subunidade e uma pequena subunidade de tamanhos diferentes entre os dois grupos. Cada subunidade é composta por rRNA e proteína. Os ribossomos organelares em células eucarióticas se assemelham aos ribossomos procarióticos.
- Cerca de 60 a 90 espécies de tRNA existem nas bactérias. Cada tRNA tem um anticódon de três nucleotídeos, bem como um sítio de ligação para um aminoácido cognato. Todos os tRNAs com um anticódon específico carregarão o mesmo aminoácido.
- O início da tradução ocorre quando a pequena subunidade ribossômica se liga aos fatores de iniciação e a um tRNA iniciador no códon inicial de um mRNA, seguido pela ligação ao complexo de iniciação da grande subunidade ribossômica.
- Nas células procarióticas, o códon inicial codifica a N-formil-metionina transportada por um iniciador especial tRNA. Nas células eucarióticas, o códon inicial codifica a metionina transportada por um iniciador especial tRNA. Além disso, enquanto a ligação ribossômica do mRNA em procariontes é facilitada pela sequência Shine-Dalgarno dentro do mRNA, os ribossomos eucarióticos se ligam à capa de 5' do mRNA.
- Durante o estágio de alongamento da tradução, um tRNA carregado se liga ao mRNA no sítio A do ribossomo; uma ligação peptídica é catalisada entre os dois aminoácidos adjacentes, quebrando a ligação entre o primeiro aminoácido e seu tRNA; o ribossomo move um códon ao longo do mRNA; e o primeiro tRNA é movido do sítio P do ribossomo para o sítio E e sai do complexo ribossômico.
- O término da tradução ocorre quando o ribossomo encontra um códon de parada, que não codifica para um tRNA. Os fatores de liberação fazem com que o polipeptídeo seja liberado e o complexo ribossômico se dissocia.
- Em procariontes, a transcrição e a tradução podem ser acopladas, com a tradução de uma molécula de mRNA começando assim que a transcrição permite exposição suficiente ao mRNA para a ligação de um ribossomo, antes do término da transcrição. A transcrição e a tradução não são acopladas em eucariotos porque a transcrição ocorre no núcleo, enquanto a tradução ocorre no citoplasma ou em associação com o retículo endoplasmático rugoso.
- Os polipeptídeos geralmente requerem uma ou mais modificações pós-traducionais para se tornarem biologicamente ativos.


