11.3: Transcrição de RNA
- Page ID
- 181358
Objetivos de
- Explicar como o RNA é sintetizado usando o DNA como modelo
- Faça a distinção entre transcrição em procariontes e eucariotos
Durante o processo de transcrição, a informação codificada na sequência de DNA de um ou mais genes é transcrita em uma fita de RNA, também chamada de transcrição de RNA. A molécula de RNA de fita simples resultante, composta por ribonucleotídeos contendo as bases adenina (A), citosina (C), guanina (G) e uracil (U), atua como uma cópia molecular móvel da sequência de DNA original. A transcrição em procariontes e eucariotos requer que a dupla hélice do DNA se desenrole parcialmente na região da síntese de RNA. A região desenrolada é chamada de bolha de transcrição. A transcrição de um gene específico sempre procede de uma das duas cadeias de DNA que atua como um modelo, a chamada fita antisense. O produto de RNA é complementar à fita modelo do DNA e é quase idêntico à fita de DNA não modelo, ou fita sensorial. A única diferença é que no RNA, todos os nucleotídeos T são substituídos por nucleotídeos U; durante a síntese do RNA, U é incorporado quando há um A na fita antisense complementar.
Transcrição em bactérias
As bactérias usam a mesma RNA polimerase para transcrever todos os seus genes. Como a DNA polimerase, a RNA polimerase adiciona nucleotídeos um a um ao grupo 3'-OH da crescente cadeia de nucleotídeos. Uma diferença crítica na atividade entre a DNA polimerase e a RNA polimerase é a necessidade de um 3'-OH para adicionar nucleotídeos: a DNA polimerase requer esse grupo 3'-OH, portanto, necessita de um primer, enquanto a RNA polimerase não. Durante a transcrição, um ribonucleotídeo complementar à fita modelo de DNA é adicionado à fita de RNA em crescimento e uma ligação fosfodiéster covalente é formada pela síntese de desidratação entre o novo nucleotídeo e o último adicionado. Em E. coli, a RNA polimerase compreende seis subunidades polipeptídicas, cinco das quais compõem a enzima central da polimerase responsável por adicionar nucleotídeos de RNA a uma fita em crescimento. A sexta subunidade é conhecida como sigma (σ). O fator σ permite que a RNA polimerase se ligue a um promotor específico, permitindo assim a transcrição de vários genes. Existem vários fatores σ que permitem a transcrição de vários genes.
Iniciação
O início da transcrição começa em um promotor, uma sequência de DNA na qual o mecanismo de transcrição se liga e inicia a transcrição. O par de nucleotídeos na dupla hélice do DNA que corresponde ao local a partir do qual o primeiro nucleotídeo de 5' RNA é transcrito é o local de iniciação. Os nucleotídeos que precedem o local de iniciação são designados “a montante”, enquanto os nucleotídeos que seguem o local de iniciação são chamados de nucleotídeos “a jusante”. Na maioria dos casos, os promotores estão localizados logo acima dos genes que eles regulam. Embora as sequências de promotores variem entre os genomas bacterianos, alguns elementos são conservados. Nas posições —10 e —35 dentro do DNA antes do local de iniciação (designado +1), há duas sequências de consenso do promotor, ou regiões, que são semelhantes em todos os promotores e em várias espécies bacterianas. A sequência de consenso de —10, chamada de caixa TATA, é TATAAT. A sequência —35 é reconhecida e limitada por σ.
Alongamento
O alongamento na fase de transcrição começa quando a subunidade σ se dissocia da polimerase, permitindo que a enzima central sintetize RNA complementar ao modelo de DNA na direção de 5' a 3' a uma taxa de aproximadamente 40 nucleotídeos por segundo. À medida que o alongamento prossegue, o DNA é continuamente desenrolado à frente da enzima central e rebobinado atrás dela (Figura\(\PageIndex{1}\)).
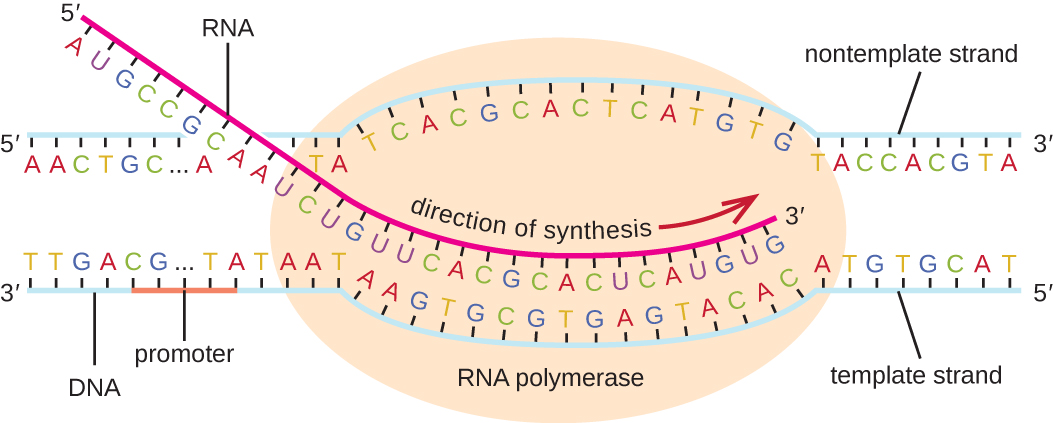
Rescisão
Depois que um gene é transcrito, a polimerase bacteriana deve se dissociar do modelo de DNA e liberar o RNA recém-criado. Isso é conhecido como encerramento da transcrição. O modelo de DNA inclui sequências repetidas de nucleotídeos que atuam como sinais de terminação, fazendo com que a RNA polimerase pare e se libere do modelo de DNA, liberando a transcrição do RNA.
Exercício\(\PageIndex{1}\)
- Onde o fator σ da RNA polimerase se liga ao DNA para iniciar a transcrição?
- O que ocorre para iniciar a atividade de polimerização da RNA polimerase?
- De onde vem o sinal para finalizar a transcrição?
Transcrição em eucariotos
Procariontes e eucariotos realizam fundamentalmente o mesmo processo de transcrição, com algumas diferenças significativas (consulte a Tabela\(\PageIndex{1}\)). Os eucariotos usam três polimerases diferentes, RNA polimerases I, II e III, todas estruturalmente distintas da RNA polimerase bacteriana. Cada um transcreve um subconjunto diferente de genes. Curiosamente, as arquéias contêm uma única RNA polimerase que está mais intimamente relacionada à RNA polimerase II eucariótica do que à sua contraparte bacteriana. Os mRNAs eucarióticos também são geralmente monocistrônicos, o que significa que cada um deles codifica apenas um único polipeptídeo, enquanto os mRNAs procarióticos de bactérias e arquéias são comumente policistrônicos, o que significa que eles codificam vários polipeptídeos.
A diferença mais importante entre procariontes e eucariotos é o núcleo ligado à membrana deste último, que influencia a facilidade de uso das moléculas de RNA para a síntese de proteínas. Com os genes ligados a um núcleo, a célula eucariótica deve transportar moléculas de RNA codificadoras de proteínas para o citoplasma para serem traduzidas. Os transcritos primários codificadores de proteínas, as moléculas de RNA sintetizadas diretamente pela RNA polimerase, devem passar por várias etapas de processamento para proteger essas moléculas de RNA da degradação durante o tempo em que são transferidas do núcleo para o citoplasma e traduzidas em uma proteína. Por exemplo, mRNAs eucarióticos podem durar várias horas, enquanto o mRNA procariótico típico não dura mais do que 5 segundos.
O transcrito primário (também chamado de pré-mRNA) é primeiro revestido com proteínas estabilizadoras de RNA para protegê-lo da degradação enquanto é processado e exportado para fora do núcleo. O primeiro tipo de processamento começa enquanto a transcrição primária ainda está sendo sintetizada; um nucleotídeo especial de 7-metilguanosina, chamado 5' cap, é adicionado à extremidade 5' da transcrição crescente. Além de impedir a degradação, os fatores envolvidos na síntese proteica subsequente reconhecem a tampa, que ajuda a iniciar a tradução pelos ribossomos. Quando o alongamento é concluído, outra enzima de processamento adiciona uma sequência de aproximadamente 200 nucleotídeos de adenina na extremidade 3', chamada cauda de poli-A. Essa modificação protege ainda mais o pré-mRNA da degradação e sinaliza aos fatores celulares que a transcrição precisa ser exportada para o citoplasma.
Os genes eucarióticos que codificam polipeptídeos são compostos por sequências codificadoras chamadas éxons (ex -on significa que eles são expressos) e sequências intermediárias chamadas introns (int -ron denota seu papel int ervening). As sequências de RNA transcritas correspondentes aos íntrons não codificam regiões do polipeptídeo funcional e são removidas do pré-mRNA durante o processamento. É essencial que todas as sequências de RNA codificadas por íntron sejam removidas completa e com precisão de um pré-mRNA antes da síntese protéica, para que as sequências de RNA codificadas em éxon sejam devidamente unidas para codificar um polipeptídeo funcional. Se o processo errar até mesmo por um único nucleotídeo, as sequências dos éxons reunidos seriam deslocadas e o polipeptídeo resultante não funcionaria. O processo de remoção de sequências de RNA codificadas por íntron e reconectar aquelas codificadas por éxons é chamado de splicing de RNA e é facilitado pela ação de um spliceossomo contendo pequenas proteínas ribonucleares nucleares (SNRNPs). As sequências de RNA codificadas com intrões são removidas do pré-mRNA enquanto ele ainda está no núcleo. Embora não sejam traduzidos, os introns parecem ter várias funções, incluindo regulação gênica e transporte de mRNA. Após a conclusão dessas modificações, o transcrito maduro, o mRNA que codifica um polipeptídeo, é transportado para fora do núcleo, destinado ao citoplasma para tradução. Os íntrons podem ser separados de forma diferente, resultando na inclusão ou exclusão de vários éxons do produto final de mRNA. Esse processo é conhecido como empalme alternativo. A vantagem do splicing alternativo é que diferentes tipos de transcritos de mRNA podem ser gerados, todos derivados da mesma sequência de DNA. Nos últimos anos, foi demonstrado que algumas arquéias também têm a capacidade de unir seu pré-mRNA.
| Propriedade | Bactérias | Eucariotos |
|---|---|---|
| Número de polipeptídeos codificados por mRNA | Monocistrônico ou policistrônico | Exclusivamente monocistrônico |
| Alongamento do fio | núcleo + σ = holoenzima | RNA polimerases I, II ou III |
| Adição de uma tampa de 5 pés | Não | sim |
| Adição de cauda de poli-A de 3' | Não | sim |
| Splicing do pré-mRNA | Não | sim |
Visualize como acontece o empalme de mRNA observando o processo em ação neste vídeo. Veja como os introns são removidos durante o empalme de RNA aqui.
Exercício\(\PageIndex{2}\)
- Nas células eucarióticas, como a transcrição do RNA de um gene para uma proteína é modificada após a transcrição?
- Os éxons ou introns contêm informações para sequências de proteínas?
Foco clínico: Parte 2
No pronto-socorro, uma enfermeira disse a Mark que ele havia tomado uma boa decisão de ir ao hospital porque seus sintomas indicavam uma infecção que havia saído do controle. Os sintomas de Mark haviam progredido, com a área da pele afetada e a quantidade de inchaço aumentando. Dentro da área afetada, uma erupção cutânea havia começado, bolhas e pequenas bolsas de gás sob a camada mais externa da pele se formaram, e parte da pele estava ficando cinza. Com base no cheiro pútrido do pus drenado de uma das bolhas, na rápida progressão da infecção e na aparência visual da pele afetada, o médico imediatamente iniciou o tratamento da fascite necrosante. O médico de Mark solicitou uma cultura do fluido drenado do blister e também solicitou exames de sangue, incluindo uma contagem de glóbulos brancos.
Mark foi internado na unidade de terapia intensiva e iniciou a administração intravenosa de um antibiótico de amplo espectro para tentar minimizar a disseminação da infecção. Apesar da antibioticoterapia, a condição de Mark piorou rapidamente. Mark ficou confuso e tonto. Poucas horas após sua admissão no hospital, sua pressão arterial caiu significativamente e sua respiração ficou mais rasa e mais rápida. Além disso, a formação de bolhas aumentou, com as bolhas se intensificando para preto arroxeado, e a ferida em si parecia estar progredindo rapidamente pela perna de Mark.
Exercício\(\PageIndex{3}\)
- Quais são os possíveis agentes causadores da fascite necrosante de Mark?
- Quais são algumas das possíveis explicações para o motivo pelo qual o tratamento com antibióticos parece não estar funcionando?
Conceitos principais e resumo
- Durante a transcrição, as informações codificadas no DNA são usadas para produzir RNA.
- A RNA polimerase sintetiza o RNA, usando a fita antisense do DNA como modelo, adicionando nucleotídeos de RNA complementares à extremidade 3' da fita em crescimento.
- A RNA polimerase se liga ao DNA em uma sequência chamada promotor durante o início da transcrição.
- Genes que codificam proteínas de funções relacionadas são frequentemente transcritos sob o controle de um único promotor em procariontes, resultando na formação de uma molécula de mRNA policistrônico que codifica vários polipeptídeos.
- Ao contrário da DNA polimerase, a RNA polimerase não requer um grupo 3'-OH para adicionar nucleotídeos, portanto, um primer não é necessário durante a iniciação.
- O término da transcrição em bactérias ocorre quando a RNA polimerase encontra sequências específicas de DNA que levam à paralisação da polimerase. Isso resulta na liberação de RNA polimerase da fita modelo de DNA, liberando a transcrição do RNA.
- Os eucariotos têm três RNA polimerases diferentes. Os eucariotos também têm mRNA monocistrônico, cada um codificando apenas um único polipeptídeo.
- Os transcritos primários eucarióticos são processados de várias maneiras, incluindo a adição de uma tampa de 5' e uma cauda de 3′-poli-A, bem como o splicing, para gerar uma molécula madura de mRNA que pode ser transportada para fora do núcleo e protegida da degradação.


