11.2: Replicação do DNA
- Page ID
- 181359
Objetivos de
- Explicar o significado da replicação semiconservadora do DNA
- Explicar por que a replicação do DNA é bidirecional e inclui uma fita dianteira e uma cadeia atrasada
- Explique por que os fragmentos de Okazaki são formados
- Descreva o processo de replicação do DNA e as funções das enzimas envolvidas
- Identifique as diferenças entre a replicação do DNA em bactérias e eucariotos
- Explicar o processo de replicação circular
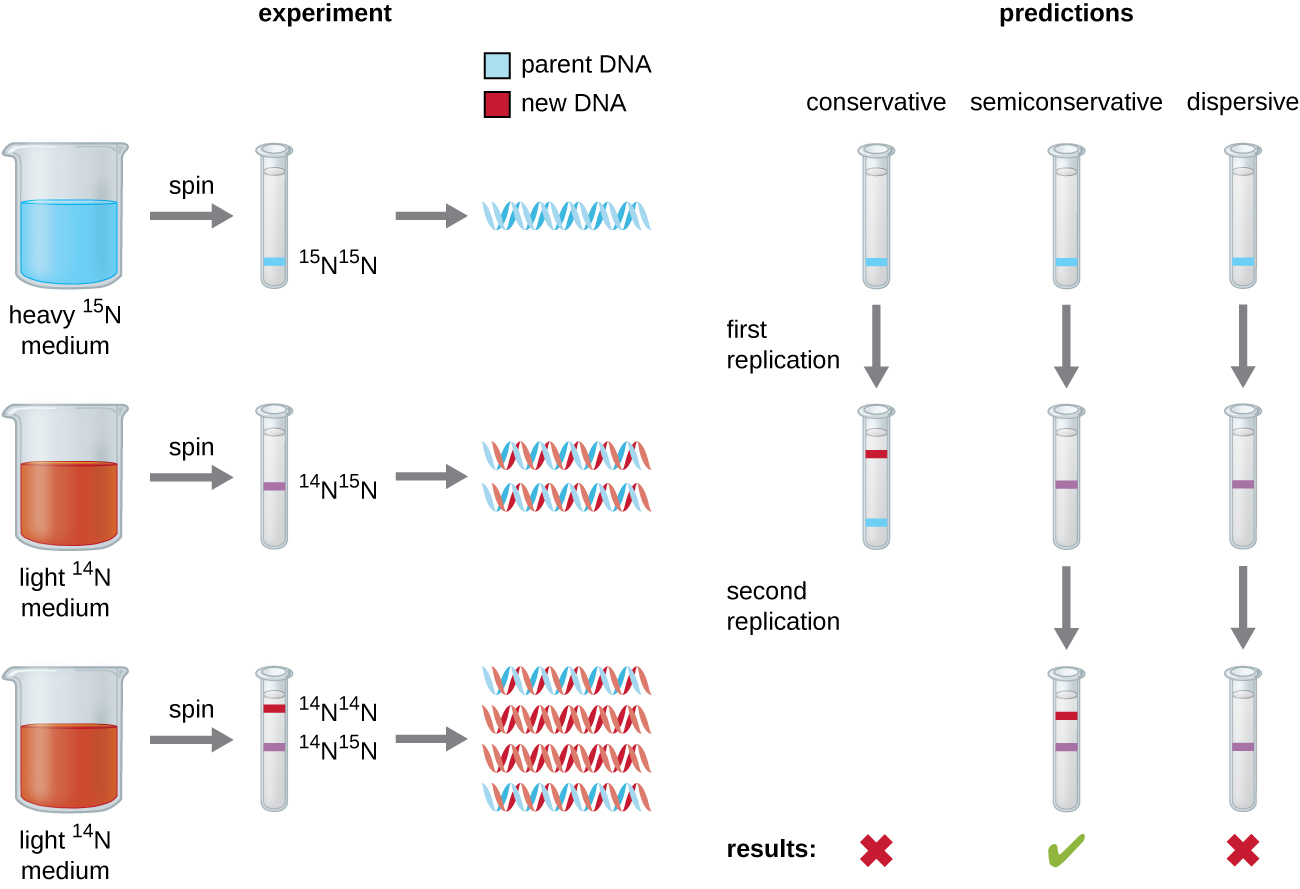
A elucidação da estrutura da dupla hélice por James Watson e Francis Crick em 1953 forneceu uma dica de como o DNA é copiado durante o processo de replicação. A separação das fitas da dupla hélice forneceria dois modelos para a síntese de novas fitas complementares, mas ainda não estava claro exatamente como as novas moléculas de DNA foram construídas. Em um modelo, replicação semiconservadora, as duas fitas da dupla hélice se separam durante a replicação do DNA, e cada fita serve como um modelo a partir do qual a nova fita complementar é copiada; após a replicação, cada DNA de fita dupla inclui uma fita parental ou “antiga” e uma fita “nova”. Também foram sugeridos dois modelos concorrentes: conservador e dispersivo, que são mostrados na Figura\(\PageIndex{1}\).

Matthew Meselson (1930—) e Franklin Stahl (1929—) criaram um experimento em 1958 para testar qual desses modelos representa corretamente a replicação do DNA (Figura\(\PageIndex{2}\)). Eles cultivaram E. coli por várias gerações em um meio contendo um isótopo “pesado” de nitrogênio (15 N) que foi incorporado às bases nitrogenadas e, eventualmente, ao DNA. Isso rotulou o DNA dos pais. A cultura de E. coli foi então transferida para um meio contendo 14 N e deixada crescer por uma geração. As células foram colhidas e o DNA foi isolado. O DNA foi separado por ultracentrifugação, durante a qual o DNA formou bandas de acordo com sua densidade. Espera-se que o DNA cultivado em 15 N forme uma banda em uma posição de maior densidade do que a cultivada em 14 N. Meselson e Stahl observaram que após uma geração de crescimento em 14 N, a banda única observada era intermediária na posição entre o DNA das células cultivadas exclusivamente em 15 N ou 14 N. Isso sugeriu um modo de replicação semiconservador ou dispersivo. Foi permitido que algumas células crescessem por mais uma geração em 14 N e girassem novamente. O DNA colhido de células cultivadas por duas gerações em 14 N formou duas bandas: uma banda de DNA estava na posição intermediária entre 15 N e 14 N e a outra correspondia à banda de 14 N de DNA. Esses resultados só poderiam ser explicados se o DNA se replicar de maneira semiconservadora. Portanto, os outros dois modelos foram descartados. Como resultado desse experimento, agora sabemos que, durante a replicação do DNA, cada uma das duas fitas que compõem a dupla hélice serve como um modelo a partir do qual novas fitas são copiadas. A nova vertente será complementar à vertente parental ou “antiga”. As moléculas de DNA resultantes têm a mesma sequência e são divididas igualmente nas duas células-filhas.
Exercício\(\PageIndex{1}\)
Qual teria sido a conclusão do experimento de Meselson e Stahl se, após a primeira geração, eles tivessem encontrado duas bandas de DNA?
Replicação de DNA em bactérias
A replicação do DNA tem sido bem estudada em bactérias, principalmente por causa do pequeno tamanho do genoma e dos mutantes disponíveis. E. coli tem 4,6 milhões de pares de bases (Mbp) em um único cromossomo circular e tudo isso é replicado em aproximadamente 42 minutos, começando de uma única origem de replicação e prosseguindo ao redor do círculo bidirecionalmente (ou seja, em ambas as direções). Isso significa que aproximadamente 1000 nucleotídeos são adicionados por segundo. O processo é bastante rápido e ocorre com poucos erros.
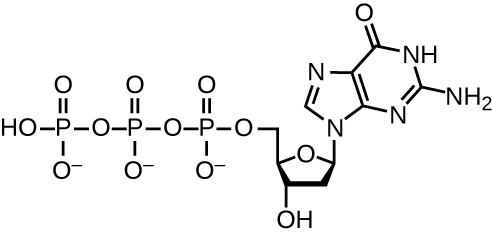
A replicação do DNA usa um grande número de proteínas e enzimas (Tabela\(\PageIndex{1}\)). Um dos principais atores é a enzima DNA polimerase, também conhecida como DNA pol. Nas bactérias, três tipos principais de DNA polimerases são conhecidos: DNA pol I, DNA pol II e DNA pol III. Sabe-se agora que o DNA pol III é a enzima necessária para a síntese de DNA; DNA pol I e DNA pol II são necessários principalmente para reparo. O DNA pol III adiciona desoxirribonucleotídeos, cada um complementar a um nucleotídeo na fita modelo, um a um ao grupo 3'-OH da cadeia de DNA em crescimento. A adição desses nucleotídeos requer energia. Essa energia está presente nas ligações de três grupos fosfato ligados a cada nucleotídeo (um nucleotídeo trifosfato), semelhante à forma como a energia é armazenada nas ligações fosfatadas do trifosfato de adenosina (ATP) (Figura\(\PageIndex{3}\)). Quando a ligação entre os fosfatos é quebrada e o difosfato é liberado, a energia liberada permite a formação de uma ligação fosfodiéster covalente por síntese de desidratação entre o nucleotídeo entrante e o grupo 3'-OH livre na fita de DNA em crescimento.
Iniciação
O início da replicação ocorre em uma sequência específica de nucleotídeos chamada origem da replicação, onde várias proteínas se ligam para iniciar o processo de replicação. A E. coli tem uma única origem de replicação (assim como a maioria dos procariontes), chamada ORIC, em seu único cromossomo. A origem da replicação tem aproximadamente 245 pares de bases e é rica em sequências de adenina-timina (AT).
Algumas das proteínas que se ligam à origem da replicação são importantes para tornar as regiões de fita simples do DNA acessíveis para replicação. O DNA cromossômico é normalmente enrolado em torno de histonas (em eucariotos e arquéias) ou proteínas semelhantes a histonas (em bactérias) e é superenrolado ou extensivamente envolto e torcido sobre si mesmo. Essa embalagem torna as informações na molécula de DNA inacessíveis. No entanto, enzimas chamadas topoisomerases alteram a forma e o superenrolamento do cromossomo. Para que a replicação do DNA bacteriano comece, o cromossomo superenrolado é relaxado pela topoisomerase II, também chamada de DNA girase. Uma enzima chamada helicase então separa as cadeias de DNA quebrando as ligações de hidrogênio entre os pares de bases nitrogenadas. Lembre-se de que as sequências AT têm menos ligações de hidrogênio e, portanto, têm interações mais fracas do que as sequências de guanina-citosina (GC). Essas enzimas requerem hidrólise de ATP. À medida que o DNA se abre, estruturas em forma de Y chamadas garfos de replicação são formadas. Dois garfos de replicação são formados na origem da replicação, permitindo a replicação bidirecional e a formação de uma estrutura que parece uma bolha quando vista com um microscópio eletrônico de transmissão; como resultado, essa estrutura é chamada de bolha de replicação. O DNA próximo a cada garfo de replicação é revestido com proteínas de ligação de fita simples para evitar que o DNA de fita simples se rebobine em uma dupla hélice.
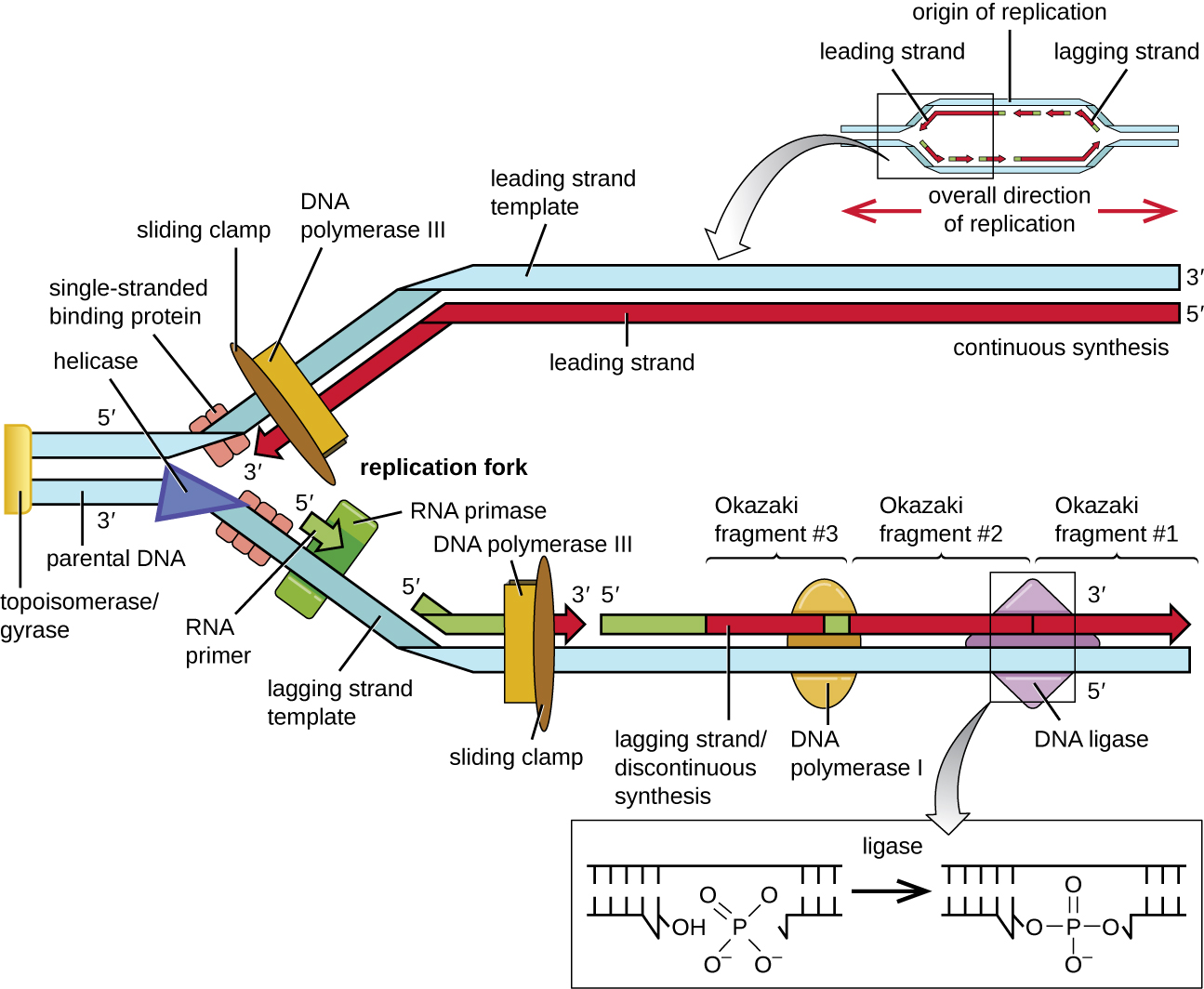
Uma vez que o DNA de fita simples esteja acessível na origem da replicação, a replicação do DNA pode começar. No entanto, o DNA pol III é capaz de adicionar nucleotídeos somente na direção 5' a 3' (uma nova fita de DNA só pode ser estendida nessa direção). Isso ocorre porque a DNA polimerase requer um grupo 3'-OH livre ao qual ela pode adicionar nucleotídeos formando uma ligação fosfodiéster covalente entre a extremidade 3'-OH e o fosfato 5' do próximo nucleotídeo. Isso também significa que ele não pode adicionar nucleotídeos se um grupo 3'-OH livre não estiver disponível, o que é o caso de uma única fita de DNA. O problema é resolvido com a ajuda de uma sequência de RNA que fornece a extremidade livre de 3'-OH. Como essa sequência permite o início da síntese de DNA, ela é apropriadamente chamada de primer. O primer tem de cinco a 10 nucleotídeos e é complementar ao DNA parental ou modelo. É sintetizado pela RNA primase, que é uma RNA polimerase. Ao contrário das DNA polimerases, as RNA polimerases não precisam de um grupo 3'-OH livre para sintetizar uma molécula de RNA. Agora que o primer fornece o grupo 3'-OH livre, a DNA polimerase III agora pode estender esse primer de RNA, adicionando nucleotídeos de DNA, um por um, que são complementares à fita modelo (Figura\(\PageIndex{1}\)).
Alongamento
Durante o alongamento na replicação do DNA, a adição de nucleotídeos ocorre em sua taxa máxima de cerca de 1000 nucleotídeos por segundo. A DNA polimerase III só pode se estender na direção de 5' a 3', o que representa um problema na bifurcação de replicação. A dupla hélice do DNA é antiparalela; ou seja, uma fita é orientada na direção 5' a 3' e a outra é orientada na direção 3' a 5' (veja Estrutura e Função do DNA). Durante a replicação, uma fita, que é complementar à fita de DNA parental de 3' a 5', é sintetizada continuamente em direção ao garfo de replicação porque a polimerase pode adicionar nucleotídeos nessa direção. Essa fita sintetizada continuamente é conhecida como a fita principal. A outra fita, complementar ao DNA parental de 5' a 3', cresce longe do garfo de replicação, então a polimerase deve voltar para o garfo de replicação para começar a adicionar bases a um novo primer, novamente na direção longe do garfo de replicação. Ele faz isso até esbarrar no fio previamente sintetizado e, em seguida, voltar novamente (Figura\(\PageIndex{4}\)). Essas etapas produzem pequenos fragmentos de sequência de DNA conhecidos como fragmentos de Okazaki, cada um separado por primer de RNA. Os fragmentos de Okazaki receberam o nome da equipe de pesquisa japonesa e do casal Reiji e Tsuneko Okazaki, que os descobriram pela primeira vez em 1966. O fio com os fragmentos de Okazaki é conhecido como fio defasado, e sua síntese é considerada descontínua.
O fio principal pode ser estendido a partir de um único primer, enquanto o fio atrasado precisa de um novo primer para cada um dos fragmentos curtos de Okazaki. A direção geral do fio defasado será de 3' a 5', e a do fio principal de 5' a 3'. Uma proteína chamada pinça deslizante mantém a DNA polimerase no lugar enquanto continua adicionando nucleotídeos. A pinça deslizante é uma proteína em forma de anel que se liga ao DNA e mantém a polimerase no lugar. Além de seu papel na iniciação, a topoisomerase também evita o enrolamento excessivo da dupla hélice do DNA à frente da bifurcação de replicação quando o DNA se abre; ela faz isso causando cortes temporários na hélice do DNA e depois selando-a novamente. Conforme a síntese prossegue, os primers de RNA são substituídos pelo DNA. Os primers são removidos pela atividade da exonuclease da DNA polimerase I e as lacunas são preenchidas. Os cortes que permanecem entre o DNA recém-sintetizado (que substituiu o primer de RNA) e o DNA sintetizado anteriormente são selados pela enzima DNA ligase que catalisa a formação da ligação fosfodiéster covalente entre a extremidade 3'-OH de um fragmento de DNA e a extremidade de fosfato 5' do outro fragmento, estabilizando a espinha dorsal de açúcar-fosfato da molécula de DNA.
Rescisão
Uma vez que o cromossomo completo tenha sido replicado, o término da replicação do DNA deve ocorrer. Embora se saiba muito sobre o início da replicação, menos se sabe sobre o processo de encerramento. Após a replicação, os genomas circulares completos resultantes dos procariontes são concatenados, o que significa que os cromossomos circulares de DNA estão interligados e devem ser separados uns dos outros. Isso é feito por meio da atividade da topoisomerase IV bacteriana, que introduz quebras de fita dupla nas moléculas de DNA, permitindo que elas se separem umas das outras; a enzima então fecha novamente os cromossomos circulares. A resolução dos concatêmeros é um problema exclusivo da replicação do DNA procariótico por causa de seus cromossomos circulares. Como a DNA girase bacteriana e a topoisomerase IV são distintas de suas contrapartes eucarióticas, essas enzimas servem como alvos para uma classe de medicamentos antimicrobianos chamados quinolonas.
| Enzima ou fator | Função |
|---|---|
| Pesquisa de DNA I | A atividade da exonuclease remove o primer de RNA e o substitui por DNA recém-sintetizado |
| Pesquisa de DNA III | Enzima principal que adiciona nucleotídeos na direção 5' a 3' |
| Helicase | Abre a hélice do DNA quebrando as ligações de hidrogênio entre as bases nitrogenadas |
| Ligase | Sela as lacunas entre os fragmentos de Okazaki na fita defasada para criar uma fita contínua de DNA |
| Primase | Sintetiza os primers de RNA necessários para iniciar a replicação |
| Proteínas de ligação de fita simples | Lige-se ao DNA de fita simples para evitar a ligação de hidrogênio entre fitas de DNA, reformando o DNA de fita dupla |
| Braçadeira deslizante | Ajuda a manter o DNA pol III no lugar quando nucleotídeos estão sendo adicionados |
| Topoisomerase II (DNA girase) | Relaxa o cromossomo superenrolado para tornar o DNA mais acessível para o início da replicação; ajuda a aliviar o estresse no DNA ao se desenrolar, causando quebras e depois selando novamente o DNA |
| Topoisomerase IV | Introduz a quebra de fita simples em cromossomos concatenados para liberá-los uns dos outros e, em seguida, revela o DNA |
Exercício\(\PageIndex{2}\)
- Qual enzima quebra as ligações de hidrogênio que mantêm as duas fitas de DNA juntas para que a replicação possa ocorrer?
- É o fio atrasado ou o fio principal que é sintetizado na direção da abertura do garfo de replicação?
- Qual enzima é responsável pela remoção dos primers de RNA no DNA bacteriano recém-replicado?
Replicação de DNA em eucariotos
Os genomas eucarióticos são muito mais complexos e maiores do que os genomas procarióticos e são normalmente compostos por vários cromossomos lineares (Tabela\(\PageIndex{2}\)). O genoma humano, por exemplo, tem 3 bilhões de pares de bases por conjunto haplóide de cromossomos, e 6 bilhões de pares de bases são inseridos durante a replicação. Existem várias origens de replicação em cada cromossomo eucariótico (Figura\(\PageIndex{5}\)); o genoma humano tem 30.000 a 50.000 origens de replicação. A taxa de replicação é de aproximadamente 100 nucleotídeos por segundo — 10 vezes mais lenta do que a replicação procariótica.
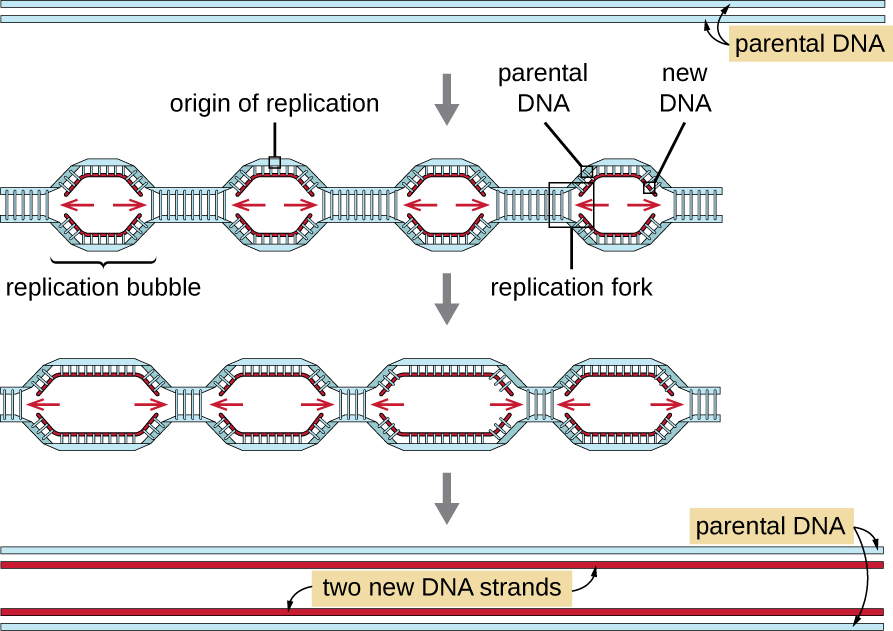
As etapas essenciais de replicação em eucariotos são as mesmas dos procariontes. Antes que a replicação possa começar, o DNA precisa ser disponibilizado como um modelo. O DNA eucariótico é altamente superenrolado e empacotado, o que é facilitado por muitas proteínas, incluindo histonas (consulte Estrutura e função dos genomas celulares). Na origem da replicação, um complexo de pré-replicação composto por várias proteínas, incluindo a helicase, forma e recruta outras enzimas envolvidas no início da replicação, incluindo topoisomerase para relaxar o superenrolamento, proteína de ligação de fita simples, RNA primase e DNA polimerase. Após o início da replicação, em um processo semelhante ao encontrado nos procariontes, o alongamento é facilitado pelas DNA polimerases eucarióticas. A fita principal é sintetizada continuamente pela enzima polimerase eucariótica pol δ, enquanto a fita defasada é sintetizada por pol ε. Uma proteína de pinça deslizante mantém a DNA polimerase no lugar para que ela não caia do DNA. A enzima ribonuclease H (RNase H), em vez de uma DNA polimerase como nas bactérias, remove o primer de RNA, que é então substituído por nucleotídeos de DNA. As lacunas que permanecem são seladas pela DNA ligase.
Como os cromossomos eucarióticos são lineares, pode-se esperar que sua replicação seja mais direta. Como nos procariontes, a DNA polimerase eucariótica pode adicionar nucleotídeos somente na direção 5' a 3'. Na cadeia principal, a síntese continua até atingir o final do cromossomo ou outra bifurcação de replicação progredindo na direção oposta. Na fita defasada, o DNA é sintetizado em trechos curtos, cada um dos quais é iniciado por um primer separado. Quando a bifurcação de replicação atinge o final do cromossomo linear, não há lugar para fazer um primer para que o fragmento de DNA seja copiado no final do cromossomo. Essas extremidades, portanto, permanecem desemparelhadas e, com o tempo, podem ficar progressivamente mais curtas à medida que as células continuam a se dividir.
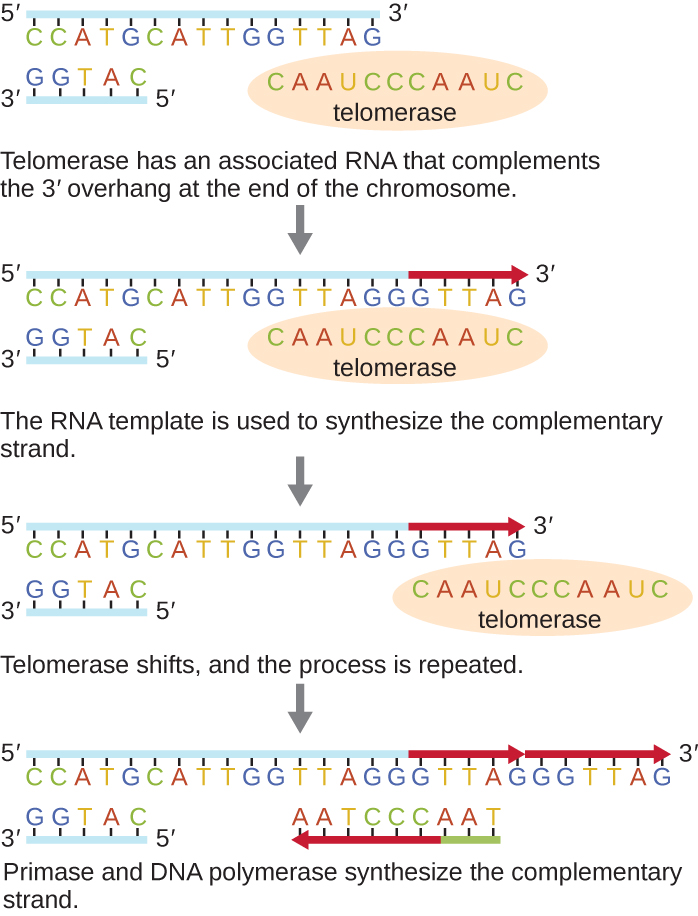
As extremidades dos cromossomos lineares são conhecidas como telômeros e consistem em sequências repetitivas não codificantes. Os telômeros protegem as sequências de codificação de serem perdidas à medida que as células continuam a se dividir. Em humanos, uma sequência de seis pares de bases, TTAGGG, é repetida 100 a 1000 vezes para formar o telômero. A descoberta da enzima telomerase (Figura\(\PageIndex{6}\)) esclareceu nossa compreensão de como as extremidades dos cromossomos são mantidas. A telomerase contém uma parte catalítica e um modelo de RNA embutido. Ele se liga ao final do cromossomo e bases complementares ao modelo de RNA são adicionadas na extremidade 3' da fita de DNA. Uma vez que a extremidade de 3 pés do modelo de fita defasada é suficientemente alongada, a DNA polimerase pode adicionar nucleotídeos complementares às extremidades dos cromossomos. Dessa forma, as extremidades dos cromossomos são replicadas. Em humanos, a telomerase é tipicamente ativa em células germinativas e células-tronco adultas; não é ativa em células somáticas adultas e pode estar associada ao envelhecimento dessas células. Micróbios eucarióticos, incluindo fungos e protozoários, também produzem telomerase para manter a integridade cromossômica. Por sua descoberta da telomerase e sua ação, Elizabeth Blackburn (1948—) recebeu o Prêmio Nobel de Medicina ou Fisiologia em 2009.
| Propriedade | Bactérias | Eucariotos |
|---|---|---|
| Estrutura do genoma | Cromossomo circular único | Vários cromossomos lineares |
| Número de origens por cromossomo | Solteiro | Múltiplos |
| Taxa de replicação | 1000 nucleotídeos por segundo | 100 nucleotídeos por segundo |
| Telomerase | Não presente | Presente |
| Remoção de primer de RNA | Pesquisa de DNA I | RNase H |
| Alongamento do fio | Pesquisa de DNA III | pol δ, pol ε |
Exercício\(\PageIndex{3}\)
- Como a origem da replicação difere entre eucariotos e procariontes?
- Quais enzimas da polimerase são responsáveis pela síntese de DNA durante a replicação eucariótica?
- O que é encontrado nas extremidades dos cromossomos nos eucariotos e por quê?
Replicação de DNA de elementos extracromossômicos: plasmídeos e vírus
Para copiar seus ácidos nucléicos, plasmídeos e vírus freqüentemente usam variações no padrão de replicação do DNA descrito para genomas de procariontes. Para obter mais informações sobre a ampla variedade de estratégias de replicação viral, consulte O ciclo de vida viral.
Replicação em círculo rol
Enquanto muitos plasmídeos bacterianos (veja Características Únicas das Células Procarióticas) se replicam por um processo semelhante ao usado para copiar o cromossomo bacteriano, outros plasmídeos, vários bacteriófagos e alguns vírus de eucariotos usam replicação em círculo contínuo (Figura\(\PageIndex{7}\)). A natureza circular dos plasmídeos e a circularização de alguns genomas virais na infecção tornam isso possível. A replicação do círculo rolante começa com o corte enzimático de uma fita da molécula circular de fita dupla no local de origem de fita dupla (dso). Nas bactérias, a DNA polimerase III se liga ao grupo 3'-OH da fita cortada e começa a replicar unidirecionalmente o DNA usando a fita não cortada como molde, deslocando a fita cortada à medida que o faz. A conclusão da replicação do DNA no local do corte original resulta no deslocamento total da fita cortada, que pode então se recircularizar em uma molécula de DNA de fita simples. A RNA primase então sintetiza um primer para iniciar a replicação do DNA no local de origem de fita simples (sso) da molécula de DNA de fita simples (ssDNA), resultando em uma molécula de DNA de fita dupla (dsDNA) idêntica à outra molécula circular de DNA.
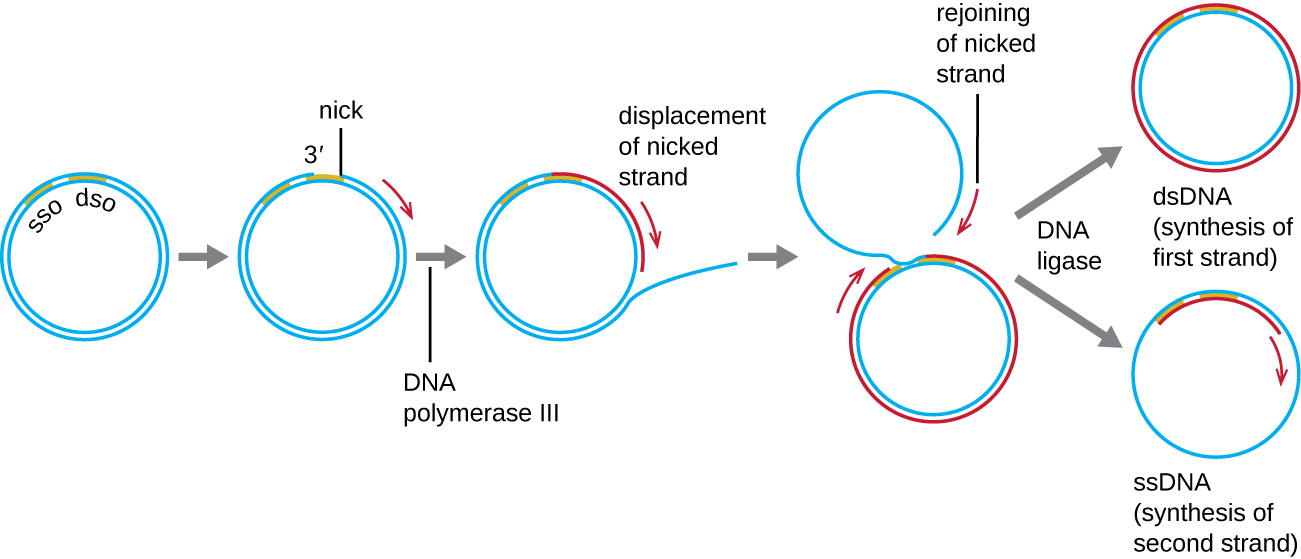
Exercício\(\PageIndex{4}\)
Existe uma vertente atrasada na replicação de círculos ondulantes? Por que ou por que não?
Conceitos principais e resumo
- O processo de replicação do DNA é semiconservador, o que resulta em duas moléculas de DNA, cada uma com uma fita parental de DNA e uma fita recém-sintetizada.
- Nas bactérias, o início da replicação ocorre na origem da replicação, onde o DNA superenrolado é desenrolado pela DNA girase, feito de fita simples pela helicase e ligado por proteína de ligação de fita simples para manter sua única estado encalhado. A Primase sintetiza um primer de RNA curto, fornecendo um grupo 3'-OH livre ao qual a DNA polimerase III pode adicionar nucleotídeos de DNA.
- Durante o alongamento, a fita principal do DNA é sintetizada continuamente a partir de um único primer. A fita defasada é sintetizada de forma descontínua em pequenos fragmentos de Okazaki, cada um exigindo seu próprio primer. Os primers de RNA são removidos e substituídos por nucleotídeos de DNA pela DNA polimerase I bacteriana, e a DNA ligase sela as lacunas entre esses fragmentos.
- O término da replicação em bactérias envolve a resolução de concatêmeros circulares de DNA pela topoisomerase IV para liberar as duas cópias do cromossomo circular.
- Os eucariotos normalmente têm vários cromossomos lineares, cada um com várias origens de replicação. No geral, a replicação em eucariotos é semelhante à dos procariontes.
- A natureza linear dos cromossomos eucarióticos requer telômeros para proteger os genes próximos ao final dos cromossomos. A telomerase estende os telômeros, impedindo sua degradação, em alguns tipos de células.
- A replicação em círculo rolante é um tipo de síntese rápida de DNA unidirecional de uma molécula de DNA circular usada para a replicação de alguns plasmídeos.


