12.1: Micróbios e as ferramentas da engenharia genética
- Page ID
- 181708
Objetivos de
- Identifique ferramentas de genética molecular derivadas de microrganismos
- Descreva os métodos usados para criar moléculas de DNA recombinante
- Descreva os métodos usados para introduzir DNA em células procarióticas
- Liste os tipos de bibliotecas genômicas e descreva seus usos
- Descreva os métodos usados para introduzir DNA em células eucarióticas
Foco clínico: Parte 1
Kayla, engenheira elétrica de 24 anos e entusiasta da corrida, acabou de se mudar do Arizona para New Hampshire para conseguir um novo emprego. Nos fins de semana de folga, ela adora explorar seus novos arredores, fazendo longas corridas nas florestas de pinheiros. Em julho, ela passou uma semana caminhando pelas montanhas. No início de agosto, Kayla desenvolveu febre baixa, dor de cabeça e dores musculares leves, e ela se sentiu um pouco cansada. Sem pensar muito nisso, ela tomou um pouco de ibuprofeno para combater seus sintomas e prometeu descansar mais.
Exercício\(\PageIndex{1}\)
Quais tipos de condições médicas podem ser responsáveis pelos sintomas de Kayla?
A ciência de usar sistemas vivos para beneficiar a humanidade é chamada de biotecnologia. Tecnicamente falando, a domesticação de plantas e animais por meio de práticas agrícolas e de criação é um tipo de biotecnologia. No entanto, em um sentido contemporâneo, associamos a biotecnologia à alteração direta da genética de um organismo para alcançar características desejáveis por meio do processo de engenharia genética. A engenharia genética envolve o uso da tecnologia de DNA recombinante, o processo pelo qual uma sequência de DNA é manipulada in vitro, criando moléculas de DNA recombinante que têm novas combinações de material genético. O DNA recombinante é então introduzido em um organismo hospedeiro. Se o DNA introduzido vem de uma espécie diferente, o organismo hospedeiro agora é considerado transgênico.
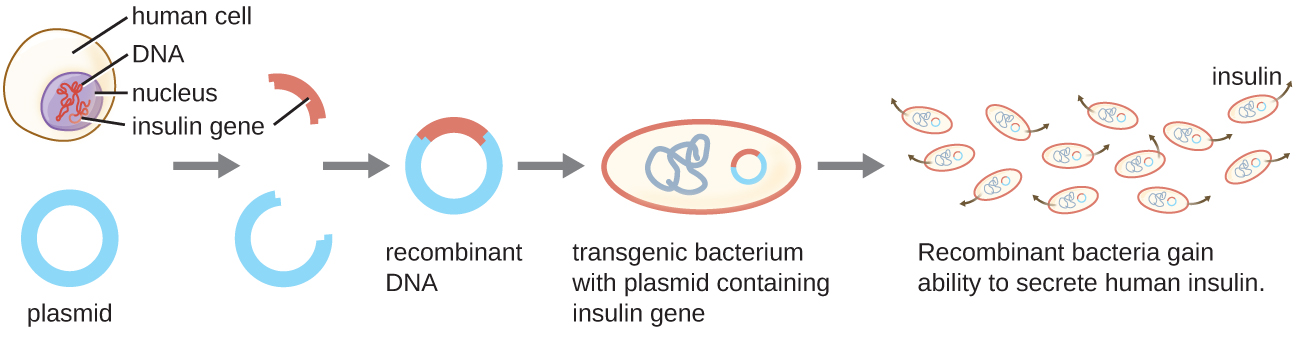
Um exemplo de microrganismo transgênico é a cepa bacteriana que produz insulina humana (Figura\(\PageIndex{1}\)). O gene da insulina de humanos foi inserido em um plasmídeo. Esse plasmídeo de DNA recombinante foi então inserido nas bactérias. Como resultado, esses micróbios transgênicos são capazes de produzir e secretar insulina humana. Muitos procariontes são capazes de adquirir DNA estranho e incorporar genes funcionais em seu próprio genoma por meio do “acasalamento” com outras células (conjugação), infecção viral (transdução) e captação de DNA do meio ambiente (transformação). Lembre-se de que esses mecanismos são exemplos de transferência horizontal de genes - a transferência de material genético entre células da mesma geração.
Clonagem molecular
Herbert Boyer e Stanley Cohen demonstraram pela primeira vez o processo completo de clonagem molecular em 1973, quando clonaram com sucesso genes do sapo africano com garras (Xenopus laevis) em um plasmídeo bacteriano que foi então introduzido na hospedeira bacteriana Escherichia coli. A clonagem molecular é um conjunto de métodos usados para construir DNA recombinante e incorporá-lo a um organismo hospedeiro; ela faz uso de várias ferramentas moleculares derivadas de microrganismos.
Enzimas de restrição e ligases
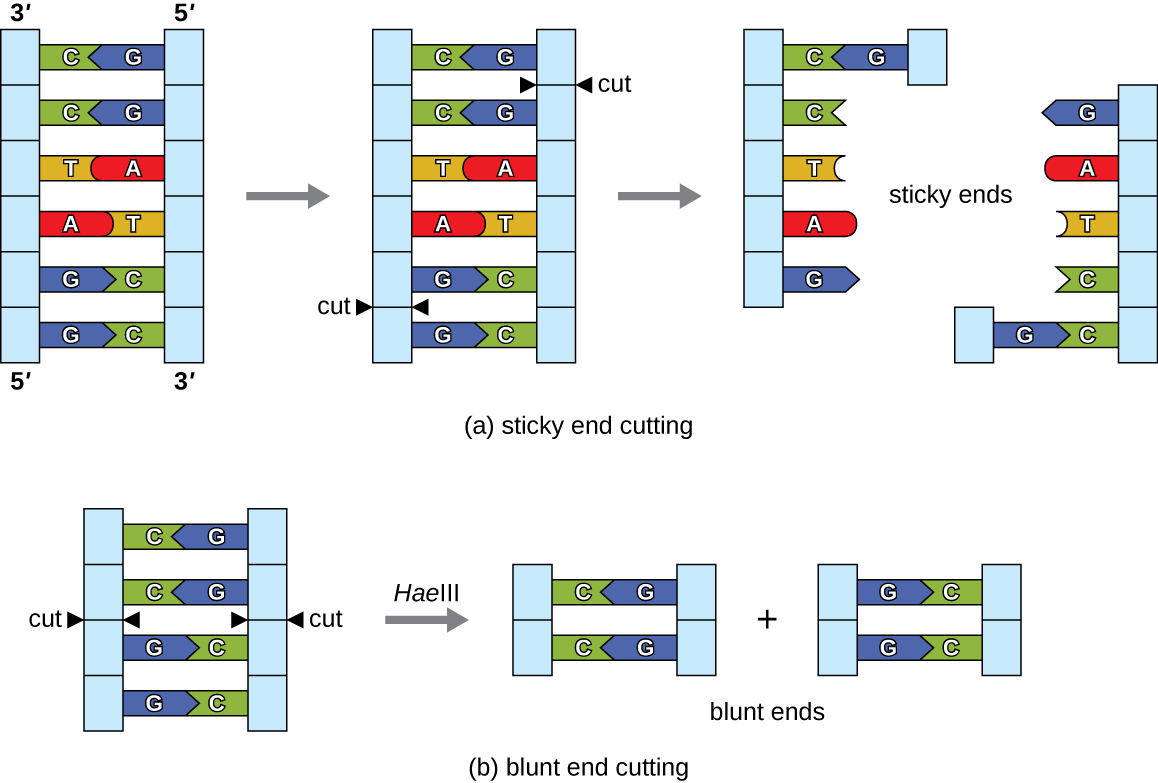
Na tecnologia de DNA recombinante, as moléculas de DNA são manipuladas usando enzimas de ocorrência natural derivadas principalmente de bactérias e vírus. A criação de moléculas de DNA recombinante é possível devido ao uso de endonucleases de restrição naturais (enzimas de restrição), enzimas bacterianas produzidas como um mecanismo de proteção para cortar e destruir o DNA citoplasmático estranho que é mais comumente resultado da infecção por bacteriófagos. Stewart Linn e Werner Arber descobriram enzimas de restrição em seus estudos da década de 1960 sobre como a E. coli limita a replicação de bacteriófagos na infecção. Hoje, usamos enzimas de restrição extensivamente para cortar fragmentos de DNA que podem então ser unidos em outra molécula de DNA para formar moléculas recombinantes. Cada enzima de restrição corta o DNA em um local de reconhecimento característico, uma sequência de DNA específica, geralmente palíndrômica, tipicamente entre quatro a seis pares de bases de comprimento. Um palíndromo é uma sequência de letras que tem a mesma leitura para frente e para trás. (A palavra “nível” é um exemplo de palíndromo.) As sequências de DNA palíndrômico contêm as mesmas sequências de base na direção 5a 3em uma fita e na direção 5a 3na fita complementar. Uma enzima de restrição reconhece o palíndromo de DNA e corta cada coluna vertebral em posições idênticas no palíndromo. Algumas enzimas de restrição cortam para produzir moléculas que têm saliências complementares (extremidades pegajosas), enquanto outras cortam sem gerar tais saliências, em vez disso, produzem pontas sem corte (Figura\(\PageIndex{2}\)).
Moléculas com pontas adesivas complementares podem facilmente se recozer ou formar ligações de hidrogênio entre bases complementares, em suas extremidades pegajosas. A etapa de recozimento permite a hibridização das saliências de fita simples. A hibridização se refere à união de duas fitas únicas complementares de DNA. As pontas sem corte também podem se unir, mas com menos eficiência do que as pontas adesivas devido à falta de saliências complementares que facilitam o processo. Em ambos os casos, a ligação por DNA ligase pode então se juntar novamente às duas espinhas dorsais açúcar-fosfato do DNA por meio de ligação covalente, tornando a molécula uma fita dupla contínua. Em 1972, Paul Berg, bioquímico de Stanford, foi o primeiro a produzir uma molécula de DNA recombinante usando essa técnica, combinando o vírus do macaco SV40 com o bacteriófago lambda E. coli para criar um híbrido.
Plasmídeos
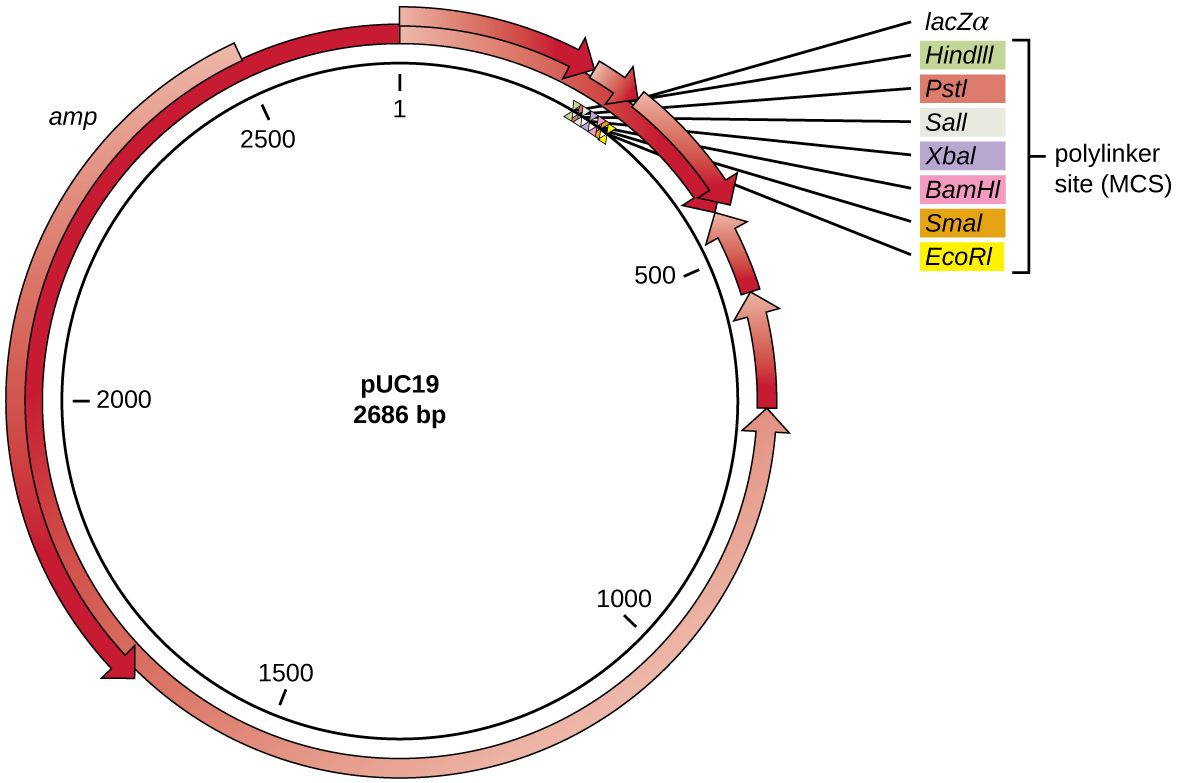
Após a digestão restrita, os genes de interesse são comumente inseridos em plasmídeos, pequenos pedaços de DNA tipicamente circular de fita dupla que se replicam independentemente do cromossomo bacteriano (consulte Características únicas das células procarióticas). Na tecnologia de DNA recombinante, os plasmídeos são frequentemente usados como vetores, moléculas de DNA que transportam fragmentos de DNA de um organismo para outro. Os plasmídeos usados como vetores podem ser geneticamente modificados por pesquisadores e empresas de suprimentos científicos para ter propriedades especializadas, conforme ilustrado pelo vetor de plasmídeo PuC19 comumente usado (Figura\(\PageIndex{3}\)). Alguns vetores de plasmídeo contêm genes que conferem resistência a antibióticos; esses genes de resistência permitem que os pesquisadores encontrem facilmente colônias contendo plasmídeo colocando-as em meios contendo o antibiótico correspondente. O antibiótico mata todas as células hospedeiras que não abrigam o vetor plasmídeo desejado, mas aquelas que contêm o vetor são capazes de sobreviver e crescer.
Os vetores de plasmídeo usados para clonagem geralmente têm um local de polilinagem ou um local de clonagem múltipla (MCS). Um sítio poliginador é uma sequência curta contendo vários locais exclusivos de reconhecimento de enzimas de restrição que são usados para inserir DNA no plasmídeo após a digestão restrita do DNA e do plasmídeo. Ter esses vários locais de reconhecimento de enzimas de restrição dentro do local do poliginker torna o vetor plasmídeo versátil, portanto, ele pode ser usado para muitos experimentos de clonagem diferentes envolvendo diferentes enzimas de restrição.
Esse sítio poliginador é frequentemente encontrado em um gene repórter, outra sequência genética artificialmente projetada no plasmídeo que codifica uma proteína que permite a visualização da inserção de DNA. O gene repórter permite que um pesquisador distinga as células hospedeiras que contêm plasmídeos recombinantes com fragmentos de DNA clonados das células hospedeiras que contêm apenas o vetor plasmídeo não recombinante. O gene repórter mais comum usado em vetores de plasmídeo é o gene bacteriano LacZ que codifica a beta-galactosidase, uma enzima que naturalmente degrada a lactose, mas também pode degradar um análogo sintético incolor X-gal, produzindo colônias azuis em meios contendo X-Gal. O gene repórter LacZ é desativado quando o DNA recombinante é unido ao plasmídeo. Como a proteína LacZ não é produzida quando o gene é desativado, a X-gal não é degradada e colônias brancas são produzidas, que podem então ser isoladas. Esse método de triagem azul-branco é descrito posteriormente e mostrado na Figura\(\PageIndex{4}\). Além dessas características, alguns plasmídeos vêm pré-digeridos e com uma enzima ligada ao plasmídeo linearizado para auxiliar na ligação após a inserção de fragmentos estranhos de DNA.
Clonagem molecular usando transformação
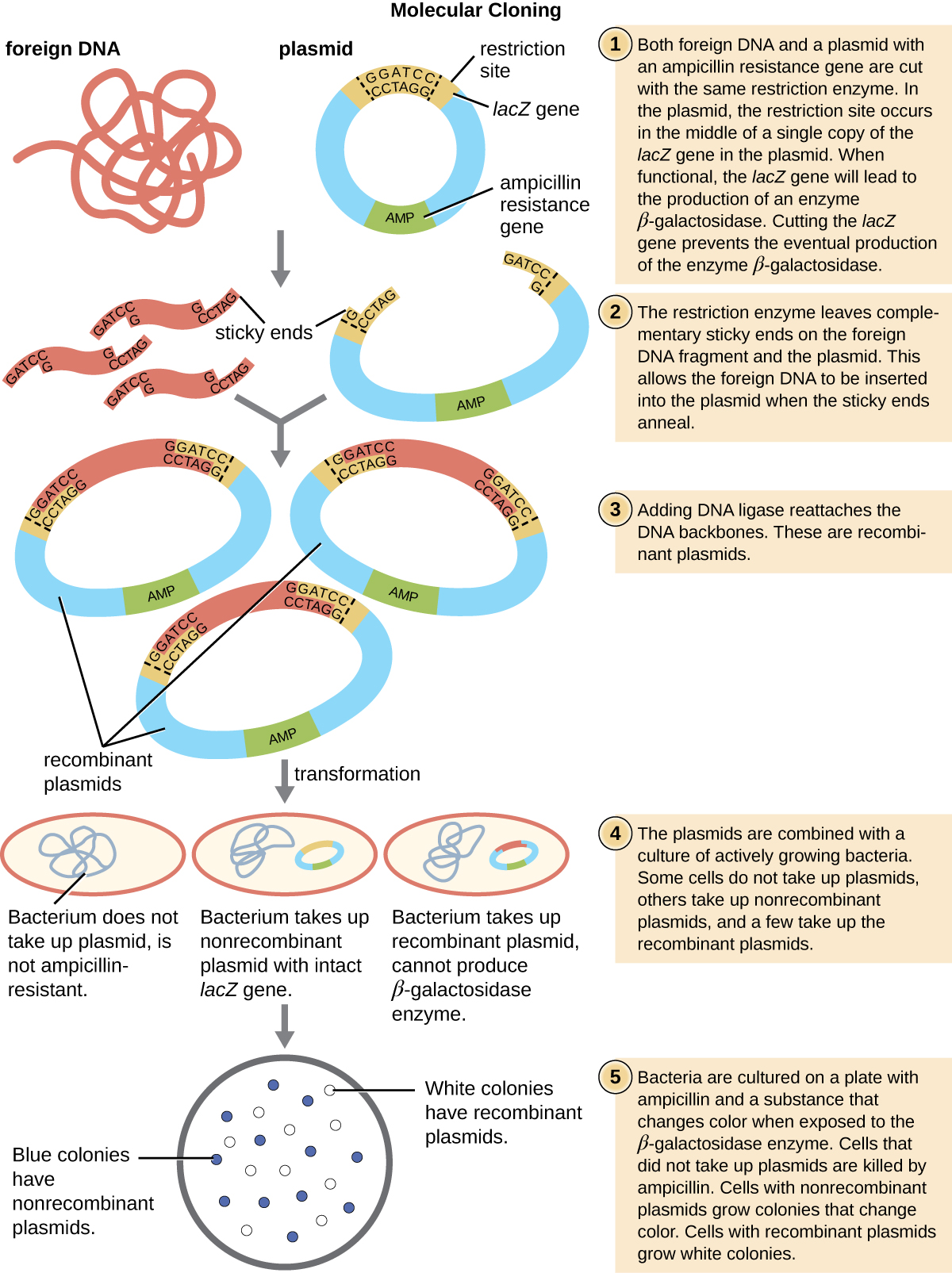
O mecanismo mais comumente usado para introduzir plasmídeos modificados em uma célula bacteriana é a transformação, um processo no qual as bactérias absorvem DNA livre de seus arredores. Na natureza, o DNA livre normalmente vem de outras células bacterianas lisadas; no laboratório, o DNA livre na forma de plasmídeos recombinantes é introduzido no entorno da célula.
Algumas bactérias, como Bacillus spp., são naturalmente competentes, o que significa que são capazes de captar DNA estranho. No entanto, nem todas as bactérias são naturalmente competentes. Na maioria dos casos, as bactérias devem se tornar artificialmente competentes em laboratório, aumentando a permeabilidade da membrana celular. Isso pode ser obtido por meio de tratamentos químicos que neutralizam as cargas na membrana celular ou expondo a bactéria a um campo elétrico que cria poros microscópicos na membrana celular. Esses métodos produzem bactérias quimicamente competentes ou eletrocompetentes, respectivamente.
Seguindo o protocolo de transformação, as células bacterianas são colocadas em um meio contendo antibióticos para inibir o crescimento de muitas células hospedeiras que não foram transformadas pelo plasmídeo que confere resistência aos antibióticos. Uma técnica chamada triagem azul-branca é então usada para vetores de plasmídeo codificadores de lacZ, como PuC19. As colônias azuis têm uma enzima beta-galactosidase funcional porque o gene LacZ é ininterrupto, sem DNA estranho inserido no local do polinker. Essas colônias normalmente resultam da religação do plasmídeo linearizado e digerido para si mesmo. No entanto, as colônias brancas não possuem uma enzima beta-galactosidase funcional, indicando a inserção de DNA estranho no sítio poligante do vetor plasmídeo, interrompendo assim o gene lacZ. Assim, as colônias brancas resultantes dessa triagem azul-branca contêm plasmídeos com uma inserção e podem ser rastreadas posteriormente para caracterizar o DNA estranho. Para ter certeza de que o DNA correto foi incorporado ao plasmídeo, o inserto de DNA pode então ser sequenciado.
Veja uma animação da clonagem molecular do DNA Learning Center.
Exercício\(\PageIndex{2}\)
Na exibição azul-branca, o que significa uma colônia azul e por que ela é azul?
Clonagem molecular usando conjugação ou transdução
O processo bacteriano de conjugação (veja Como os procariontes assexuais alcançam a diversidade genética) também pode ser manipulado para clonagem molecular. Os plasmídeos F, ou plasmídeos de fertilidade, são transferidos entre as células bacterianas por meio do processo de conjugação. O DNA recombinante pode ser transferido por conjugação quando células bacterianas contendo um plasmídeo F recombinante são misturadas com células bacterianas compatíveis sem o plasmídeo. Os plasmídeos F codificam uma estrutura de superfície chamada F pilus que facilita o contato entre uma célula contendo um plasmídeo F e outra sem plasmídeo F. Em contato, uma ponte citoplasmática se forma entre as duas células e a célula contendo plasmídeo F replica seu plasmídeo, transferindo uma cópia do plasmídeo F recombinante para a célula receptora. Depois de receber o plasmídeo F recombinante, a célula receptora pode produzir seu próprio pilus F e facilitar a transferência do plasmídeo F recombinante para uma célula adicional. O uso da conjugação para transferir plasmídeos F recombinantes para as células receptoras é outra forma eficaz de introduzir moléculas de DNA recombinante nas células hospedeiras.
Alternativamente, os bacteriófagos podem ser usados para introduzir DNA recombinante nas células bacterianas hospedeiras por meio da manipulação do processo de transdução (veja Como os procariontes assexuais alcançam a diversidade genética). No laboratório, fragmentos de DNA de interesse podem ser transformados em fagêmides, que são plasmídeos que têm sequências de fagos que permitem que sejam empacotados em bacteriófagos. As células bacterianas podem então ser infectadas com esses bacteriófagos para que os fagêmides recombinantes possam ser introduzidos nas células bacterianas. Dependendo do tipo de fago, o DNA recombinante pode ser integrado ao genoma bacteriano do hospedeiro (lisogenia) ou pode existir como um plasmídeo no citoplasma do hospedeiro.
Exercício\(\PageIndex{2}\)
- Qual é a função original de uma enzima de restrição?
- Quais são os dois processos explorados para obter DNA recombinante em uma célula hospedeira bacteriana?
- Distinguir os usos de um gene de resistência a antibióticos e de um gene repórter em um vetor de plasmídeo.
Criando uma biblioteca genômica
A clonagem molecular também pode ser usada para gerar uma biblioteca genômica. A biblioteca é uma cópia completa (ou quase completa) do genoma de um organismo contida como plasmídeos de DNA recombinante transformados em clones únicos de bactérias. Ter essa biblioteca permite que um pesquisador crie grandes quantidades de cada fragmento cultivando o hospedeiro bacteriano desse fragmento. Esses fragmentos podem ser usados para determinar a sequência do DNA e a função de qualquer gene presente.
Um método para gerar uma biblioteca genômica é ligar fragmentos genômicos digeridos pela enzima de restrição individual em vetores de plasmídeo cortados com a mesma enzima de restrição (Figura\(\PageIndex{5}\)). Após a transformação em um hospedeiro bacteriano, cada célula bacteriana transformada ocupa um único plasmídeo recombinante e se transforma em uma colônia de células. Todas as células desta colônia são clones idênticos e carregam o mesmo plasmídeo recombinante. A biblioteca resultante é uma coleção de colônias, cada uma contendo um fragmento do genoma original do organismo, que são separadas e distintas e podem ser usadas para estudos adicionais. Isso possibilita que os pesquisadores rastreiem esses diferentes clones para descobrir aquele que contém um gene de interesse do genoma original do organismo.
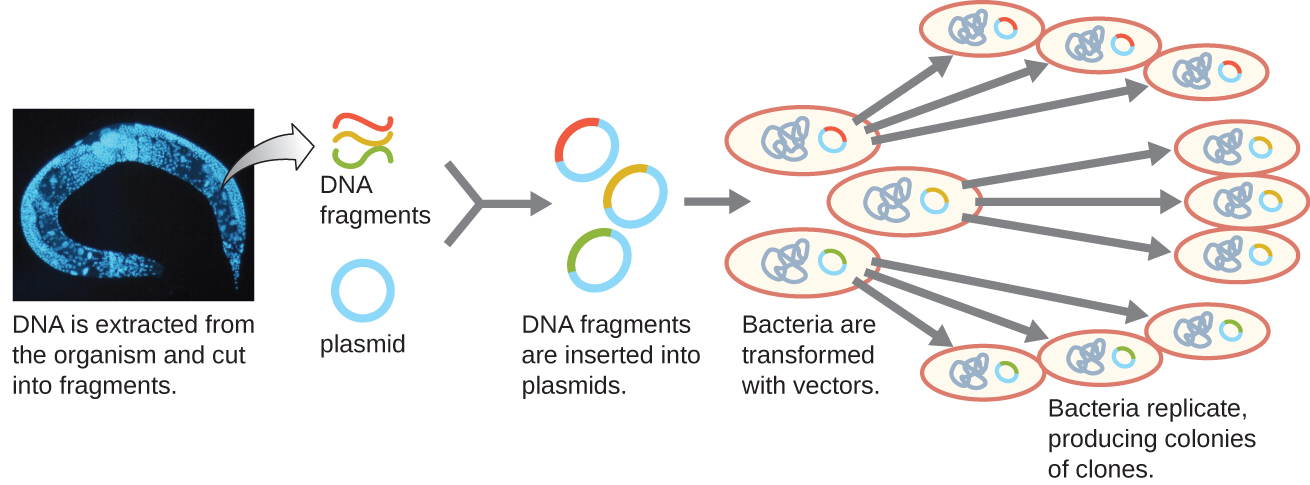
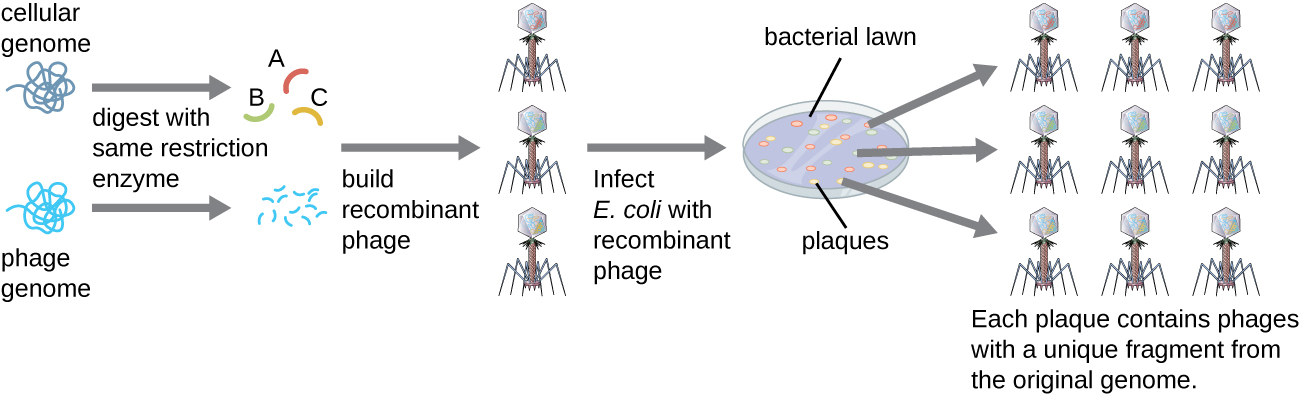
Para construir uma biblioteca genômica usando fragmentos maiores de DNA genômico, um bacteriófago de E. coli, como lambda, pode ser usado como hospedeiro (Figura\(\PageIndex{6}\)). O DNA genômico pode ser cortado ou digerido enzimaticamente e ligado a um vetor de DNA lambda do bacteriófago pré-digerido. Em seguida, essas moléculas de DNA de fagos recombinantes podem ser empacotadas em partículas de fago e usadas para infectar células hospedeiras de E. coli em uma placa. Durante a infecção dentro de cada célula, cada fago recombinante fará muitas cópias de si mesmo e lisará o gramado de E. coli, formando uma placa. Assim, cada placa de uma biblioteca de fagos representa um fago recombinante exclusivo contendo um fragmento de DNA genômico distinto. As placas podem então ser examinadas posteriormente para procurar genes de interesse. Uma vantagem de produzir uma biblioteca usando fagos em vez de plasmídeos é que uma partícula de fago contém uma inserção muito maior de DNA estranho em comparação com um vetor de plasmídeo, exigindo assim um número muito menor de culturas para representar totalmente todo o genoma do organismo original.
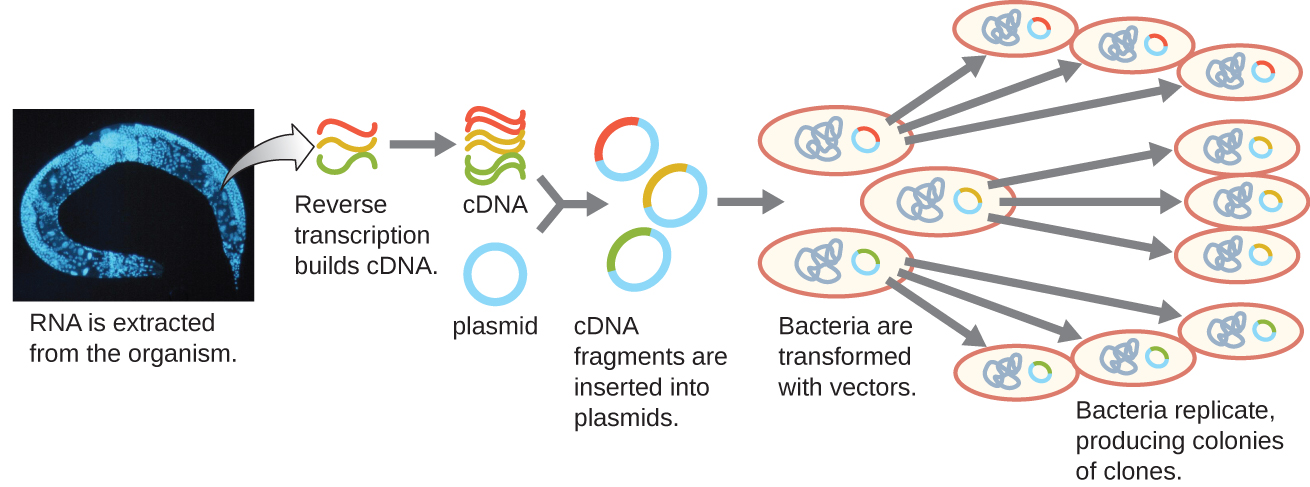
Para focar nos genes expressos em um organismo ou mesmo em um tecido, os pesquisadores constroem bibliotecas usando o RNA mensageiro (mRNA) do organismo em vez de seu DNA genômico. Enquanto todas as células de um único organismo terão o mesmo DNA genômico, tecidos diferentes expressam genes diferentes, produzindo diferentes complementos de mRNA. Por exemplo, o DNA genômico de todas as células humanas contém o gene da insulina, mas somente as células do pâncreas expressam o mRNA direcionando a produção de insulina. Como o mRNA não pode ser clonado diretamente, no laboratório, o mRNA deve ser usado como modelo pela enzima retroviral transcriptase reversa para produzir DNA complementar (cDNA). O complemento completo de mRNA de uma célula pode ser transcrito reversamente em moléculas de cDNA, que podem ser usadas como um modelo para a DNA polimerase fazer cópias de DNA de fita dupla; esses fragmentos podem ser posteriormente ligados a vetores de plasmídeo ou bacteriófago para produzir uma biblioteca de cDNA. O benefício de uma biblioteca de cDNA é que ela contém DNA apenas dos genes expressos na célula. Isso significa que os íntrons, as sequências de controle, como promotores, e o DNA não destinado a ser traduzido em proteínas não estão representados na biblioteca. O foco nas sequências traduzidas significa que a biblioteca não pode ser usada para estudar a sequência e a estrutura do genoma em sua totalidade. A construção de uma biblioteca genômica de cDNA é mostrada na Figura\(\PageIndex{7}\).
Exercício\(\PageIndex{3}\)
- Quais são os hospedeiros das bibliotecas genômicas descritas?
- O que é cDNA?
Introdução de moléculas recombinantes em hospedeiros eucarióticos
O uso de hospedeiros bacterianos para engenharia genética lançou as bases para a tecnologia de DNA recombinante; no entanto, os pesquisadores também tiveram grande interesse na engenharia genética de células eucarióticas, particularmente aquelas de plantas e animais. A introdução de moléculas de DNA recombinante em hospedeiros eucarióticos é chamada de transfecção. Plantas geneticamente modificadas, chamadas de plantas transgênicas, são de grande interesse para fins agrícolas e farmacêuticos. A primeira planta transgênica vendida comercialmente foi o tomate de amadurecimento retardado Flavr Savr, que chegou ao mercado em 1994. O gado geneticamente modificado também foi produzido com sucesso, resultando, por exemplo, em porcos com maior valor nutricional 1 e cabras que secretam produtos farmacêuticos em seu leite. 2
Eletroporação
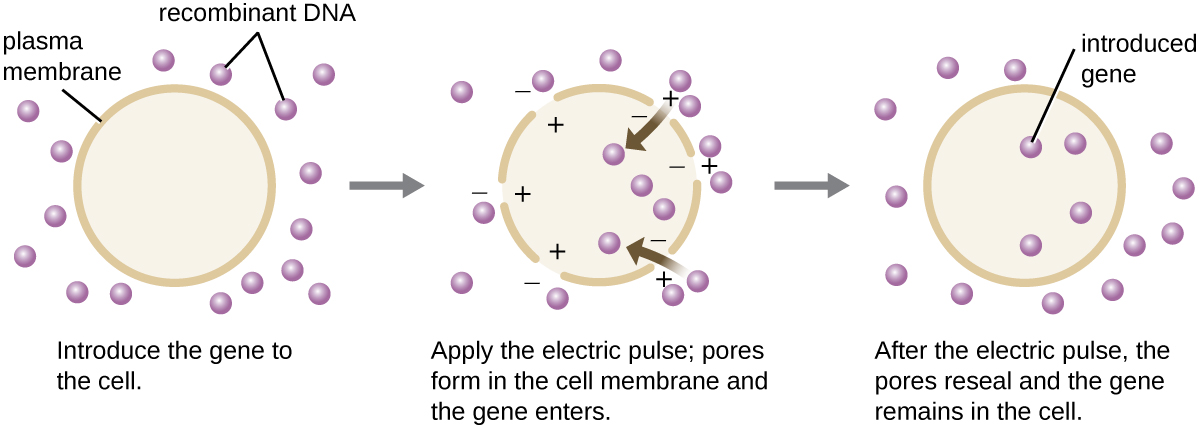
Em comparação com as células bacterianas, as células eucarióticas tendem a ser menos receptivas como hospedeiras para moléculas de DNA recombinante. Como os eucariotos normalmente não são competentes para captar DNA estranho nem são capazes de manter plasmídeos, a transfecção de hospedeiros eucarióticos é muito mais desafiadora e requer técnicas mais intrusivas para o sucesso. Um método usado para transfectar células em cultura celular é chamado de eletroporação. Um breve pulso elétrico induz a formação de poros transitórios nas bicamadas fosfolipídicas das células através das quais o gene pode ser introduzido. Ao mesmo tempo, o pulso elétrico gera uma carga positiva de curta duração em um lado do interior da célula e uma carga negativa no lado oposto; a diferença de carga atrai moléculas de DNA carregadas negativamente para dentro da célula (Figura\(\PageIndex{8}\)).
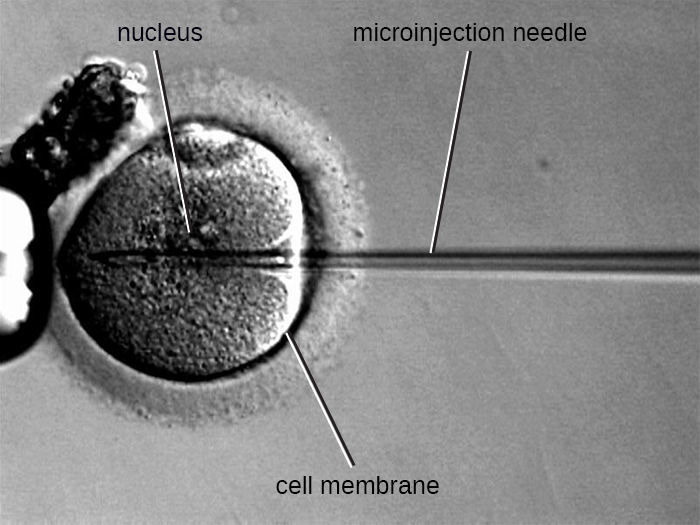
Microinjeção
Um método alternativo de transfecção é chamado de microinjeção. Como as células eucarióticas são normalmente maiores do que as dos procariontes, fragmentos de DNA às vezes podem ser injetados diretamente no citoplasma usando uma micropipeta de vidro, conforme mostrado na Figura\(\PageIndex{9}\).
Armas genéticas
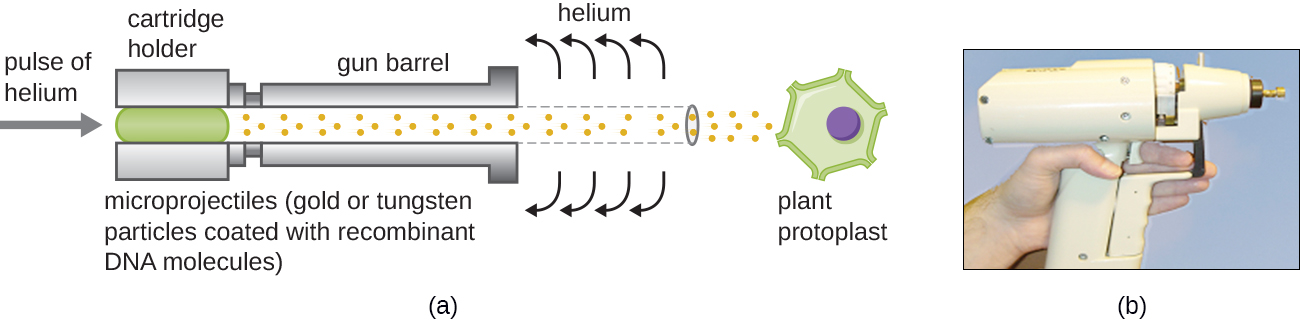
Transfectar células vegetais pode ser ainda mais difícil do que células animais por causa de suas paredes celulares espessas. Uma abordagem envolve o tratamento de células vegetais com enzimas para remover suas paredes celulares, produzindo protoplastos. Em seguida, uma pistola genética é usada para disparar partículas de ouro ou tungstênio revestidas com moléculas de DNA recombinante nos protoplastos da planta em alta velocidade. As células protoplastáticas receptoras podem então se recuperar e ser usadas para gerar novas plantas transgênicas (Figura\(\PageIndex{10}\)).
Vetores de ônibus
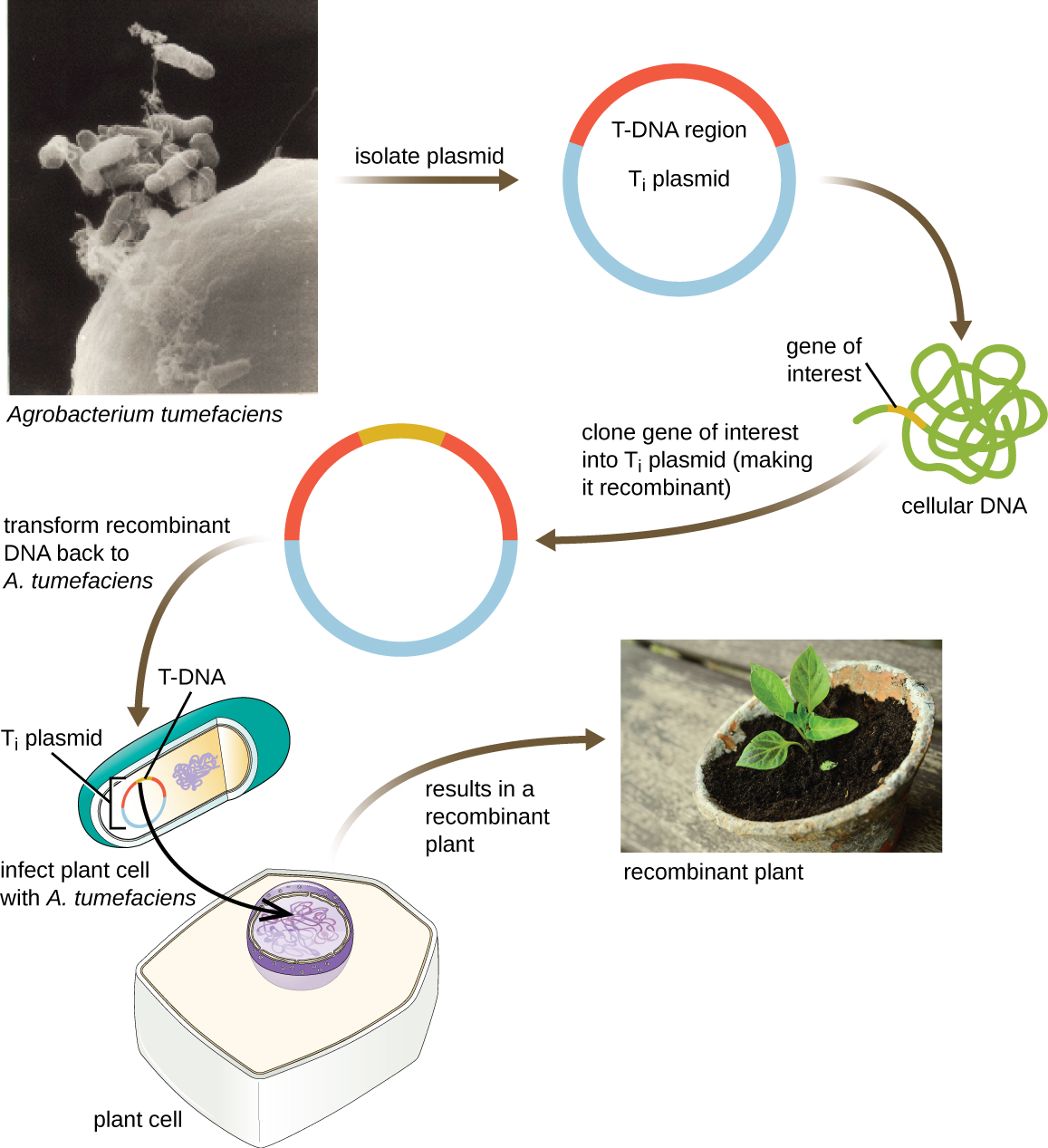
Outro método de transfecção de plantas envolve vetores de transporte, plasmídeos que podem se mover entre células bacterianas e eucarióticas. Os plasmídeos indutores de tumor (T i) originários da bactéria Agrobacterium tumefaciens são comumente usados como vetores auxiliares para incorporar genes às plantas (Figura\(\PageIndex{11}\)). Na natureza, os plasmídeos T i de A. tumefaciens fazem com que as plantas desenvolvam tumores quando são transferidas das células bacterianas para as células vegetais. Os pesquisadores conseguiram manipular esses plasmídeos naturais para remover seus genes causadores de tumores e inserir fragmentos de DNA desejáveis. Os plasmídeos T i recombinantes resultantes podem ser transferidos para o genoma da planta por meio da transferência natural de plasmídeos T i da bactéria para a planta hospedeira. Uma vez dentro da célula hospedeira da planta, o gene de interesse se recombina com o genoma da célula vegetal.
Vetores virais
Vetores virais também podem ser usados para transfectar células eucarióticas. Na verdade, esse método é frequentemente usado na terapia gênica (consulte Terapia gênica) para introduzir genes saudáveis em pacientes humanos que sofrem de doenças que resultam de mutações genéticas. Os genes virais podem ser excluídos e substituídos pelo gene a ser entregue ao paciente; 3 o vírus então infecta a célula hospedeira e entrega o DNA estranho ao genoma da célula-alvo. Os adenovírus são frequentemente usados para essa finalidade porque podem ser cultivados até um título alto e podem infectar células hospedeiras não divididas e divididas. No entanto, o uso de vetores virais para terapia gênica pode representar alguns riscos para os pacientes, conforme discutido em Terapia Gênica.
Exercício\(\PageIndex{4}\)
- Quais são os métodos usados para introduzir vetores de DNA recombinante em células animais?
- Compare e contraste vetores de transporte e vetores virais.
Conceitos principais e resumo
- A biotecnologia é a ciência da utilização de sistemas vivos para beneficiar a humanidade. Nos últimos anos, a capacidade de alterar diretamente o genoma de um organismo por meio da engenharia genética foi possível devido aos avanços na tecnologia de DNA recombinante, que permite aos pesquisadores criar moléculas de DNA recombinante com novas combinações de material genético.
- A clonagem molecular envolve métodos usados para construir DNA recombinante e facilitar sua replicação em organismos hospedeiros. Esses métodos incluem o uso de enzimas de restrição (para cortar vetores estranhos de DNA e plasmídeo), ligadura (para colar fragmentos de DNA juntos) e a introdução de DNA recombinante em um organismo hospedeiro (geralmente bactérias).
- A triagem azul-branca permite a seleção de transformantes bacterianos que contêm plasmídeos recombinantes usando o fenótipo de um gene repórter que é desativado pela inserção do fragmento de DNA.
- As bibliotecas genômicas podem ser criadas clonando fragmentos genômicos de um organismo em vetores de plasmídeos ou em bacteriófagos.
- Bibliotecas de cDNA podem ser geradas para representar as moléculas de mRNA expressas em uma célula em um determinado ponto.
- A transfecção de hospedeiros eucarióticos pode ser obtida por meio de vários métodos usando eletroporação, armas genéticas, microinjeção, vetores de transporte e vetores virais.
Notas de pé
- 1 Liangxue Lai, Jing X. Kang, Rongfeng Li, Jingdong Wang, William T. Witt, Hwan Yul Yong, Yanhong Hao e outros. “Geração de porcos transgênicos clonados ricos em ácidos graxos ômega-3.” Nature Biotechnology 24 no. 4 (2006): 435—436.
- 2 Raylene Ramos Moura, Luciana Magalhães Melo e Vicente José de Figueirêdo Freitas. “Produção de proteínas recombinantes no leite de cabras transgênicas e não transgênicas.” Arquivos Brasileiros de Biologia e Tecnologia 54 no. 5 (2011): 927—938.
- 3 William S.M. Wold e Karoly Toth. “Vetores de adenovírus para terapia gênica, vacinação e terapia gênica do câncer.” Terapia gênica atual 13 no. 6 (2013): 421.


