11.7: Regulação gênica - Teoria dos Operons
- Page ID
- 181327
objetivos de aprendizagem
- Compare operons indutíveis e operons reprimíveis
- Descreva por que a regulação dos operons é importante
Cada célula nucleada em um organismo multicelular contém cópias do mesmo DNA. Da mesma forma, todas as células em duas culturas bacterianas puras inoculadas da mesma colônia inicial contêm o mesmo DNA, com exceção das alterações decorrentes de mutações espontâneas. Se cada célula de um organismo multicelular tem o mesmo DNA, então como é que células em diferentes partes do corpo do organismo exibem características diferentes? Da mesma forma, como é que as mesmas células bacterianas em duas culturas puras expostas a diferentes condições ambientais podem exibir fenótipos diferentes? Em ambos os casos, cada célula geneticamente idêntica não ativa nem expressa o mesmo conjunto de genes. Somente um subconjunto de proteínas em uma célula em um determinado momento é expresso.
O DNA genômico contém genes estruturais, que codificam produtos que servem como estruturas celulares ou enzimas, e genes regulatórios, que codificam produtos que regulam a expressão gênica. A expressão de um gene é um processo altamente regulado. Enquanto a regulação da expressão gênica em organismos multicelulares permite a diferenciação celular, em organismos unicelulares, como procariontes, ela garante principalmente que os recursos de uma célula não sejam desperdiçados produzindo proteínas de que a célula não precisa naquele momento.
Elucidar os mecanismos que controlam a expressão gênica é importante para a compreensão da saúde humana. O mau funcionamento desse processo em humanos leva ao desenvolvimento de câncer e outras doenças. Compreender a interação entre a expressão gênica de um patógeno e a de seu hospedeiro humano é importante para a compreensão de uma doença infecciosa específica. A regulação gênica envolve uma complexa rede de interações dentro de uma determinada célula entre sinais do ambiente da célula, moléculas sinalizadoras dentro da célula e o DNA da célula. Essas interações levam à expressão de alguns genes e à supressão de outros, dependendo das circunstâncias.
Procariontes e eucariotos compartilham algumas semelhanças em seus mecanismos para regular a expressão gênica; no entanto, a expressão gênica em eucariotos é mais complicada devido à separação temporal e espacial entre os processos de transcrição e tradução. Assim, embora a maior parte da regulação da expressão gênica ocorra por meio do controle transcricional em procariontes, a regulação da expressão gênica em eucariotos ocorre no nível transcricional e pós-transcricional (após a transcrição primária ter sido feita).
Regulação do gene procariótico
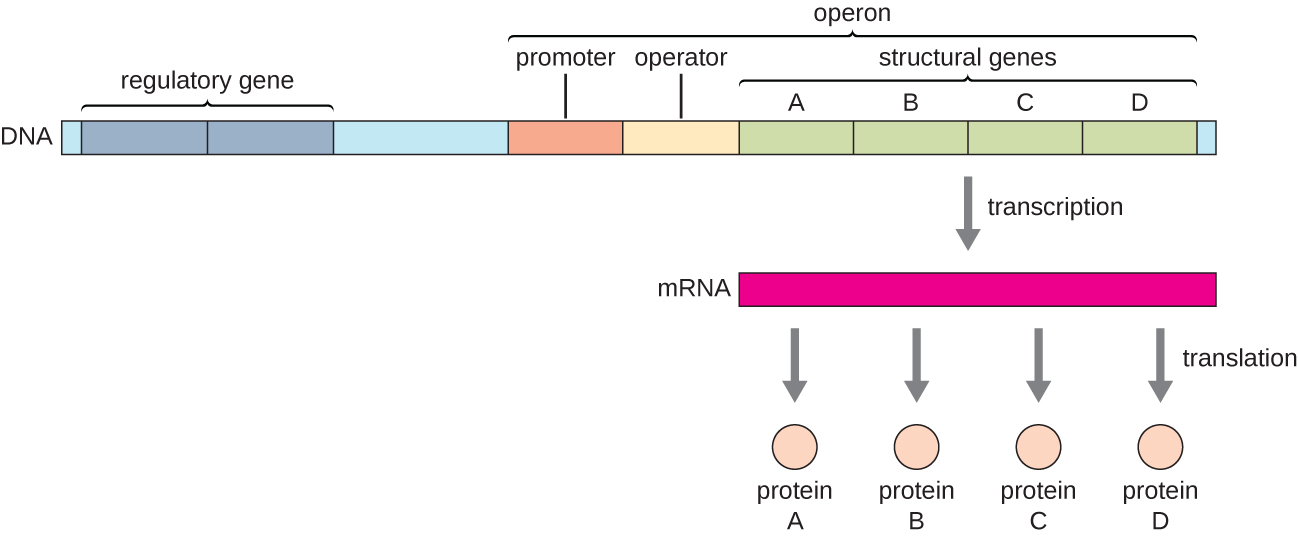
Em bactérias e arquéias, proteínas estruturais com funções relacionadas geralmente são codificadas juntas dentro do genoma em um bloco chamado operon e são transcritas juntas sob o controle de um único promotor, resultando na formação de uma transcrição policistronica (Figura\(\PageIndex{1}\)). Dessa forma, a regulação da transcrição de todos os genes estruturais que codificam as enzimas que catalisam as várias etapas em uma única via bioquímica pode ser controlada simultaneamente, porque todos serão necessários ao mesmo tempo ou nenhum será necessário. Por exemplo, em E. coli, todos os genes estruturais que codificam as enzimas necessárias para usar a lactose como fonte de energia estão próximos uns dos outros no operon lactose (ou lac) sob o controle de um único promotor, o promotor lac. Os cientistas franceses François Jacob (1920—2013) e Jacques Monod, do Instituto Pasteur, foram os primeiros a mostrar a organização de genes bacterianos em operons, por meio de seus estudos sobre o operon lac de E. coli. Por esse trabalho, eles ganharam o Prêmio Nobel de Fisiologia ou Medicina em 1965. Embora os genes eucarióticos não estejam organizados em operons, os operons procarióticos são excelentes modelos para aprender sobre a regulação gênica em geral. Existem alguns grupos de genes em eucariotos que funcionam de forma semelhante aos operons. Muitos dos princípios podem ser aplicados a sistemas eucarióticos e contribuir para nossa compreensão das mudanças na expressão gênica em eucariotos que podem resultar em alterações patológicas, como câncer.
Cada operon inclui sequências de DNA que influenciam sua própria transcrição; elas estão localizadas em uma região chamada região regulatória. A região reguladora inclui o promotor e a região ao redor do promotor, à qual os fatores de transcrição, proteínas codificadas por genes reguladores, podem se ligar. Os fatores de transcrição influenciam a ligação da RNA polimerase ao promotor e permitem que sua progressão transcreva genes estruturais. Um repressor é um fator de transcrição que suprime a transcrição de um gene em resposta a um estímulo externo por meio da ligação a uma sequência de DNA dentro da região regulatória chamada operador, que está localizada entre o sítio de ligação da RNA polimerase do promotor e o local inicial da transcrição do primeiro gene estrutural. A ligação ao repressor impede fisicamente a RNA polimerase de transcrever genes estruturais. Por outro lado, um ativador é um fator de transcrição que aumenta a transcrição de um gene em resposta a um estímulo externo, facilitando a ligação da RNA polimerase ao promotor. Um indutor, um terceiro tipo de molécula reguladora, é uma molécula pequena que ativa ou reprime a transcrição ao interagir com um repressor ou um ativador.
Em procariontes, existem exemplos de operons cujos produtos genéticos são necessários de forma bastante consistente e cuja expressão, portanto, não é regulamentada. Esses operons são expressos constitutivamente, o que significa que são transcritos e traduzidos continuamente para fornecer à célula níveis intermediários constantes dos produtos proteicos. Esses genes codificam enzimas envolvidas nas funções domésticas necessárias para a manutenção celular, incluindo replicação, reparo e expressão do DNA, bem como enzimas envolvidas no metabolismo central. Em contraste, existem outros operons procarióticos que são expressos somente quando necessário e são regulados por repressores, ativadores e indutores.
Exercício\(\PageIndex{1}\)
- Quais são as partes na sequência de DNA de um operon?
- Que tipos de moléculas reguladoras existem?
Regulação pela repressão
Operons procarióticos são comumente controlados pela ligação de repressores às regiões operadoras, impedindo assim a transcrição dos genes estruturais. Esses operons são classificados como operons reprimíveis ou operons indutíveis. Operons reprimíveis, como o operon triptofano (trp), normalmente contêm genes que codificam enzimas necessárias para uma via biossintética. Enquanto o produto da via, como o triptofano, continuar sendo exigido pela célula, um operon reprimível continuará sendo expresso. No entanto, quando o produto da via biossintética começa a se acumular na célula, eliminando a necessidade de a célula continuar produzindo mais, a expressão do operon é reprimida. Por outro lado, operons induzíveis, como o operon lac de E. coli, geralmente contêm genes que codificam enzimas em uma via envolvida no metabolismo de um substrato específico, como a lactose. Essas enzimas só são necessárias quando esse substrato está disponível, portanto, a expressão dos operons é normalmente induzida apenas na presença do substrato.
A armadilha Operon: um operon reprimível
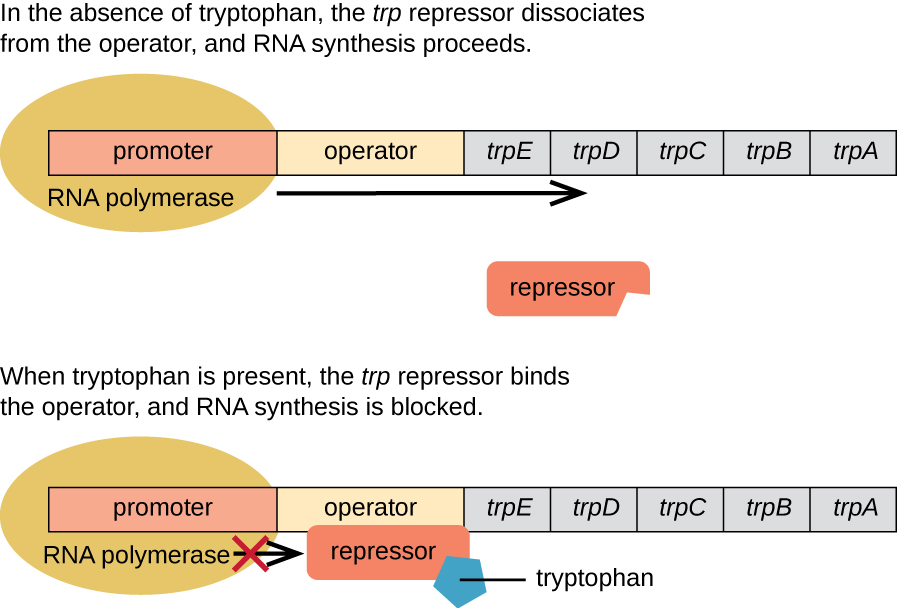
A E. coli pode sintetizar triptofano usando enzimas que são codificadas por cinco genes estruturais localizados próximos um do outro no operon trp (Figura\(\PageIndex{2}\)). Quando o triptofano ambiental está baixo, o operon é ativado. Isso significa que a transcrição é iniciada, os genes são expressos e o triptofano é sintetizado. No entanto, se o triptofano estiver presente no ambiente, o operon trp será desligado. A transcrição não ocorre e o triptofano não é sintetizado.
Quando o triptofano não está presente na célula, o repressor por si só não se liga ao operador; portanto, o operon está ativo e o triptofano é sintetizado. No entanto, quando o triptofano se acumula na célula, duas moléculas de triptofano se ligam à molécula repressora trp, que muda de forma, permitindo que ela se ligue ao operador trp. Essa ligação da forma ativa do repressor trp ao operador impede a RNA polimerase de transcrever os genes estruturais, impedindo a expressão do operon. Assim, o produto real da via biossintética controlada pelo operon regula a expressão do operon.
Assista a este vídeo para saber mais sobre o trp operon.
The lac Operon: um operon induzível
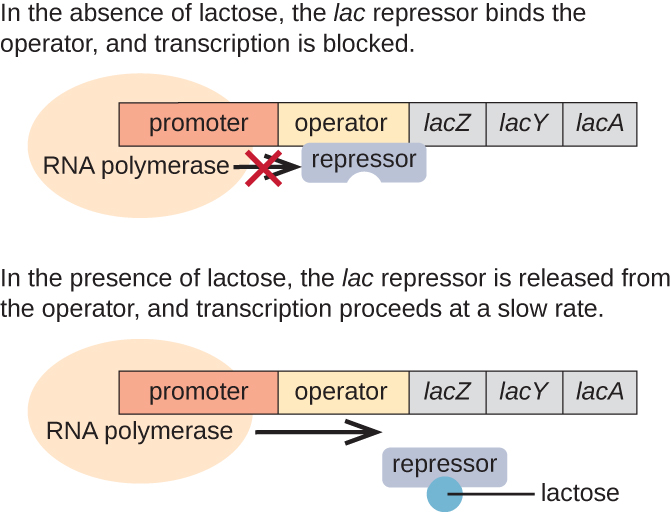
O operon lac é um exemplo de operon induzível que também está sujeito à ativação na ausência de glicose (Figura\(\PageIndex{3}\)). O operão lac codifica três genes estruturais necessários para adquirir e processar o dissacarídeo lactose do meio ambiente, decompondo-o em açúcares simples glicose e galactose. Para que o lacoperon seja expresso, a lactose deve estar presente. Isso faz sentido para a célula, pois seria um desperdício energético criar enzimas para processar a lactose se a lactose não estivesse disponível.
Na ausência de lactose, o repressor lac está ligado à região operadora do operon lac, impedindo fisicamente a RNA polimerase de transcrever os genes estruturais. No entanto, quando a lactose está presente, a lactose dentro da célula é convertida em alolactose. A alolactose serve como uma molécula indutora, ligando-se ao repressor e mudando sua forma para que não seja mais capaz de se ligar ao DNA do operador. A remoção do repressor na presença de lactose permite que a RNA polimerase se mova pela região operadora e inicie a transcrição dos genes estruturais da lactose.
The lac Operon: ativação por proteína ativadora de catabólitos
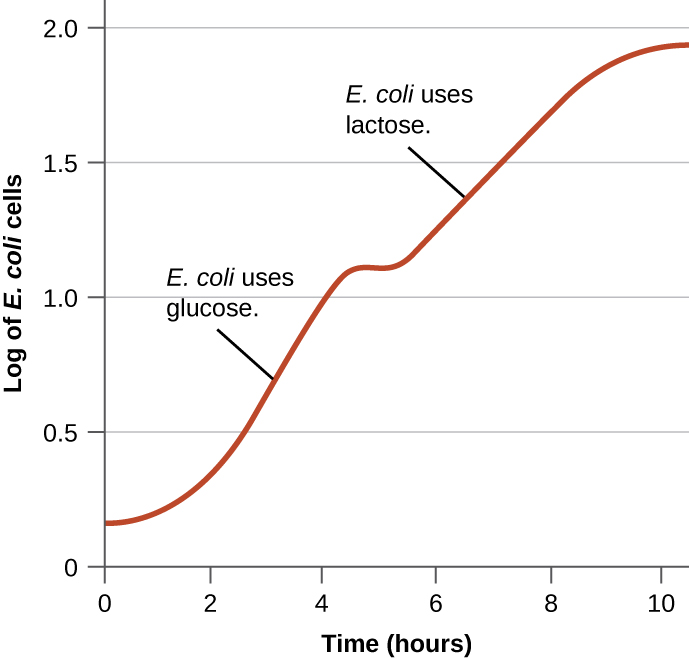
As bactérias normalmente têm a capacidade de usar uma variedade de substratos como fontes de carbono. No entanto, como a glicose geralmente é preferível a outros substratos, as bactérias têm mecanismos para garantir que substratos alternativos sejam usados apenas quando a glicose estiver esgotada. Além disso, as bactérias têm mecanismos para garantir que os genes que codificam enzimas para o uso de substratos alternativos sejam expressos somente quando o substrato alternativo estiver disponível. Na década de 1940, Jacques Monod foi o primeiro a demonstrar a preferência por certos substratos em relação a outros por meio de seus estudos sobre o crescimento da E. coli quando cultivada na presença de dois substratos diferentes simultaneamente. Esses estudos geraram curvas de crescimento diauxicas, como a mostrada na Figura\(\PageIndex{4}\). Embora o substrato preferido de glicose seja usado primeiro, a E. coli cresce rapidamente e as enzimas para o metabolismo da lactose estão ausentes. No entanto, uma vez que os níveis de glicose se esgotam, as taxas de crescimento diminuem, induzindo a expressão das enzimas necessárias para o metabolismo do segundo substrato, a lactose. Observe como a taxa de crescimento da lactose é mais lenta, conforme indicado pela menor inclinação da curva de crescimento.
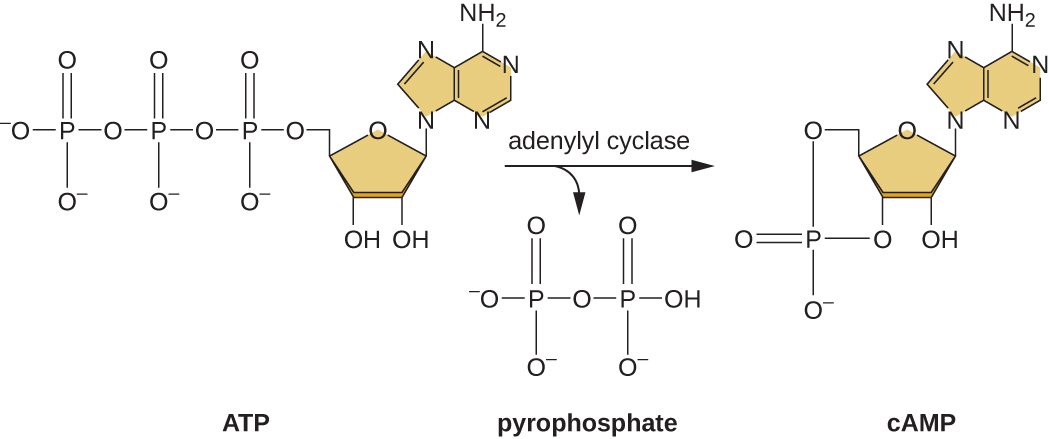
A capacidade de mudar do uso de glicose para outro substrato, como a lactose, é uma consequência da atividade de uma enzima chamada Enzima IIA (EIIA). Quando os níveis de glicose caem, as células produzem menos ATP devido ao catabolismo (veja Catabolismo de carboidratos) e o EIIA se torna fosforilado. O EIIA fosforilado ativa a adenilil ciclase, uma enzima que converte parte do ATP restante em AMP cíclico (cAMP), um derivado cíclico do AMP e importante molécula sinalizadora envolvida no metabolismo da glicose e da energia em E. coli. Como resultado, os níveis de cAMP começam a aumentar na célula (Figura\(\PageIndex{5}\)).
O operão lac também desempenha um papel nessa mudança do uso de glicose para o uso de lactose. Quando a glicose é escassa, o acúmulo de cAMP causado pelo aumento da atividade da adenilil ciclase se liga à proteína ativadora de catabólitos (CAP), também conhecida como proteína receptora cAMP (CRP). O complexo se liga à região promotora do operon lac (Figura\(\PageIndex{6}\)). Nas regiões regulatórias desses operons, um sítio de ligação do CAP está localizado a montante do sítio de ligação à RNA polimerase no promotor. A ligação do complexo Cap-camp a este local aumenta a capacidade de ligação da RNA polimerase à região promotora para iniciar a transcrição dos genes estruturais. Assim, no caso do operon lac, para que a transcrição ocorra, a lactose deve estar presente (removendo a proteína repressora de lac) e os níveis de glicose devem estar esgotados (permitindo a ligação de uma proteína ativadora). Quando os níveis de glicose estão altos, há uma repressão catabólica dos operons que codificam enzimas para o metabolismo de substratos alternativos. Devido aos baixos níveis de cAMP nessas condições, não há uma quantidade suficiente do complexo Cap-camp para ativar a transcrição desses operons. Consulte a tabela\(\PageIndex{1}\) para obter um resumo da regulação do operon lac.
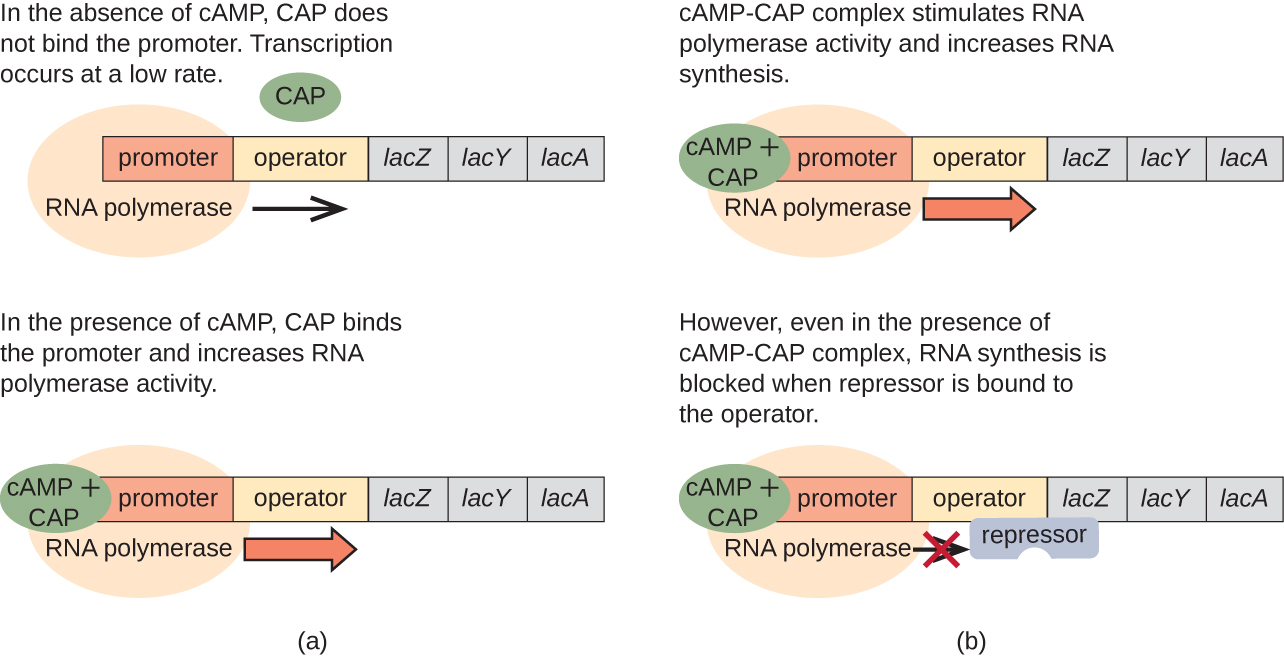
| Glicose | O CAP se liga | Lactose | Ligações repressoras | Transcrição |
|---|---|---|---|---|
| + | — | — | + | Não |
| + | — | + | — | Alguns |
| — | + | — | + | Não |
| — | + | + | — | sim |
Assista a um tutorial animado sobre o funcionamento do lac operon aqui.
Exercício\(\PageIndex{2}\)
- O que afeta a ligação do repressor do operon trp ao operador?
- Como e quando o comportamento da proteína repressora de lac é alterado?
- Além de ser reprimível, de que outra forma o operon lac é regulado?
Respostas globais de procariontes
Em procariontes, também existem vários níveis mais altos de regulação gênica que têm a capacidade de controlar a transcrição de muitos operons relacionados simultaneamente em resposta a um sinal ambiental. Um grupo de operons, todos controlados simultaneamente, é chamado de regulon.
Alarmones
Ao sentir o estresse iminente, os procariontes alteram a expressão de uma grande variedade de operons para responder em coordenação. Eles fazem isso por meio da produção de alarmonas, que são pequenos derivados de nucleotídeos intracelulares. Os alarmonas alteram quais genes são expressos e estimulam a expressão de genes específicos de resposta ao estresse. O uso de alarmones para alterar a expressão gênica em resposta ao estresse parece ser importante em bactérias patogênicas. Ao encontrar mecanismos de defesa do hospedeiro e outras condições adversas durante a infecção, muitos operons que codificam genes de virulência são regulados positivamente em resposta à sinalização de alarme. O conhecimento dessas respostas é fundamental para compreender completamente o processo de infecção de muitos patógenos e para o desenvolvimento de terapias para combater esse processo.
Fatores σ alternativos
Como a subunidade σ da RNA polimerase bacteriana confere especificidade sobre quais promotores devem ser transcritos, alterar o fator σ usado é outra forma de as bactérias alterarem rápida e globalmente quais regulons são transcritos em um determinado momento. O fator σ reconhece sequências dentro de um promotor bacteriano, então diferentes fatores σ reconhecerão sequências promotoras ligeiramente diferentes. Dessa forma, quando a célula detecta condições ambientais específicas, ela pode responder alterando o fator σ que ela expressa, degradando o antigo e produzindo um novo para transcrever os operons que codificam genes cujos produtos serão úteis sob a nova condição ambiental. Por exemplo, em bactérias esporuladoras dos gêneros Bacillus e Clostridium (que incluem muitos patógenos), um grupo de fatores σ controla a expressão dos muitos genes necessários para a esporulação em resposta a sinais estimulantes da esporulação.
Exercício\(\PageIndex{3}\)
- Qual é o nome dado a uma coleção de operons que podem ser regulados como um grupo?
- Que tipo de estímulo desencadearia a transcrição de um fator σ diferente?
Métodos adicionais de regulação em bactérias: atenuação e ribointerruptores
Embora a maior parte da expressão gênica seja regulada no nível de início da transcrição em procariontes, também existem mecanismos para controlar a conclusão da transcrição e a tradução simultaneamente. Desde sua descoberta, foi demonstrado que esses mecanismos controlam a conclusão da transcrição e tradução de muitos operons procarióticos. Como esses mecanismos vinculam diretamente a regulação da transcrição e da tradução, eles são específicos dos procariontes, porque esses processos são fisicamente separados nos eucariotos.
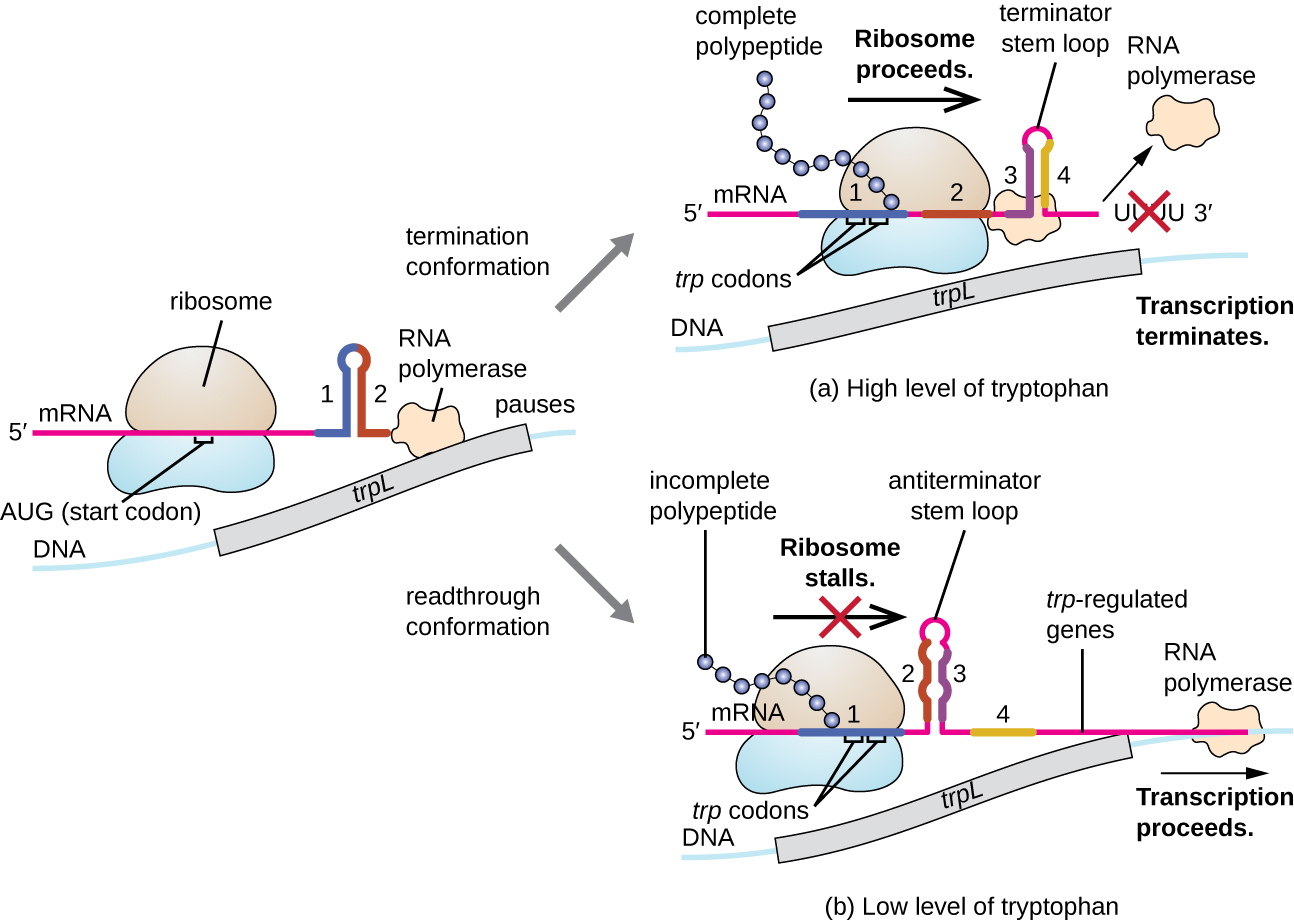
Um desses sistemas regulatórios é a atenuação, em que estruturas secundárias de alça estaminal formadas na extremidade 5' de um mRNA sendo transcrito determinam se a transcrição para completar a síntese desse mRNA ocorrerá e se esse mRNA será usado para tradução. Além do mecanismo de repressão transcricional já discutido, a atenuação também controla a expressão do operon trp em E. coli (Figura\(\PageIndex{7}\)). A região reguladora do operon trp contém uma sequência líder chamada TrPL entre o operador e o primeiro gene estrutural, que tem quatro trechos de RNA que podem se emparelhar em combinações diferentes. Quando uma alça estaminal terminadora se forma, a transcrição termina, liberando a RNA polimerase do mRNA. No entanto, quando uma alça estaminal antiterminadora se forma, isso impede a formação da alça estaminal terminadora, de modo que a RNA polimerase pode transcrever os genes estruturais.
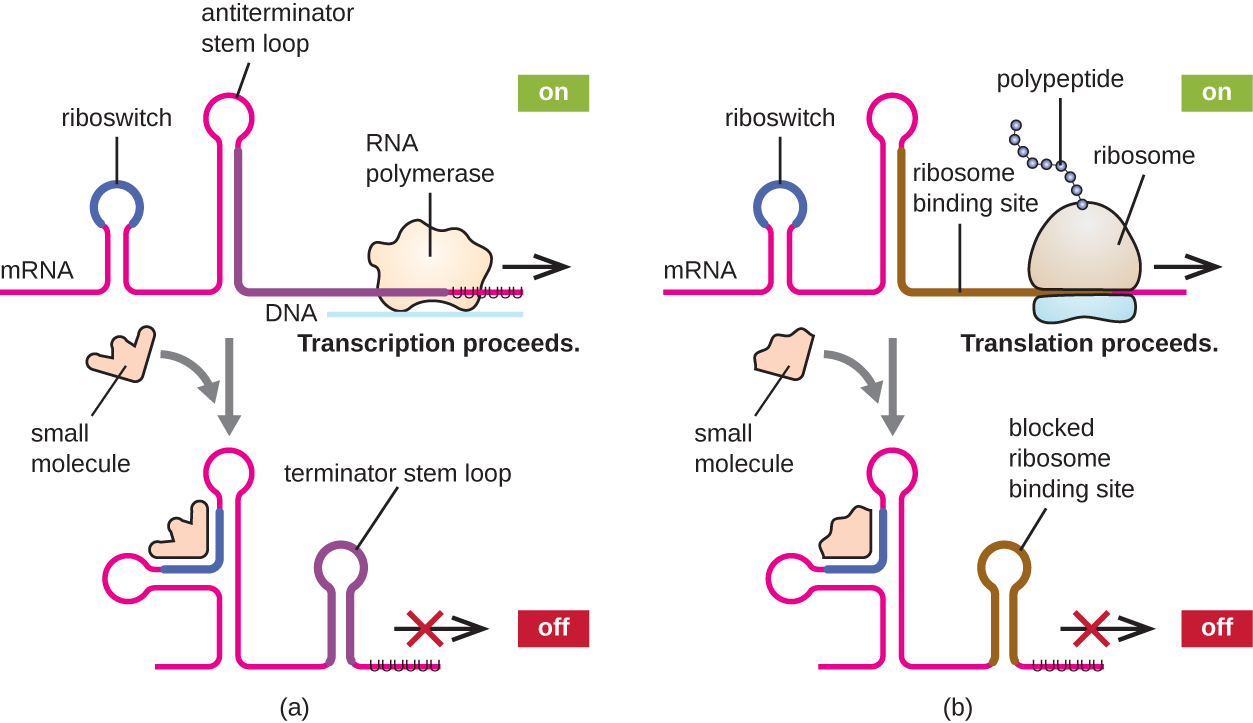
Um mecanismo relacionado de regulação simultânea da transcrição e tradução em procariontes é o uso de um riboswitch, uma pequena região de RNA não codificador encontrada na extremidade 5' de algumas moléculas de mRNA procariótico (Figura\(\PageIndex{8}\)). Um riboswitch pode se ligar a uma pequena molécula intracelular para estabilizar certas estruturas secundárias da molécula de mRNA. A ligação da pequena molécula determina qual estrutura de alça estaminal se forma, influenciando assim a conclusão da síntese de mRNA e da síntese de proteínas.
Outros fatores que afetam a expressão gênica em procariontes e eucariontes
Embora o foco em nossa discussão sobre controle transcricional tenha usado operons procarióticos como exemplos, o controle transcricional eucariótico é semelhante em muitos aspectos. Como nos procariontes, a transcrição eucariótica pode ser controlada por meio da ligação de fatores de transcrição, incluindo repressores e ativadores. Curiosamente, a transcrição eucariótica pode ser influenciada pela ligação de proteínas a regiões do DNA, chamadas potenciadoras, um pouco distantes do gene, por meio do loop de DNA facilitado entre o potenciador e o promotor (Figura\(\PageIndex{9}\)). No geral, regular a transcrição é uma forma altamente eficaz de controlar a expressão gênica em procariontes e eucariotos. No entanto, o controle da expressão gênica em eucariotos em resposta a estresses ambientais e celulares pode ser realizado de maneiras adicionais sem a ligação de fatores de transcrição às regiões regulatórias.
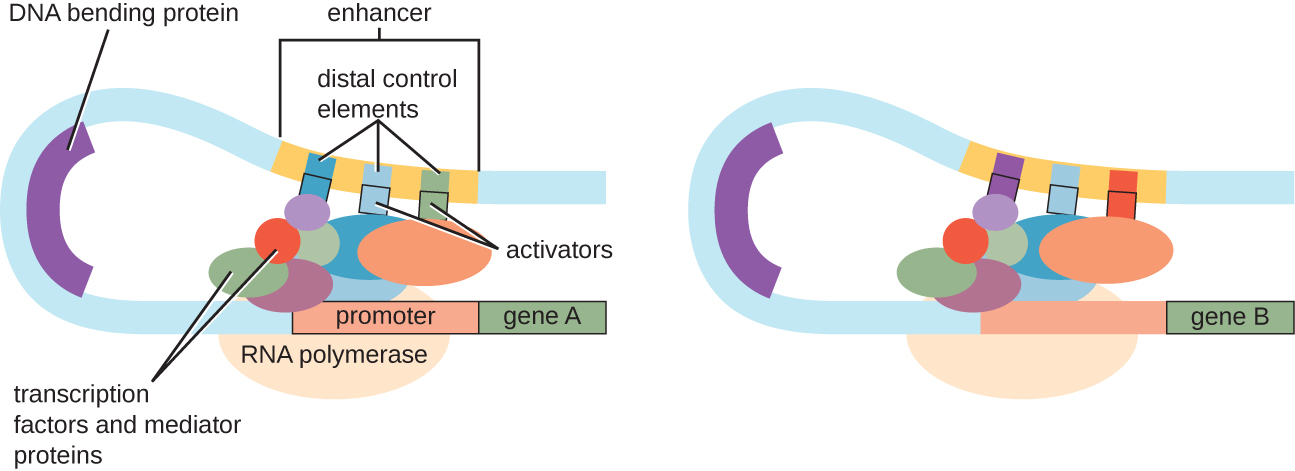
Controle de nível de DNA
Em eucariotos, as moléculas de DNA ou histonas associadas podem ser modificadas quimicamente de forma a influenciar a transcrição; isso é chamado de regulação epigenética. Foi demonstrado que a metilação de certos nucleotídeos de citosina no DNA em resposta a fatores ambientais influencia o uso desse DNA para transcrição, com a metilação do DNA comumente se correlacionando com níveis reduzidos de expressão gênica. Além disso, em resposta a fatores ambientais, as proteínas histônicas para empacotar o DNA também podem ser modificadas quimicamente de várias maneiras, incluindo acetilação e desacetilação, influenciando o estado de embalagem do DNA e, assim, afetando a disponibilidade de DNA solto para transcrição. Essas modificações químicas às vezes podem ser mantidas por meio de várias rodadas de divisão celular, tornando pelo menos algumas dessas mudanças epigenéticas hereditárias.
Este vídeo descreve como a regulação epigenética controla a expressão gênica.
Exercício\(\PageIndex{}\)
- O que interrompe ou permite que a transcrição continue quando a atenuação está em operação?
- O que determina o estado de um riboswitch?
- Descreva a função de um potenciador.
- Descreva dois mecanismos de regulação epigenética em eucariotos.
Foco clínico: Resolução
Embora Mark tenha sobrevivido à sua luta com fascite necrosante, ele agora teria que passar por uma cirurgia de enxerto de pele, seguida de fisioterapia de longo prazo. Com base na quantidade de massa muscular que ele perdeu, é improvável que sua perna volte a ter força total, mas seu fisioterapeuta está otimista de que ele recuperará o uso da perna.
Testes laboratoriais revelaram que o agente causador da infecção de Mark era uma cepa do estreptococo do grupo A (estreptococo do grupo A). Conforme exigido por lei, o caso de Mark foi denunciado ao departamento estadual de saúde e, finalmente, aos Centros de Controle e Prevenção de Doenças (CDC). No CDC, a cepa do estreptococo do grupo A isolada de Mark foi analisada mais detalhadamente quanto à resistência à meticilina.
A resistência à meticilina é geneticamente codificada e está se tornando mais comum no estreptococo do grupo A por meio da transferência horizontal de genes. Na fasciite necrosante, o fluxo sanguíneo para a área infectada é normalmente limitado devido à ação de várias toxinas bacterianas geneticamente codificadas. É por isso que normalmente há pouco ou nenhum sangramento como resultado do teste de incisão. Infelizmente, essas toxinas bacterianas limitam a eficácia dos antibióticos intravenosos na eliminação da infecção da pele e do tecido subjacente, o que significa que a resistência aos antibióticos por si só não explica a ineficácia do tratamento de Mark. No entanto, a antibioticoterapia intravenosa foi necessária para ajudar a minimizar o possível desfecho da sepse, que é um resultado comum da fasciite necrosante. Por meio da análise genômica pelo CDC da cepa isolada de Mark, foi demonstrado que vários dos importantes genes de virulência estão codificados em profagos, indicando que a transdução é importante na transferência horizontal desses genes de uma célula bacteriana para outra.
Conceitos principais e resumo
- A expressão gênica é um processo rigidamente regulado.
- A expressão gênica em procariontes é amplamente regulada no ponto de transcrição. A expressão gênica em eucariotos também é regulada pós-transcricionalmente.
- Genes estruturais procarióticos de função relacionada são frequentemente organizados em operons, todos controlados pela transcrição de um único promotor. A região regulatória de um operon inclui o próprio promotor e a região ao redor do promotor, à qual os fatores de transcrição podem se vincular para influenciar a transcrição.
- Embora alguns operons sejam expressos constitutivamente, a maioria está sujeita à regulação por meio do uso de fatores de transcrição (repressores e ativadores). Um repressor se liga a um operador, uma sequência de DNA dentro da região regulatória entre o sítio de ligação da RNA polimerase no promotor e o primeiro gene estrutural, bloqueando assim fisicamente a transcrição desses operons. Um ativador se liga dentro da região regulatória de um operon, ajudando a RNA polimerase a se ligar ao promotor, melhorando assim a transcrição desse operon. Um indutor influencia a transcrição ao interagir com um repressor ou ativador.
- O operon trp é um exemplo clássico de operon reprimível. Quando o triptofano se acumula, o triptofano se liga a um repressor, que então se liga ao operador, impedindo a posterior transcrição.
- O operon lac é um exemplo clássico de operon induzível. Quando a lactose está presente na célula, ela é convertida em alolactose. A alolactose atua como um indutor, ligando-se ao repressor e impedindo que o repressor se ligue ao operador. Isso permite a transcrição dos genes estruturais.
- O operon lac também está sujeito à ativação. Quando os níveis de glicose estão esgotados, algum ATP celular é convertido em cAMP, que se liga à proteína ativadora de catabólitos (CAP). O complexo Camp-cap ativa a transcrição do operon lac. Quando os níveis de glicose estão altos, sua presença impede a transcrição do operon lac e de outros operons pela repressão catabólica.
- Pequenas moléculas intracelulares chamadas alarmonas são produzidas em resposta a vários estresses ambientais, permitindo que as bactérias controlem a transcrição de um grupo de operons, chamado regulon.
- As bactérias têm a capacidade de alterar qual fator σ da RNA polimerase elas usam em resposta às condições ambientais para alterar rápida e globalmente quais regulons são transcritos.
- Os procariontes têm mecanismos regulatórios, incluindo atenuação e uso de ribointerruptores, para controlar simultaneamente a conclusão da transcrição e tradução dessa transcrição. Esses mecanismos funcionam por meio da formação de alças estaminais na extremidade 5' de uma molécula de mRNA atualmente sendo sintetizada.
- Existem pontos adicionais de regulação da expressão gênica em procariontes e eucariotos. Em eucariotos, a regulação epigenética por modificação química do DNA ou histonas e a regulação do processamento de RNA são dois métodos.


