7.5: Usando a bioquímica para identificar microrganismos
- Page ID
- 181605
Objetivos de
- Descreva exemplos de produtos de biossíntese dentro de uma célula que podem ser detectados para identificar bactérias
A identificação precisa de isolados bacterianos é essencial em um laboratório de microbiologia clínica porque os resultados geralmente informam as decisões sobre o tratamento que afetam diretamente os resultados dos pacientes. Por exemplo, casos de intoxicação alimentar exigem uma identificação precisa do agente causador para que os médicos possam prescrever o tratamento adequado. Da mesma forma, é importante identificar com precisão o patógeno causador durante um surto da doença, para que estratégias apropriadas possam ser empregadas para conter a epidemia.
Há muitas maneiras de detectar, caracterizar e identificar microrganismos. Alguns métodos dependem de características bioquímicas fenotípicas, enquanto outros usam identificação genotípica. As características bioquímicas de uma bactéria fornecem muitas características que são úteis para classificação e identificação. Analisar as capacidades nutricionais e metabólicas do isolado bacteriano é uma abordagem comum para determinar o gênero e a espécie da bactéria. Algumas das vias metabólicas mais importantes que as bactérias usam para sobreviver serão discutidas em Metabolismo microbiano. Nesta seção, discutiremos alguns métodos que usam características bioquímicas para identificar microrganismos.
Alguns microrganismos armazenam certos compostos como grânulos em seu citoplasma, e o conteúdo desses grânulos pode ser usado para fins de identificação. Por exemplo, o poli-β-hidroxibutirato (PHB) é um composto de armazenamento de carbono e energia encontrado em algumas bactérias não fluorescentes do gênero Pseudomonas. Diferentes espécies desse gênero podem ser classificadas pela presença ou ausência de PHB e pigmentos fluorescentes. O patógeno humano P. aeruginosa e o patógeno vegetal P. syringae são dois exemplos de espécies fluorescentes de Pseudomonas que não acumulam grânulos de PHB.
Outros sistemas dependem de características bioquímicas para identificar microrganismos por meio de suas reações bioquímicas, como a utilização de carbono e outros testes metabólicos. Em pequenos laboratórios ou em laboratórios de ensino, esses ensaios são realizados usando um número limitado de tubos de ensaio. No entanto, sistemas mais modernos, como o desenvolvido pela Biolog, Inc., são baseados em painéis de reações bioquímicas realizadas simultaneamente e analisadas por software. O sistema da Biolog identifica as células com base em sua capacidade de metabolizar certos produtos bioquímicos e em suas propriedades fisiológicas, incluindo pH e sensibilidade química. Ele usa todas as principais classes de bioquímicos em sua análise. As identificações podem ser realizadas manualmente ou com os instrumentos semi ou totalmente automatizados.
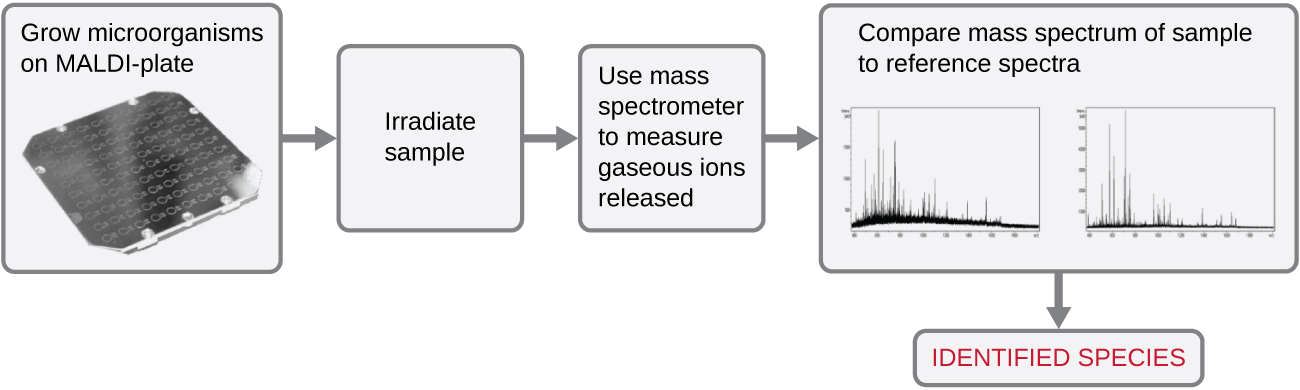
Outro sistema automatizado identifica microrganismos determinando o espectro de massa da amostra e, em seguida, comparando-a com um banco de dados que contém espectros de massa conhecidos para milhares de microrganismos. Esse método é baseado na espectrometria de massa de dessorção/ionização com tempo de voo assistida por matriz (MALDI-TOF) e usa placas MALDI descartáveis nas quais o microrganismo é misturado com um reagente de matriz especializado (Figura\(\PageIndex{1}\)). A mistura amostra/reagente é irradiada com um laser ultravioleta pulsado de alta intensidade, resultando na ejeção de íons gasosos gerados a partir dos vários constituintes químicos do microrganismo. Esses íons gasosos são coletados e acelerados por meio do espectrômetro de massa, com íons viajando a uma velocidade determinada por sua relação massa-carga (m/z), atingindo assim o detector em momentos diferentes. Um gráfico do sinal do detector versus m/z produz um espectro de massa para o organismo que está exclusivamente relacionado à sua composição bioquímica. A comparação do espectro de massa com uma biblioteca de espectros de referência obtidos a partir de análises idênticas de microrganismos conhecidos permite a identificação do micróbio desconhecido.
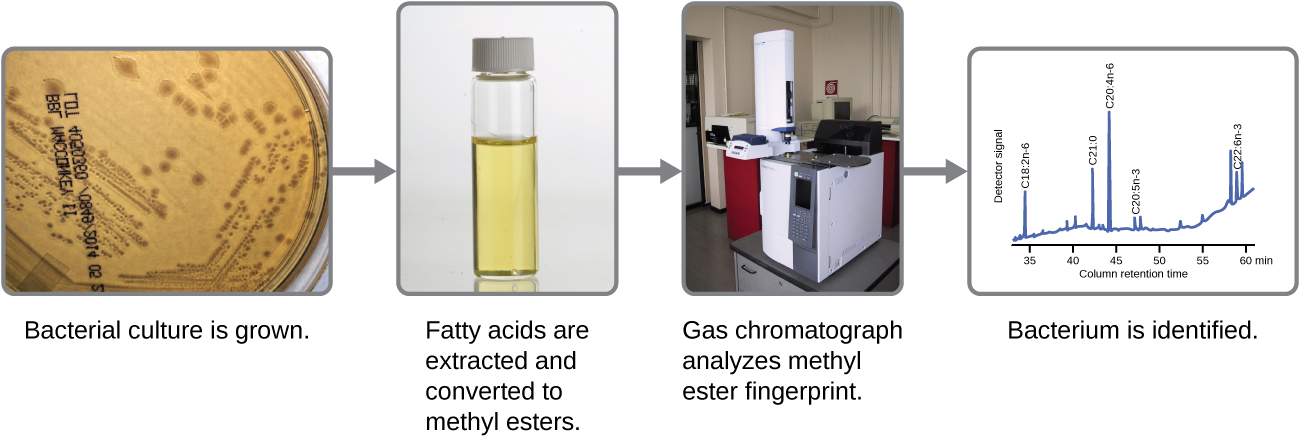
Os micróbios também podem ser identificados medindo seus perfis lipídicos exclusivos. Como aprendemos, os ácidos graxos dos lipídios podem variar no comprimento da cadeia, na presença ou ausência de ligações duplas e no número de ligações duplas, grupos hidroxila, ramos e anéis. Para identificar um micróbio por sua composição lipídica, os ácidos graxos presentes em suas membranas são analisados. Uma análise bioquímica comum usada para esse fim é uma técnica usada em laboratórios clínicos, de saúde pública e de alimentos. Ele se baseia na detecção de diferenças únicas em ácidos graxos e é chamado de análise de éster metílico de ácidos graxos (FAME). Em uma análise de FAME, os ácidos graxos são extraídos das membranas dos microrganismos, alterados quimicamente para formar ésteres metílicos voláteis e analisados por cromatografia gasosa (GC). O cromatograma GC resultante é comparado com os cromatogramas de referência em um banco de dados contendo dados de milhares de isolados bacterianos para identificar o microrganismo desconhecido (Figura\(\PageIndex{2}\)).
Um método relacionado para identificação de microrganismos é chamado de análise de ácidos graxos derivados de fosfolipídios (PLFA). As membranas são compostas principalmente de fosfolipídios, que podem ser saponificados (hidrolisados com álcali) para liberar os ácidos graxos. A mistura de ácidos graxos resultante é então submetida à análise de FAME, e os perfis lipídicos medidos podem ser comparados com os de microrganismos conhecidos para identificar o microrganismo desconhecido.
A identificação bacteriana também pode ser baseada nas proteínas produzidas sob condições específicas de crescimento no corpo humano. Esses tipos de procedimentos de identificação são chamados de análise proteômica. Para realizar a análise proteômica, as proteínas do patógeno são primeiro separadas por cromatografia líquida de alta pressão (HPLC) e as frações coletadas são então digeridas para produzir fragmentos peptídicos menores. Esses peptídeos são identificados por espectrometria de massa e comparados com os de microrganismos conhecidos para identificar o microrganismo desconhecido na amostra original.
Os microrganismos também podem ser identificados pelos carboidratos ligados às proteínas (glicoproteínas) na membrana plasmática ou na parede celular. Anticorpos e outras proteínas ligadoras de carboidratos podem se ligar a carboidratos específicos na superfície celular, fazendo com que as células se agrupem. Os testes sorológicos (por exemplo, os testes dos grupos de Lancefield, que são usados para identificação de espécies de Streptococcus) são realizados para detectar os carboidratos exclusivos localizados na superfície da célula.
Foco clínico: Resolução
Penny parou de usar seu novo protetor solar e aplicou o creme de corticosteróide na erupção cutânea conforme as instruções. No entanto, depois de vários dias, sua erupção cutânea não havia melhorado e, na verdade, parecia estar piorando. Ela marcou uma consulta de acompanhamento com seu médico, que observou uma erupção cutânea vermelha irregular e bolhas cheias de pus ao redor dos folículos pilosos (Figura\(\PageIndex{3}\)). A erupção cutânea estava especialmente concentrada em áreas que teriam sido cobertas por um traje de banho. Depois de alguns questionamentos, Penny disse ao médico que recentemente participou de uma festa na piscina e passou algum tempo em uma banheira de hidromassagem. Diante dessas novas informações, o médico suspeitou de um caso de erupção cutânea na banheira de hidromassagem, uma infecção frequentemente causada pela bactéria Pseudomonas aeruginosa, um patógeno oportunista que pode se desenvolver em banheiras de hidromassagem e piscinas, especialmente quando a água não está suficientemente clorada. P. aeruginosa é a mesma bactéria associada a infecções nos pulmões de pacientes com fibrose cística.
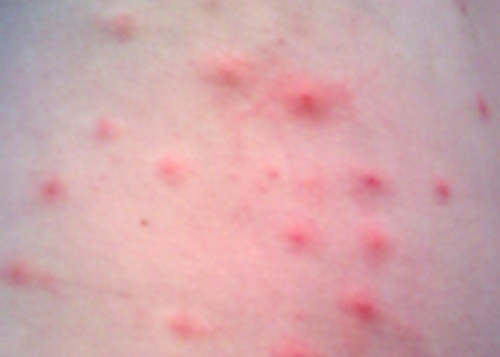
O médico coletou uma amostra da erupção cutânea de Penny para ser enviada ao laboratório de microbiologia clínica. Testes confirmatórios foram realizados para distinguir P. aeruginosa de patógenos entéricos que também podem estar presentes na água da piscina e da banheira de hidromassagem. O teste incluiu a produção do pigmento azul esverdeado piocianina em ágar cetrimida e o crescimento a 42 °C. A cetrimida é um agente seletivo que inibe o crescimento de outras espécies da flora microbiana e também aumenta a produção de pigmentos de P. aeruginosa piocianina e fluoresceína, que são um azul esverdeado característico e amarelo-verde, respectivamente.
Os testes confirmaram a presença de P. aeruginosa na amostra de pele de Penny, mas o médico decidiu não prescrever um antibiótico. Embora a P. aeruginosa seja uma bactéria, as espécies de Pseudomonas geralmente são resistentes a muitos antibióticos. Felizmente, infecções de pele como a de Penny geralmente são autolimitadas; a erupção cutânea geralmente dura cerca de 2 semanas e se resolve sozinha, com ou sem tratamento médico. O médico aconselhou Penny a esperar e continuar usando o creme de corticosteróides. O creme não mata a P. aeruginosa na pele de Penny, mas deve acalmar a erupção cutânea e minimizar a coceira, suprimindo a resposta inflamatória do corpo à bactéria.
Conceitos principais e resumo
- A identificação precisa de bactérias é essencial em um laboratório clínico para diagnóstico e gerenciamento de epidemias, pandemias e intoxicações alimentares causadas por surtos bacterianos.
- A identificação fenotípica de microrganismos envolve o uso de características observáveis, incluindo perfis de componentes estruturais, como lipídios, produtos biossintéticos, como açúcares ou aminoácidos, ou compostos de armazenamento, como poli-β-hidroxibutirato.
- Um micróbio desconhecido pode ser identificado a partir do espectro de massa exclusivo produzido quando é analisado pelo tempo de dessorção/ionização a laser assistido por matriz da espectrometria de massa de voo (MALDI-TOF).
- Os micróbios podem ser identificados determinando suas composições lipídicas, usando a análise de ésteres metílicos de ácidos graxos (FAME) ou ácidos graxos derivados de fosfolipídios (PLFA).
- A análise proteômica, o estudo de todas as proteínas acumuladas de um organismo, também pode ser usada para identificação bacteriana.
- As glicoproteínas na membrana plasmática ou nas estruturas da parede celular podem se ligar a lectinas ou anticorpos e podem ser usadas para identificação.


