14.5 : Tester l'efficacité des antimicrobiens
- Page ID
- 187936
Objectifs d'apprentissage
- Expliquer le concept de résistance aux médicaments
- Décrire comment les micro-organismes développent ou acquièrent une résistance aux médicaments
- Décrire les différents mécanismes de résistance aux médicaments antimicrobiens
La résistance aux antimicrobiens n'est pas un phénomène nouveau. Dans la nature, les microbes évoluent constamment afin de vaincre les composés antimicrobiens produits par d'autres microorganismes. Le développement humain de médicaments antimicrobiens et leur utilisation clinique généralisée n'ont fait qu'exercer une pression sélective supplémentaire qui favorise une évolution ultérieure. Plusieurs facteurs importants peuvent accélérer l'évolution de la résistance aux médicaments. Il s'agit notamment de la surutilisation et de la mauvaise utilisation d'antimicrobiens, de l'utilisation inappropriée d'antimicrobiens, de la posologie sous-thérapeutique et du non-respect par les patients du traitement recommandé.
L'exposition d'un agent pathogène à un composé antimicrobien permet de sélectionner des mutations chromosomiques conférant une résistance, qui peuvent être transférées verticalement aux générations microbiennes suivantes et finalement devenir prédominantes dans une population microbienne exposée de façon répétée à l'antimicrobien. Par ailleurs, de nombreux gènes responsables de la résistance aux médicaments se trouvent sur des plasmides ou dans des transposons qui peuvent être facilement transférés entre microbes par transfert horizontal de gènes (voir Comment les procaryotes asexués atteignent la diversité génétique). Les transposons ont également la capacité de déplacer les gènes de résistance entre les plasmides et les chromosomes afin de favoriser davantage la propagation de la résistance.
Mécanismes de résistance aux médicaments
Il existe plusieurs mécanismes courants de résistance aux médicaments, qui sont résumés dans la Figure\(\PageIndex{1}\). Ces mécanismes incluent la modification enzymatique du médicament, la modification de la cible antimicrobienne et la prévention de la pénétration ou de l'accumulation du médicament.
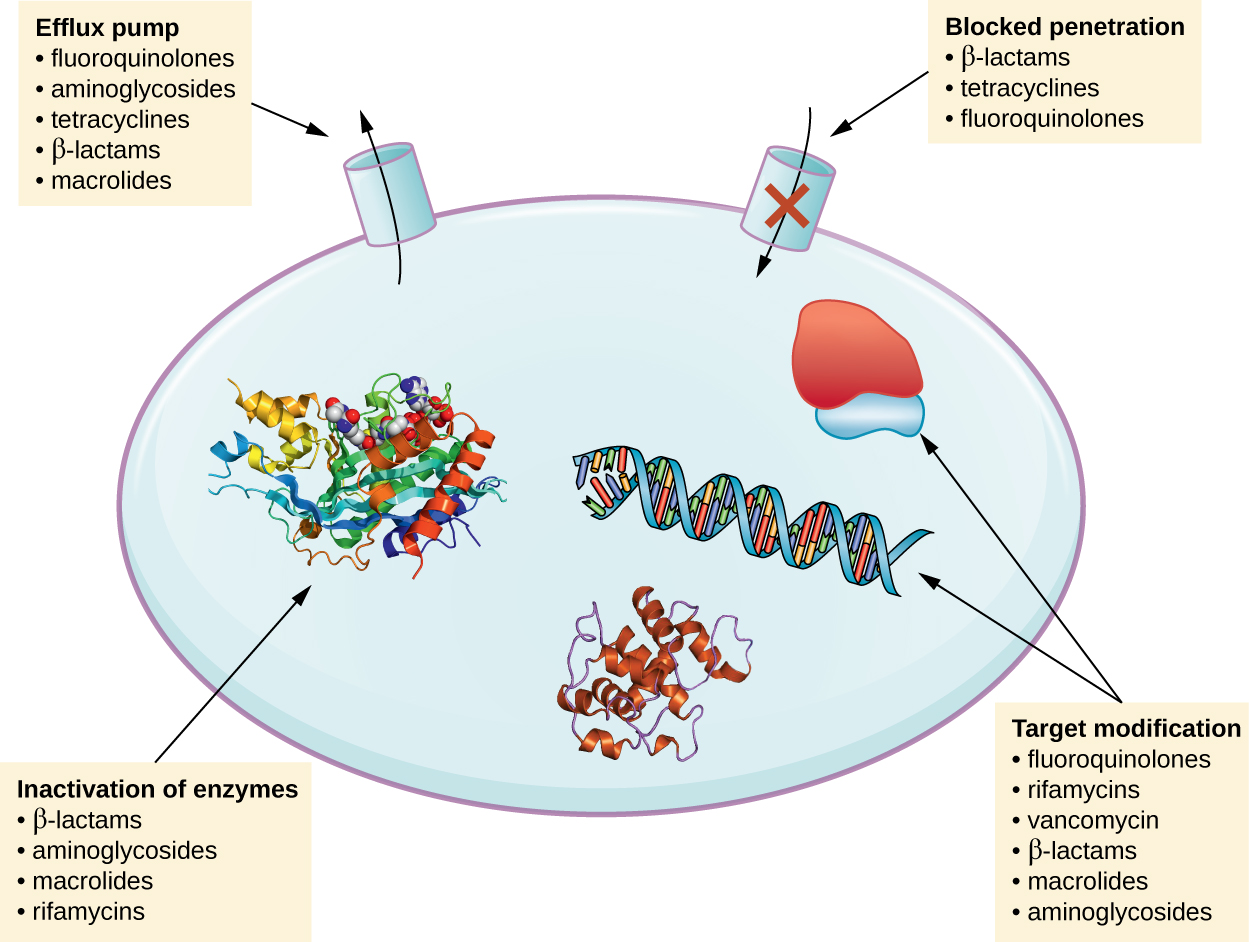
Modification ou inactivation d'un médicament
Les gènes de résistance peuvent coder pour des enzymes qui modifient chimiquement un antimicrobien, l'inactivant ainsi, ou détruisent un antimicrobien par hydrolyse. La résistance à de nombreux types d'antimicrobiens se produit grâce à ce mécanisme. Par exemple, la résistance aux aminoglycosides peut se produire par transfert enzymatique de groupes chimiques vers la molécule médicamenteuse, altérant ainsi la liaison du médicament à sa cible bactérienne. Pour les β-lactamines, la résistance bactérienne peut impliquer l'hydrolyse enzymatique de la liaison β-lactame au sein du cycle β-lactame de la molécule médicamenteuse. Une fois la liaison β-lactame rompue, le médicament perd son activité antibactérienne. Ce mécanisme de résistance est médié par les β-lactamases, qui sont le mécanisme le plus courant de résistance aux β-lactamines. L'inactivation de la rifampicine se produit généralement par glycosylation, phosphorylation ou ribosylation de l'adénosine diphosphate (ADP), et une résistance aux macrolides et aux lincosamides peut également survenir en raison de l'inactivation enzymatique du médicament ou de sa modification.
Prévention de l'absorption ou de l'efflux cellulaire
Les microbes peuvent développer des mécanismes de résistance qui consistent à inhiber l'accumulation d'un médicament antimicrobien, ce qui empêche le médicament d'atteindre sa cible cellulaire. Cette stratégie est courante chez les agents pathogènes à Gram négatif et peut impliquer des modifications de la composition lipidique de la membrane externe, de la sélectivité des canaux poriques et/ou de la concentration des canaux poriques. Par exemple, un mécanisme courant de résistance aux carbapénèmes chez Pseudomonas aeruginosa consiste à diminuer la quantité de sa porine OprD, qui est la principale porte d'entrée des carbapénèmes à travers la membrane externe de ce pathogène. De plus, de nombreuses bactéries pathogènes gram-positives et gram-négatives produisent des pompes d'efflux qui transportent activement un médicament antimicrobien hors de la cellule et empêchent l'accumulation du médicament à un niveau qui serait antibactérien. Par exemple, la résistance aux β-lactamines, aux tétracyclines et aux fluoroquinolones se produit généralement par efflux actif hors de la cellule, et il est assez fréquent qu'une seule pompe d'efflux ait la capacité de transférer plusieurs types d'antimicrobiens.
Modification de la cible
Étant donné que les médicaments antimicrobiens ont des cibles très spécifiques, les modifications structurelles apportées à ces cibles peuvent empêcher la fixation du médicament, ce qui rend le médicament inefficace. Grâce à des mutations spontanées dans les gènes codant pour les cibles des médicaments antibactériens, les bactéries ont un avantage évolutif qui leur permet de développer une résistance aux médicaments. Ce mécanisme de développement de la résistance est assez courant. Les modifications génétiques ayant un impact sur le site actif des protéines liant les pénicillines (PBP) peuvent inhiber la liaison des bêta-lactamines et créer une résistance à de nombreux médicaments de cette classe. Ce mécanisme est très fréquent chez les souches de Streptococcus pneumoniae, qui modifient leurs propres PBP par des mécanismes génétiques. En revanche, les souches de Staphylococcus aureus développent une résistance à la méthicilline (SARM) par l'acquisition d'un nouveau PBP de faible affinité, plutôt que de modifier structurellement leurs PBP existants. Non seulement ce nouveau PBP de faible affinité confère une résistance à la méthicilline, mais il résiste également à pratiquement tous les médicaments à base de β-lactamines, à l'exception des nouvelles céphalosporines de cinquième génération conçues spécifiquement pour tuer le SARM. D'autres exemples de cette stratégie de résistance incluent les modifications apportées à
- sous-unités de ribosomes, conférant une résistance aux macrolides, aux tétracyclines et aux aminoglycosides ;
- structure des lipopolysaccharides (LPS), offrant une résistance aux polymyxines ;
- ARN polymérase, conférant une résistance à la rifampicine ;
- ADN gyrase, résistant aux fluoroquinolones ;
- des enzymes métaboliques, conférant une résistance aux sulfamides, aux sulfones et au triméthoprime ; et
- chaînes peptidiques sous-unitaires du peptidoglycane, conférant une résistance aux glycopeptides.
Surproduction cible ou dérivation enzymatique
Lorsqu'un médicament antimicrobien agit comme un antimétabolite, ciblant une enzyme spécifique pour inhiber son activité, il existe d'autres moyens de développer une résistance microbienne. Tout d'abord, le microbe peut surproduire l'enzyme cible de telle sorte qu'il existe une quantité suffisante d'enzyme exempte d'antimicrobiens pour effectuer la réaction enzymatique appropriée. Ensuite, la cellule bactérienne peut développer un pontage qui évite d'avoir recours à l'enzyme cible fonctionnelle. Ces deux stratégies se sont révélées être des mécanismes de résistance aux sulfonamides. Il a été démontré que la résistance à la vancomycine chez S. aureus implique la diminution de la réticulation des chaînes peptidiques dans la paroi cellulaire bactérienne, ce qui augmente les cibles auxquelles la vancomycine peut se lier dans la paroi cellulaire externe. La fixation accrue de la vancomycine dans la paroi cellulaire externe crée un blocage qui empêche les molécules médicamenteuses libres de pénétrer et de bloquer la synthèse de nouvelles parois cellulaires.
Mimétisme cible
Un mécanisme de résistance récemment découvert, appelé mimétisme de la cible, implique la production de protéines qui lient et séquestrent les médicaments, empêchant ainsi les médicaments de se lier à leur cible. Par exemple, Mycobacterium tuberculosis produit une protéine avec des répétitions régulières de pentapeptides qui semble imiter la structure de l'ADN. Cette protéine lie les fluoroquinolones, les séquestrant et les empêchant de se lier à l'ADN, conférant ainsi à M. tuberculosis une résistance aux fluoroquinolones. Il a été démontré que les protéines qui imitent le site A du ribosome bactérien contribuent également à la résistance aux aminoglycosides. 1
Exercice\(\PageIndex{1}\)
Énumérez plusieurs mécanismes de résistance aux médicaments.
Microbes multirésistants et résistance croisée
D'un point de vue clinique, nos principales préoccupations concernent les microbes multirésistants (MDR) et la résistance croisée. Les MDR sont communément appelés « superbactéries » et sont porteurs d'un ou de plusieurs mécanismes de résistance, ce qui les rend résistants à de multiples antimicrobiens. Dans le cas de la résistance croisée, un mécanisme de résistance unique confère une résistance à de multiples médicaments antimicrobiens. Par exemple, le fait de disposer d'une pompe d'efflux capable d'exporter plusieurs médicaments antimicrobiens est un moyen courant pour les microbes de résister à plusieurs médicaments en utilisant un seul mécanisme de résistance. Ces dernières années, plusieurs superbactéries importantes sur le plan clinique sont apparues, et les CDC indiquent que les superbactéries sont responsables de plus de 2 millions d'infections aux États-Unis chaque année, entraînant au moins 23 000 décès. 2 Plusieurs des superbactéries abordées dans les sections suivantes ont été surnommées les agents pathogènes ESKAPE. Cet acronyme fait référence aux noms des agents pathogènes (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa et Enterobacter spp.) mais il convient également du fait que ceux-ci les agents pathogènes peuvent « échapper » à de nombreuses formes classiques de traitement antimicrobien. Les infections par les agents pathogènes ESKAPE peuvent donc être difficiles à traiter et sont à l'origine d'un grand nombre d'infections nosocomiales.
Staphylococcus aureus résistant à la méthicilline (SARM)
La méthicilline, une pénicilline semi-synthétique, a été conçue pour résister à l'inactivation par les β-lactamases. Malheureusement, peu de temps après l'introduction de la méthicilline dans la pratique clinique, des souches de S. aureus résistantes à la méthicilline sont apparues et ont commencé à se propager. Le mécanisme de résistance, à savoir l'acquisition d'un nouveau PBP de faible affinité, a conféré à S. aureus une résistance à tous les β-lactamines disponibles. Les souches de S. aureus résistantes à la méthicilline (SARM) sont des agents pathogènes opportunistes très répandus qui sont particulièrement préoccupants pour les infections de la peau et d'autres plaies, mais elles peuvent également provoquer des pneumonies et des septicémies. Bien qu'elles aient été initialement un problème dans les établissements de santé (SARM d'origine hospitalière [SARM HA]), les infections à SARM sont désormais également contractées par contact avec des membres contaminés du grand public, appelés SARM associés à la communauté (SARM CA). Environ un tiers de la population est porteuse de S. aureus en tant que membre de leur microbiote nasal normal sans maladie, et environ 6 % de ces souches sont résistantes à la méthicilline. 3 4
Acide clavulanique : le petit assistant de la pénicilline
Avec l'introduction de la pénicilline au début des années 1940 et sa production de masse ultérieure, la société a commencé à considérer les antibiotiques comme des remèdes miracles pour un large éventail de maladies infectieuses. Malheureusement, dès 1945, la résistance à la pénicilline a été documentée pour la première fois et a commencé à se propager. Plus de 90 % des isolats cliniques actuels de S. aureus sont résistants à la pénicilline. 5
Bien que la mise au point de nouveaux médicaments antimicrobiens soit une solution à ce problème, les scientifiques ont exploré de nouvelles approches, notamment la mise au point de composés qui inactivent les mécanismes de résistance. Le développement de l'acide clavulanique constitue l'un des premiers exemples de cette stratégie. L'acide clavulanique est une molécule produite par la bactérie Streptococcus clavuligerus. Il contient un cycle β-lactame, ce qui le rend structurellement similaire à la pénicilline et aux autres β-lactamines, mais ne montre aucune efficacité clinique lorsqu'il est administré seul. Au lieu de cela, l'acide clavulanique se lie de manière irréversible au site actif des β-lactamases et les empêche d'inactiver une pénicilline administrée simultanément.
L'acide clavulanique a été développé pour la première fois dans les années 1970 et a été commercialisé en masse en association avec l'amoxicilline à partir des années 1980 sous le nom de marque Augmentin. Comme c'est généralement le cas, une résistance à l'association amoxicilline-acide clavulanique est rapidement apparue. La résistance résulte le plus souvent du fait que les bactéries augmentent la production de leur β-lactamase et suppriment les effets inhibiteurs de l'acide clavulanique, mutent leur β-lactamase de manière à ce qu'elle ne soit plus inhibée par l'acide clavulanique, ou qu'elles acquièrent une nouvelle β-lactamase qui n'est pas inhibée par l'acide clavulanique. Malgré les préoccupations croissantes liées à la résistance, l'acide clavulanique et les inhibiteurs connexes de la β-lactamase (sulbactam et tazobactam) constituent une nouvelle stratégie importante : le développement de composés qui inhibent directement les enzymes conférant une résistance aux antimicrobiens.
Entérocoques et Staphylococcus aureus résistants à la vancomycine
La vancomycine n'est efficace que contre les organismes à Gram positif et elle est utilisée pour traiter les infections des plaies, les infections septiques, les endocardites et les méningites causées par des agents pathogènes résistants à d'autres antibiotiques. Il est considéré comme l'une des dernières lignes de défense contre ces infections résistantes, y compris le SARM. Avec l'augmentation de la résistance aux antibiotiques dans les années 1970 et 1980, l'utilisation de la vancomycine a augmenté, et il n'est pas surprenant que nous ayons assisté à l'émergence et à la propagation d'entérocoques résistants à la vancomycine (ERV), de S. aureus résistant à la vancomycine (VRSA) et de S. aureus intermédiaire à la vancomycine (VISA). Le mécanisme de résistance à la vancomycine chez les entérocoques est la modification de la cible impliquant un changement structurel du composant peptidoglycane des sous-unités du peptidoglycane, empêchant ainsi la vancomycine de se lier. Ces souches se propagent généralement parmi les patients en milieu clinique par contact avec des agents de santé et des surfaces et équipements médicaux contaminés.
Les souches VISA et VRSA diffèrent l'une de l'autre par le mécanisme de résistance et le degré de résistance que chaque mécanisme confère. Les souches VISA présentent une résistance intermédiaire, avec une concentration inhibitrice (CMI) minimale de 4 à 8 μg/mL, et le mécanisme implique une augmentation des cibles de vancomycine. Les souches VISA diminuent la réticulation des chaînes peptidiques dans la paroi cellulaire, augmentant ainsi le nombre de cibles de vancomycine qui piègent la vancomycine dans la paroi cellulaire externe. En revanche, les souches de VRSA acquièrent une résistance à la vancomycine par transfert horizontal de gènes de résistance provenant de l'ERV, une opportunité offerte aux personnes co-infectées à la fois par l'ERV et le SARM. Les VRSA présentent un niveau de résistance plus élevé, avec des CMI de 16 μg/mL ou plus. 6 Dans le cas des trois types de bactéries résistantes à la vancomycine, une identification clinique rapide est nécessaire afin de mettre en œuvre des procédures appropriées pour limiter la propagation. Les oxazolidinones, comme le linézolide, sont utiles pour le traitement de ces agents pathogènes opportunistes résistants à la vancomycine, ainsi que du SARM.
Agents pathogènes à Gram négatif à spectre étendu produisant de la β-lactamase
Les agents pathogènes à Gram négatif qui produisent des β-lactamases à spectre étendu (BLSE) présentent une résistance bien au-delà des simples pénicillines. Le spectre des β-lactamines inactivées par les BLSE permet de résister à toutes les pénicillines, aux céphalosporines, aux monobactames et aux combinaisons d'inhibiteurs de la β-lactamase, mais pas aux carbapénèmes. Ce qui est encore plus préoccupant, c'est que les gènes codant pour les BLSE se trouvent généralement sur des plasmides mobiles qui contiennent également des gènes de résistance à d'autres classes de médicaments (par exemple, les fluoroquinolones, les aminoglycosides, les tétracyclines) et peuvent être facilement transmis à d'autres bactéries par transfert horizontal de gènes. Ces bactéries multirésistantes font partie du microbiote intestinal de certaines personnes, mais elles sont également des causes importantes d'infections opportunistes chez les patients hospitalisés, d'où elles peuvent être transmises à d'autres personnes.
Bactéries à Gram négatif résistantes aux carbapénèmes
La présence d'entérobactéries résistantes aux carbapénèmes (CRE) et de résistance aux carbapénèmes chez d'autres bactéries gram-négatives (p. ex. P. aeruginosa, Acinetobacter baumannii, Stenotrophomonas maltophila) constitue une préoccupation croissante en matière de santé. Ces agents pathogènes développent une résistance aux carbapénèmes par divers mécanismes, notamment la production de carbapénèmes (β-lactamases à large spectre qui inactivent tous les β-lactames, y compris les carbapénèmes), l'efflux actif des carbapénèmes hors de la cellule et/ou la prévention de l'entrée des carbapénèmes par les canaux poriques. À l'instar des problèmes liés aux BLSE, les agents pathogènes gram-négatifs résistants aux carbapénèmes sont généralement résistants à de multiples classes d'antibactériens, et certains ont même développé une résistance panorésistante (résistance à tous les antibactériens disponibles). Les infections par des agents pathogènes gram-négatifs résistants aux carbapénèmes surviennent généralement dans les établissements de santé par interaction avec des personnes ou des dispositifs médicaux contaminés, ou à la suite d'une intervention chirurgicale.
Mycobacterium tuberculosis multirésistant
L'émergence de Mycobacterium tuberculosis (TB-MR) multirésistante et de Mycobacterium tuberculosis ultrarésistante (XDR-TB) est également une source de préoccupation mondiale majeure. Les souches de tuberculose multirésistante sont résistantes à la fois à la rifampicine et à l'isoniazide, l'association médicamenteuse généralement prescrite pour le traitement de la tuberculose. Les souches de tuberculose ultrarésistante sont également résistantes à toutes les fluoroquinolones et à au moins l'un des trois autres médicaments (amikacine, kanamycine ou capréomycine) utilisés comme traitement de deuxième intention, ce qui laisse très peu d'options thérapeutiques à ces patients. Les deux types d'agents pathogènes sont particulièrement problématiques chez les personnes immunodéprimées, y compris celles qui souffrent d'une infection par le VIH. Le développement de la résistance chez ces souches résulte souvent de l'utilisation incorrecte d'antimicrobiens pour le traitement de la tuberculose, en sélectionnant la résistance.
Exercice\(\PageIndex{2}\)
Comment la résistance aux médicaments entraîne-t-elle l'apparition de superbactéries ?
Pour en savoir plus sur les 18 principales menaces pharmacorésistantes qui pèsent sur les États-Unis, visitez le site Web du CDC.
Agriculture industrielle et résistance aux médicaments
Bien que l'élevage occupe depuis longtemps une place importante dans l'agriculture aux États-Unis, l'essor des opérations d'alimentation animale concentrée (CAFO) depuis les années 1950 a entraîné de nouveaux problèmes environnementaux, notamment la contamination de l'eau et de l'air par des déchets biologiques, et des problèmes éthiques concernant les droits des animaux sont également associés à la croissance d'animaux de cette manière. En outre, l'augmentation du nombre de CAFO implique l'utilisation intensive de médicaments antimicrobiens dans l'élevage du bétail. Les antimicrobiens sont utilisés pour prévenir le développement de maladies infectieuses dans les zones proches des CAFO ; toutefois, la majorité des antimicrobiens utilisés dans l'élevage industriel visent à favoriser la croissance, c'est-à-dire à élever de plus gros animaux.
Le mécanisme qui sous-tend cette croissance accrue n'est toujours pas clair. Ces antibiotiques ne sont pas nécessairement les mêmes que ceux utilisés en clinique pour les humains, mais ils sont structurellement apparentés aux médicaments utilisés pour les humains. Par conséquent, l'utilisation de médicaments antimicrobiens chez les animaux peut favoriser la résistance aux antimicrobiens, ces bactéries résistantes devenant ainsi résistantes à des médicaments généralement utilisés chez l'homme. Par exemple, l'utilisation de la tylosine chez les animaux semble privilégier les bactéries présentant également une résistance croisée à d'autres macrolides, y compris l'érythromycine, couramment utilisée chez l'homme.
Les concentrations de souches bactériennes résistantes aux médicaments générées par les CAFO augmentent dans l'eau et le sol entourant ces fermes. Si elles ne sont pas directement pathogènes chez l'homme, ces bactéries résistantes peuvent servir de réservoir d'éléments génétiques mobiles qui peuvent ensuite transmettre des gènes de résistance à des agents pathogènes humains. Heureusement, le processus de cuisson désactive généralement les antimicrobiens restant dans la viande, de sorte que les humains n'ingèrent généralement pas directement ces médicaments. Néanmoins, de nombreuses personnes réclament une utilisation plus judicieuse de ces médicaments, en facturant peut-être aux agriculteurs des frais d'utilisation afin de réduire leur consommation aveugle. En fait, en 2012, la FDA a publié des directives à l'intention des agriculteurs qui abandonnent volontairement l'utilisation de médicaments antimicrobiens, sauf sous la supervision d'un vétérinaire et lorsque cela est nécessaire pour garantir la santé des animaux. Bien que le respect des directives soit volontaire pour le moment, la FDA recommande ce qu'elle appelle une utilisation « judicieuse » des médicaments antimicrobiens chez les animaux producteurs d'aliments afin de réduire la résistance aux antimicrobiens.
Orientation clinique : 3e partie
Malheureusement, l'infection des voies urinaires de Marisa n'a pas disparu avec le traitement à la ciprofloxacine. Des tests de laboratoire ont montré que son infection était causée par une souche de Klebsiella pneumoniae présentant une résistance antimicrobienne significative. Le profil de résistance de ce K. pneumoniae incluait la résistance aux antibactériens de la classe des carbapénèmes, un groupe de β-lactamines généralement réservé au traitement des bactéries hautement résistantes. K. pneumoniae est un bâtonnet gram-négatif encapsulé opportuniste qui peut faire partie du microbiote normal du tractus intestinal, mais qui peut également provoquer un certain nombre de maladies, notamment la pneumonie et les infections urinaires.
Des tests de laboratoire spécifiques visant à détecter la production de carbapénémases ont été effectués sur les échantillons de Marisa et se sont révélés positifs. Sur la base de ce résultat et de ses antécédents médicaux, la production d'une carbapénemase connue sous le nom de métallo-β-lactamase de New Delhi (NDM) a été soupçonnée. Bien que l'origine de la carbapénémase de la NDM ne soit pas complètement connue, de nombreux patients infectés par des souches contenant de la NDM ont voyagé et ont été hospitalisés en Inde ou dans les pays voisins.
Exercice\(\PageIndex{1}\)
Comment les médecins détermineraient-ils quels types de médicaments antimicrobiens devraient être administrés ?
Concepts clés et résumé
- La résistance aux antimicrobiens est en hausse et résulte de la sélection de souches résistantes aux médicaments dans les environnements cliniques, de la surutilisation et de la mauvaise utilisation d'antibactériens, de l'utilisation de doses sous-thérapeutiques de médicaments antibactériens et de la faible observance des traitements médicamenteux antibactériens par les patients.
- Les gènes de résistance aux médicaments sont souvent transportés sur des plasmides ou dans des transposons qui peuvent être facilement transférés verticalement et entre microbes par transfert horizontal de gènes.
- Les modes courants de résistance aux médicaments antimicrobiens incluent la modification ou l'inactivation des médicaments, la prévention de l'absorption ou de l'efflux cellulaires, la modification de la cible, la surproduction de cibles ou le pontage enzymatique et le mimétisme de la cible.
- Des souches microbiennes problématiques présentant une résistance élevée aux antimicrobiens apparaissent ; bon nombre de ces souches peuvent résider en tant que membres du microbiote normal des individus, mais peuvent également provoquer des infections opportunistes. La transmission d'un grand nombre de ces souches microbiennes hautement résistantes se produit souvent en milieu clinique, mais elle peut également être acquise par la communauté.
Notes
- 1 D.H. Fong, A.M. Berghuis. « Promiscuité du substrat d'une enzyme de résistance aux antibiotiques aminoglycosides via le mimétisme cible. » EMBO Journal 21 n° 10 (2002) :2323—2331.
- 2 centres pour le contrôle et la prévention des maladies. « Résistance aux antibiotiques et aux antimicrobiens ». http://www.cdc.gov/drugresistance/index.html. Consulté le 2 juin 2016.
- 3 A. S. Kalokhe et coll. « Sensibilité aux médicaments antituberculeux multirésistants et tests diagnostiques moléculaires : une revue de la littérature. Journal américain des sciences médicales 345 no 2 (2013) :143-148.
- 4 centres pour le contrôle et la prévention des maladies. « Staphylococcus aureus résistant à la méthicilline (SARM) : informations générales sur le SARM dans la communauté. » http://www.cdc.gov/mrsa/community/index.html. Consulté le 2 juin 2016
- 5 FC Lowy. « Résistance aux antimicrobiens : l'exemple du Staphylococcus aureus. » Journal of Clinical Investigation 111 n° 9 (2003) :1265—1273.
- 6 centres pour le contrôle et la prévention des maladies. « Infections associées aux soins de santé (HIA) : informations générales sur VISA/VRSA ». www.cdc.gov/HAI/Organisms/VIS... visa_vrsa.html. Consulté le 2 juin 2016.


