9.4 : Traduction
- Page ID
- 186327
La synthèse des protéines est l'un des processus métaboliques les plus énergivores d'une cellule. À leur tour, les protéines représentent plus de masse que tout autre composant des organismes vivants (à l'exception de l'eau), et les protéines remplissent une grande variété de fonctions cellulaires. Le processus de traduction, ou synthèse des protéines, consiste à décoder un message d'ARNm en un produit polypeptidique. Les acides aminés sont enchaînés de manière covalente sur des longueurs allant d'environ 50 acides aminés à plus de 1 000.
La machine de synthèse des protéines
Outre le modèle d'ARNm, de nombreuses autres molécules contribuent au processus de traduction. La composition de chaque composant peut varier d'une espèce à l'autre ; par exemple, les ribosomes peuvent être constitués d'un nombre différent d'ARN ribosomaux (ARNr) et de polypeptides selon l'organisme. Cependant, les structures générales et les fonctions de la machinerie de synthèse des protéines sont comparables des bactéries aux cellules humaines. La traduction nécessite la saisie d'un modèle d'ARNm, de ribosomes, d'ARNt et de divers facteurs enzymatiques (Figure\(\PageIndex{1}\)).
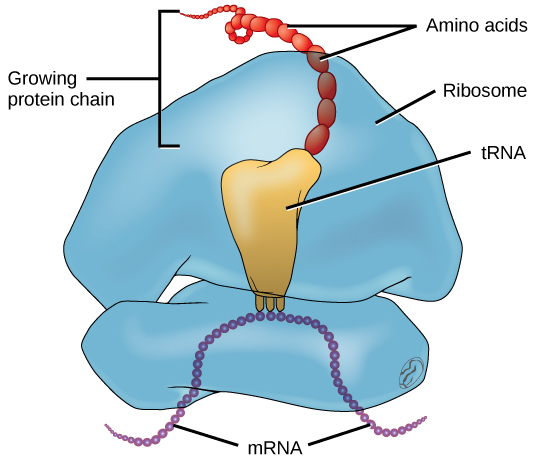
Chez E. coli, 200 000 ribosomes sont présents dans chaque cellule à un moment donné. Un ribosome est une macromolécule complexe composée d'ARNr structuraux et catalytiques et de nombreux polypeptides distincts. Chez les eucaryotes, le nucléole est complètement spécialisé dans la synthèse et l'assemblage des ARNr.
Les ribosomes sont situés dans le cytoplasme des procaryotes ainsi que dans le cytoplasme et le réticulum endoplasmique des eucaryotes. Les ribosomes sont composés d'une grande et d'une petite sous-unité qui se réunissent pour la traduction. La petite sous-unité est responsable de la liaison à la matrice d'ARNm, tandis que la grande sous-unité lie séquentiellement les ARNt, un type de molécule d'ARN qui amène les acides aminés à la chaîne croissante du polypeptide. Chaque molécule d'ARNm est traduite simultanément par de nombreux ribosomes, tous synthétisant des protéines dans la même direction.
Selon les espèces, 40 à 60 types d'ARNt existent dans le cytoplasme. En tant qu'adaptateurs, des ARNt spécifiques se lient aux séquences de la matrice d'ARNm et ajoutent l'acide aminé correspondant à la chaîne polypeptidique. Par conséquent, les ARNt sont les molécules qui « traduisent » réellement le langage de l'ARN dans le langage des protéines. Pour que chaque ARNt fonctionne, son acide aminé spécifique doit être lié à celui-ci. Lors du processus de « chargement » de l'ARNt, chaque molécule d'ARNt est liée à son acide aminé approprié.
Le code génétique
Pour résumer ce que nous savons jusqu'à présent, le processus cellulaire de transcription génère de l'ARN messager (ARNm), une copie moléculaire mobile d'un ou de plusieurs gènes avec un alphabet composé des lettres A, C, G et uracile (U). La traduction du modèle d'ARNm convertit les informations génétiques basées sur les nucléotides en un produit protéique. Les séquences protéiques se composent de 20 acides aminés courants ; par conséquent, on peut dire que l'alphabet protéique se compose de 20 lettres. Chaque acide aminé est défini par une séquence de trois nucléotides appelée codon triplet. La relation entre un codon nucléotidique et l'acide aminé correspondant est appelée code génétique.
Étant donné les différents nombres de « lettres » dans l'ARNm et dans les « alphabets » des protéines, les combinaisons de nucléotides correspondaient à des acides aminés uniques. L'utilisation d'un code à trois nucléotides signifie qu'il existe un total de 64 (4 × 4 × 4) combinaisons possibles ; par conséquent, un acide aminé donné est codé par plus d'un triplet de nucléotides (Figure\(\PageIndex{2}\)).
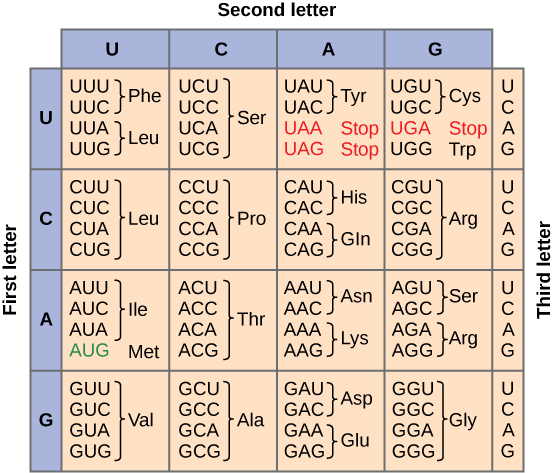
Trois des 64 codons interrompent la synthèse des protéines et libèrent le polypeptide de la machinerie de traduction. Ces triplets sont appelés codons stop. Un autre codon, AUG, possède également une fonction spéciale. En plus de spécifier l'acide aminé méthionine, il sert également de codon de départ pour initier la traduction. Le cadre de lecture pour la traduction est défini par le codon AUG start à proximité de l'extrémité 5' de l'ARNm. Le code génétique est universel. À quelques exceptions près, pratiquement toutes les espèces utilisent le même code génétique pour la synthèse des protéines, ce qui prouve clairement que toutes les formes de vie sur Terre ont une origine commune.
Le mécanisme de synthèse des protéines
Tout comme pour la synthèse des ARNm, la synthèse des protéines peut être divisée en trois phases : initiation, élongation et terminaison. Le processus de traduction est similaire chez les procaryotes et les eucaryotes. Nous explorerons ici comment se produit la traduction chez E. coli, un procaryote représentatif, et nous préciserons toute différence entre la traduction procaryote et la traduction eucaryote.
La synthèse des protéines commence par la formation d'un complexe initiatique. Chez E. coli, ce complexe implique la petite sous-unité du ribosome, la matrice d'ARNm, trois facteurs d'initiation et un ARNt initiateur spécial. L'ARNt initiateur interagit avec le codon AUG start et se lie à une forme spéciale de l'acide aminé méthionine qui est généralement éliminée du polypeptide une fois la traduction terminée.
Chez les procaryotes et les eucaryotes, les bases de l'élongation des polypeptides sont les mêmes. Nous allons donc passer en revue l'allongement du point de vue de E. coli. La grande sous-unité ribosomale d'E. coli se compose de trois compartiments : le site A lie les ARNt chargés entrants (ARNt avec leurs acides aminés spécifiques attachés). Le site P lie les ARNt chargés porteurs d'acides aminés qui ont formé des liaisons avec la chaîne polypeptidique en croissance mais qui ne se sont pas encore dissociés de leur ARNt correspondant. Le site E libère des ARNt dissociés afin qu'ils puissent être rechargés en acides aminés libres. Le ribosome déplace un codon à la fois, catalysant chaque processus qui se produit dans les trois sites. À chaque étape, un ARNt chargé entre dans le complexe, le polypeptide devient un acide aminé plus long et un ARNt non chargé part. L'énergie de chaque liaison entre les acides aminés est dérivée du GTP, une molécule similaire à l'ATP (Figure\(\PageIndex{3}\)). Étonnamment, l'appareil de traduction d'E. coli ne prend que 0,05 seconde pour ajouter chaque acide aminé, ce qui signifie qu'un polypeptide de 200 acides aminés peut être traduit en seulement 10 secondes.
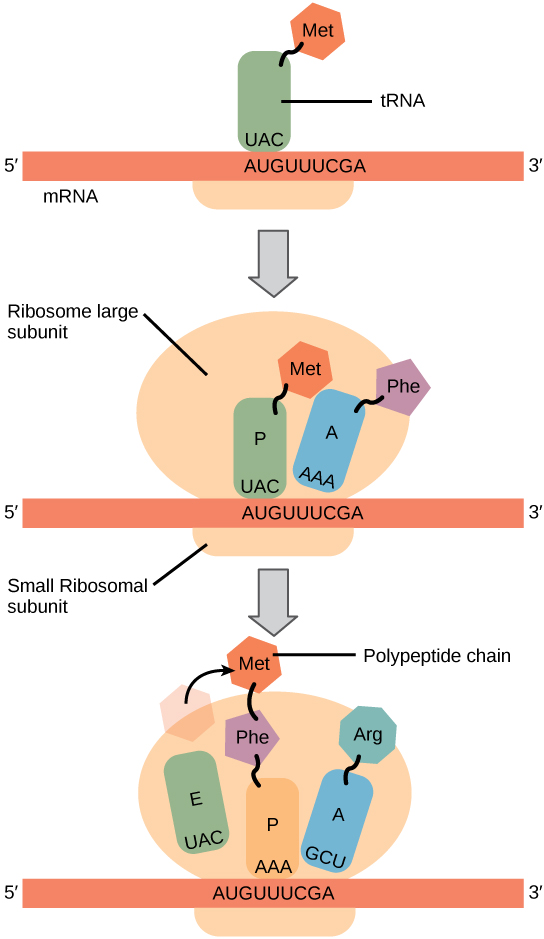
La traduction se termine lorsqu'un codon stop (UAA, UAG ou UGA) est rencontré. Lorsque le ribosome rencontre le codon stop, le polypeptide en croissance est libéré et les sous-unités du ribosome se dissocient et quittent l'ARNm. Une fois que de nombreux ribosomes ont terminé la traduction, l'ARNm est dégradé afin que les nucléotides puissent être réutilisés dans une autre réaction de transcription.
CONCEPT EN ACTION
Transcrivez un gène et traduisez-le en protéine à l'aide de l'appariement complémentaire et du code génétique de ce site.
Résumé
Le dogme central décrit le flux d'informations génétiques dans la cellule, des gènes à l'ARNm en passant par les protéines. Les gènes sont utilisés pour fabriquer l'ARNm par le processus de transcription ; l'ARNm est utilisé pour synthétiser des protéines par le processus de traduction. Le code génétique est la correspondance entre le codon de l'ARNm à trois nucléotides et un acide aminé. Le code génétique est « traduit » par les molécules d'ARNt, qui associent un codon spécifique à un acide aminé spécifique. Le code génétique est dégénéré car 64 codons triplets dans l'ARNm ne spécifient que 20 acides aminés et trois codons stop. Cela signifie que plus d'un codon correspond à un acide aminé. Presque toutes les espèces de la planète utilisent le même code génétique.
Les acteurs de la traduction incluent le modèle d'ARNm, les ribosomes, les ARNt et divers facteurs enzymatiques. La petite sous-unité ribosomale se lie au modèle d'ARNm. La traduction commence au début de l'AUG sur l'ARNm. La formation de liaisons se produit entre les acides aminés séquentiels spécifiés par le modèle d'ARNm en fonction du code génétique. Le ribosome accepte les ARNt chargés et, en marchant le long de l'ARNm, il catalyse la liaison entre le nouvel acide aminé et l'extrémité du polypeptide en croissance. L'ARNm entier est traduit en « étapes » de trois nucléotides du ribosome. Lorsqu'un codon stop est détecté, un facteur de libération lie et dissocie les composants et libère la nouvelle protéine.
Lexique
- codon
- trois nucléotides consécutifs dans l'ARNm qui spécifient l'ajout d'un acide aminé spécifique ou la libération d'une chaîne polypeptidique pendant la traduction
- code génétique
- les acides aminés qui correspondent aux codons à trois nucléotides de l'ARNm
- ARNr
- ARN ribosomal ; molécules d'ARN qui se combinent pour former une partie du ribosome
- arrêter le codon
- l'un des trois codons d'ARNm qui spécifie la fin de la traduction
- codon de démarrage
- l'AUG (ou, rarement, le GUG) sur un ARNm à partir duquel commence la traduction ; indique toujours la méthionine
- ARNt
- ARN de transfert ; molécule d'ARN qui contient une séquence d'anticodons à trois nucléotides spécifique à associer au codon d'ARNm et qui se lie également à un acide aminé spécifique


