11.2: DNA 复制
- Page ID
- 200149
学习目标
- 解释半保守 DNA 复制的意义
- 解释为什么 DNA 复制是双向的,同时包括前导链和滞后链
- 解释为什么冈崎碎片会形成
- 描述 DNA 复制过程和相关酶的功能
- 确定细菌和真核生物中 DNA 复制之间的区别
- 解释滚动圈复制的过程
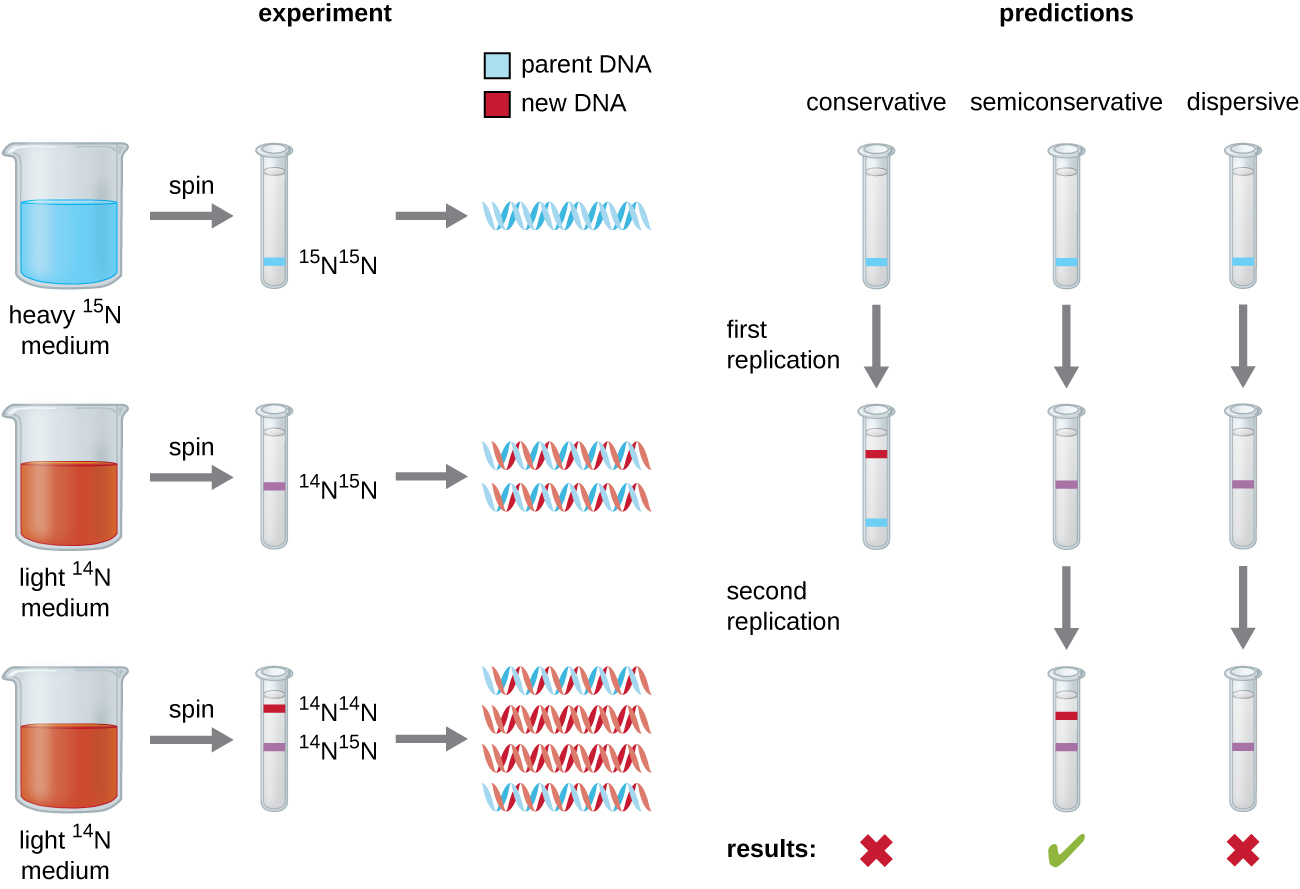
詹姆斯·沃森和弗朗西斯·克里克在1953年对双螺旋结构的阐释为在复制过程中如何复制DNA提供了暗示。 分离双螺旋链将为合成新的互补链提供两个模板,但尚不清楚新的 DNA 分子究竟是如何构造的。 在一个模型中,即半保守复制,双螺旋的两条链在DNA复制过程中分开,每条链充当复制新的互补链的模板;复制后,每个双链 DNA 包括一条亲本或 “旧” 链和一条 “新” 链。 还提出了两种相互竞争的模型:保守模型和分散模型,如图所示\(\PageIndex{1}\)。

马修·梅塞尔森(1930—)和富兰克林·斯塔尔(1929—)在 1958 年设计了一项实验,以测试这些模型中哪一个正确代表了 DNA 复制(图\(\PageIndex{2}\))。 他们在一种含有 “重” 氮同位素(15 N)的培养基中培育了好几代人的大肠杆菌,该同位素被掺入氮碱基中,最终融入到DNA中。 这标记了父母的DNA。 然后,大肠杆菌培养物被转移到含有 14 N 的培养基中,并允许其生长一代。 细胞被采集并分离出DNA。 DNA通过超速离心分离,在此期间,DNA根据其密度形成条带。 预计在 15 N 中生长的 DNA 将形成比在 14 N 中生长的 DNA 更高的密度位置的波段。梅塞尔森和斯塔尔指出,在 14 N 中生长了一代之后,观察到的单条带在完全生长的细胞 DNA 之间处于中间位置15 N 或 14 N。这表明要么是半保守的复制模式,要么是分散的复制模式。 有些细胞被允许在 14 N 中再生长一代,然后再次旋转。 从在 14 N 中生长了两代的细胞中提取的 DNA 形成了两个带:一个 DNA 带位于 15 N 和 14 N 之间的中间位置,另一个对应于 14 N DNA 的带。 只有当 DNA 以半保守的方式复制时,才能解释这些结果。 因此,排除了其他两个模型。 通过这项实验,我们现在知道在DNA复制过程中,构成双螺旋的两条链中的每条都充当了复制新链的模板。 新的链条将与父系或 “旧” 链互补。 由此产生的DNA分子具有相同的序列,平均分为两个子细胞。
练习\(\PageIndex{1}\)
如果梅塞尔森和斯塔尔在第一代人之后发现了两条DNA带,他们的实验会得出什么结论?
细菌中的 DNA 复制
已经对细菌中的DNA复制进行了很好的研究,这主要是因为基因组体积小,而且存在突变体。 大肠杆菌在单个圆形染色体中有460万个碱基对(Mbp),所有碱基对(Mbp)都可以在大约 42 分钟内复制,从单一复制源开始,然后双向(即双向)绕圈移动。 这意味着每秒添加大约 1000 个核苷酸。 该过程非常快速,几乎没有错误。
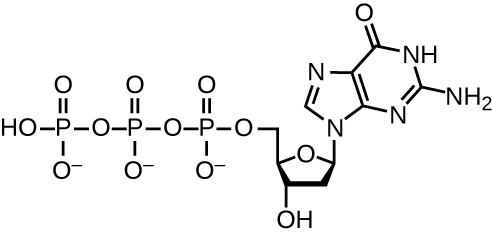
DNA复制使用大量的蛋白质和酶(表\(\PageIndex{1}\))。 其中一个关键参与者是DNA聚合酶,也称为DNA pol。 在细菌中,已知有三种主要类型的 DNA 聚合酶:DNA pol I、DNA pol II 和 DNA pol III。 现在众所周知,DNA pol III 是 DNA 合成所需的酶;DNA pol I 和 DNA pol II 主要是修复所必需的。 DNA pol III 将脱氧核糖核苷酸逐一添加到不断增长的 DNA 链的 3'-OH 组中,每个脱氧核糖核苷酸都与模板链上的核苷酸互补。 添加这些核苷酸需要能量。 这种能量存在于附着在每个核苷酸(三磷酸核苷酸)上的三个磷酸基团的键中,类似于三磷酸腺苷(ATP)的磷酸键中储存能量的方式(图\(\PageIndex{3}\))。 当磷酸盐之间的键破裂而二磷酸被释放时,释放的能量允许通过传入的核苷酸和不断增长的DNA链上的游离3'-OH基团之间的脱水合成形成共价磷酸二酯键。
启动
复制的开始发生在称为复制起源的特定核苷酸序列上,其中各种蛋白质结合以开始复制过程。 大肠杆菌在其一条染色体上有单一的复制来源(与大多数原核生物一样),称为 OriC。 复制起源长约 245 个碱基对,富含腺嘌呤胸腺嘧啶 (AT) 序列。
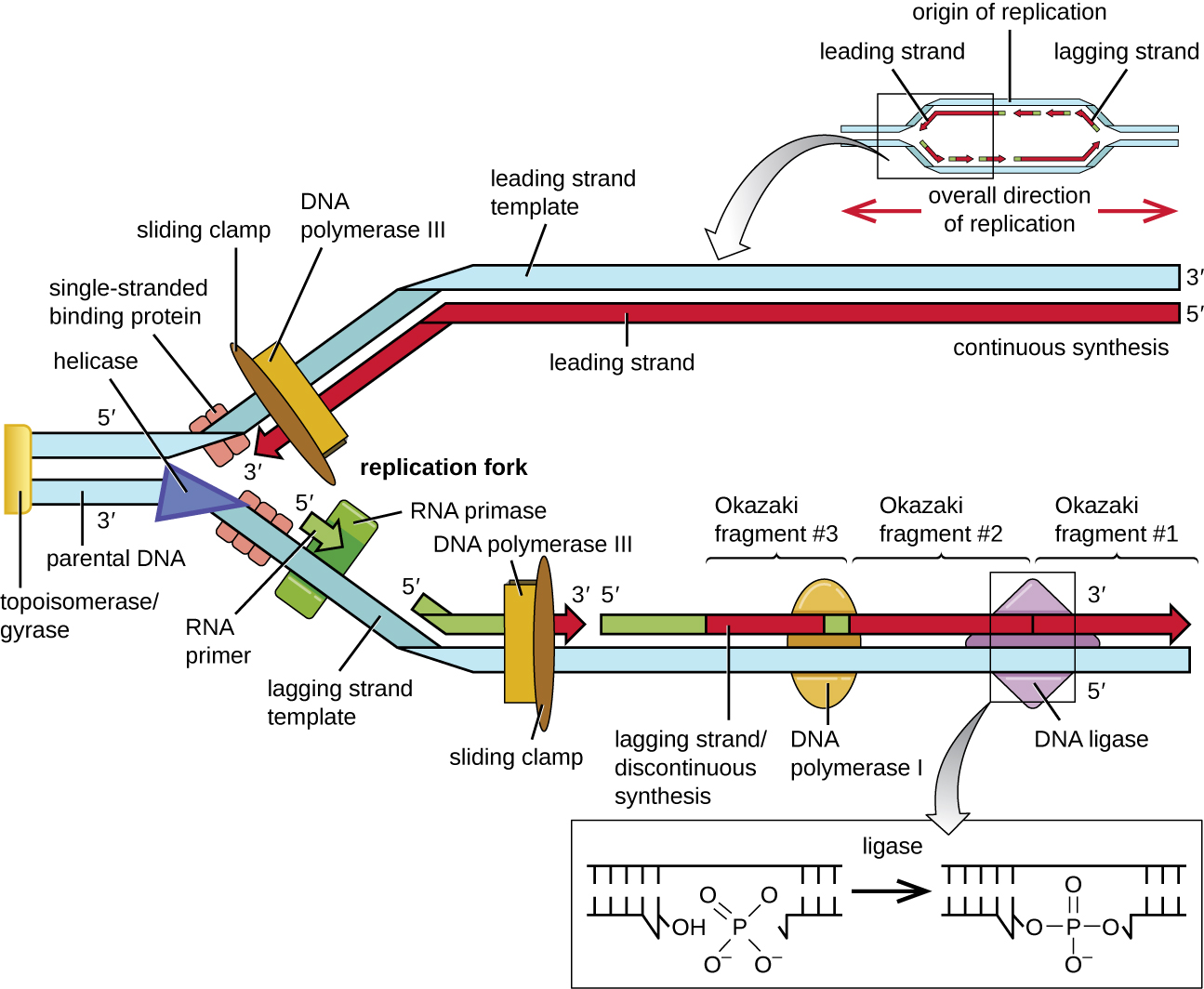
一些与复制起源结合的蛋白质对于使DNA的单链区域易于复制非常重要。 染色体DNA通常包裹在组蛋白(真核生物和古细菌中)或组蛋白样蛋白(在细菌中)周围,自身被超线圈或广泛包裹和扭曲。 这种包装使得 DNA 分子中的信息不可访问。 但是,称为拓扑异构酶的酶会改变染色体的形状和超级线圈。 为了开始细菌 DNA 复制,超线圈的染色体通过拓扑异构酶 II(也称为 DNA 回旋酶)放松。 然后,一种叫做解旋酶的酶通过破坏含氮碱基对之间的氢键来分离 DNA 链。 回想一下,与鸟嘌呤-胞嘧啶(GC)序列相比,AT 序列的氢键较少,因此相互作用较弱。 这些酶需要 ATP 水解。 随着DNA的开放,形成了称为复制叉的Y形结构。 在复制起点形成两个复制叉,允许双向复制,并形成用透射电子显微镜观察时看起来像气泡的结构;因此,这种结构被称为复制气泡。 每个复制叉附近的 DNA 都涂有单链结合蛋白,以防止单链 DNA 倒带成双螺旋。
一旦在复制起点可以获得单链 DNA,就可以开始复制。 但是,DNA pol III 只能在 5' 到 3' 方向添加核苷酸(新的 DNA 链只能向这个方向延伸)。 这是因为DNA聚合酶需要一个游离的3'-OH基团,它可以通过在3'-OH末端和下一个核苷酸的5'磷酸之间形成共价磷酸二酯键来向该基团添加核苷酸。 这也意味着,如果没有游离的 3'-OH 基团,它就无法添加核苷酸,单链 DNA 就是这种情况。 这个问题在提供免费的 3'-OH 端的 RNA 序列的帮助下得以解决。 由于该序列允许开始 DNA 合成,因此它被恰当地称为引物。 引物长度为五到十个核苷酸,与亲本或模板DNA互补。 它由 RNA primase(一种 RNA 聚合酶)合成。 与 DNA 聚合酶不同,RNA 聚合酶不需要游离的 3'-OH 基团即可合成 RNA 分子。 既然引物提供了游离的 3'-OH 基团,DNA 聚合酶 III 现在可以扩展这个 RNA 引物,逐一添加与模板链互补的 DNA 核苷酸(图\(\PageIndex{1}\))。
伸长率
在 DNA 复制伸长期间,核苷酸的添加以每秒大约 1000 个核苷酸的最大速率发生。 DNA 聚合酶 III 只能在 5' 到 3' 方向延伸,这给复制分叉带来了问题。 DNA双螺旋是反平行的;也就是说,一条链朝5'到3'方向定向,另一条链朝3'到5'方向定向(参见 DNA的结构和功能)。 在复制过程中,一条与3'至5'亲本DNA链互补的链向复制叉连续合成,因为聚合酶可以在这个方向上添加核苷酸。 这种连续合成的链被称为前导链。 另一条链与5'至3'的亲本DNA互补,从复制叉生长,因此聚合酶必须向复制叉移动,才能开始向新引物添加碱基,再次朝远离复制分叉的方向添加碱基。 它这样做直到碰到先前合成的股线然后再次向后移动(图\(\PageIndex{4}\))。 这些步骤产生称为冈崎片段的小 DNA 序列片段,每个片段由 RNA 引物分离。 冈崎碎片以日本研究小组和已婚夫妇黎二和冈崎常子的名字命名,他们于 1966 年首次发现了它们。 带有冈崎碎片的链被称为滞后链,据说其合成是不连续的。
前导链可以单独从一根引物延伸,而滞后的股线需要为每个较短的冈崎碎片添加新的引物。 滞后股线的总方向将为3英尺至5英尺,而前导股线的总方向为5英尺至3英尺。 一种叫做滑动夹的蛋白质在DNA聚合酶继续添加核苷酸时将其固定在适当的位置。 滑动夹是一种环形蛋白质,它与 DNA 结合并将聚合酶固定在适当的位置。 除了起始作用外,拓扑异构酶还可以防止 DNA 双螺旋在 DNA 开放时在复制分叉之前过度缠绕;它通过在 DNA 螺旋中造成暂时的缺口,然后将其重新密封。 随着合成的进行,RNA 引物被 DNA 所取代。 引物通过DNA聚合酶I的外切核酸酶活性被去除,空隙被填补。 新合成的 DNA(取代了 RNA 引物)和先前合成的 DNA 之间残留的缺口被 DNA 连接酶封住,该酶催化一个 DNA 片段的 3'-OH 末端与另一个片段的 5' 磷酸末端之间形成共价磷酸二酯连接,稳定 DNA 分子的磷酸糖骨干。
终止
一旦复制了完整的染色体,就必须终止 DNA 复制。 尽管对复制的启动知之甚少,但对终止过程知之甚少。 复制后,由此产生的原核生物的完整圆形基因组被串联在一起,这意味着环状DNA染色体是互锁的,必须相互分离。 这是通过细菌拓扑异构酶 IV 的活性实现的,该酶将双链断裂引入 DNA 分子,使它们能够相互分离;然后,该酶会重新密封环状染色体。 concatemers 的分辨率是原核生物 DNA 复制所独有的问题,因为它们是环状染色体。 由于细菌 DNA 回旋酶和拓扑异构酶 IV 与它们的真核生物对应物不同,因此这些酶可作为一类称为喹诺酮类的抗微生物药物的靶标。
| 酶或因子 | 函数 |
|---|---|
| DNA pol I | Exonuclease 活性可去除 RNA 引物并用新合成的 DNA 取而代之 |
| DNA pol I | 在 5' 到 3' 方向添加核苷酸的主要酶 |
| Helicase | 通过破坏含氮碱基之间的氢键来打开 DNA 螺旋 |
| Ligase | 封住滞后链上冈崎片段之间的间隙,形成一条连续的 DNA 链 |
| Primase | 合成开始复制所需的 RNA 引物 |
| 单链结合蛋白 | 与单链 DNA 结合以防止 DNA 链之间的氢键,重整双链 DNA |
| 滑动钳 | 添加核苷酸时有助于将 DNA pol III 固定在适当的位置 |
| 拓扑异构酶 II(DNA 回旋酶) | 放松超线圈的染色体,使 DNA 更容易获得,开始复制;通过造成 DNA 断裂然后重新密封,帮助缓解 DNA 解卷时对 DNA 的压力 |
| 拓扑异构酶 IV | 将单链裂解引入串联的染色体以将其相互释放,然后重新密封 DNA |
练习\(\PageIndex{2}\)
- 哪种酶会破坏将两条 DNA 链固定在一起的氢键,从而实现复制?
- 是沿着复制分叉开口的方向合成的滞后股线还是前导股线?
- 哪种酶负责去除新复制的细菌 DNA 中的 RNA 引物?
真核生物中的 DNA 复制
真核生物基因组比原核生物基因组复杂得多,也更大,通常由多个线性染色体组成(表\(\PageIndex{2}\))。 例如,人类基因组每组单倍体染色体有30亿个碱基对,在复制过程中插入了60亿个碱基对。 每条真核生物染色体上有多个复制来源(图\(\PageIndex{5}\));人类基因组有 30,000 到 50,000 个复制来源。 复制速度约为每秒 100 个核苷酸,比原核复制慢 10 倍。
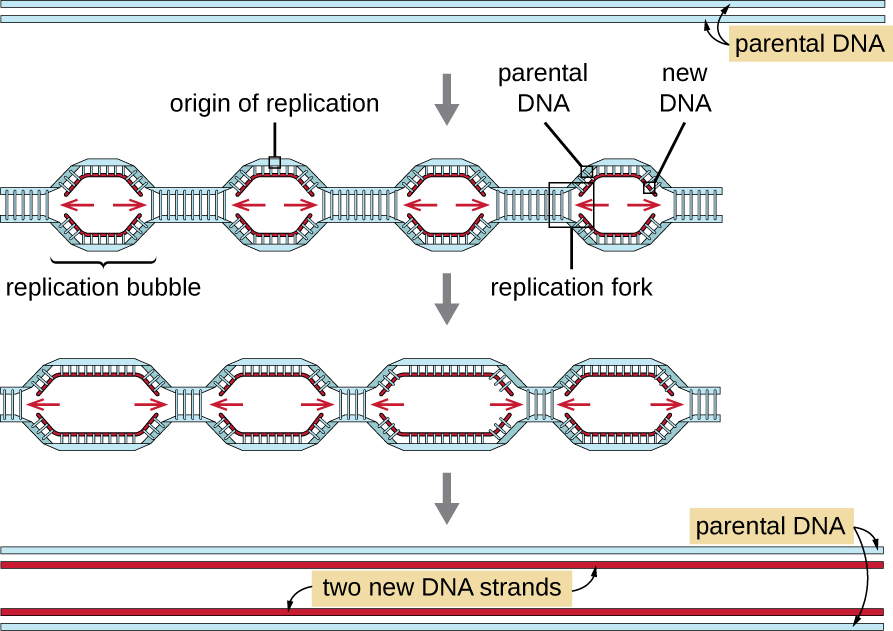
在真核生物中复制的基本步骤与在原核生物中复制的基本步骤相同。 在开始复制之前,必须将 DNA 作为模板提供。 真核生物DNA经过高度超线圈和包装,这得到了包括组蛋白在内的许多蛋白质的促进(参见细胞基因组的结构和功能)。 在复制的起源,由包括解旋酶在内的多种蛋白质组成的复制前复合物会形成并招募参与复制启动的其他酶,包括用于放松超级线圈的拓扑异构酶、单链结合蛋白、RNA primase 和 DNA 聚合酶。 复制开始后,在与原核生物中发现的过程相似的过程中,真核生物 DNA 聚合酶促进了伸长。 前导链由真核聚合酶pol α连续合成,而滞后链则由pol ε合成。 滑动钳制蛋白将 DNA 聚合酶固定在适当的位置,这样它就不会从 DNA 上掉下来。 核糖核酸酶 H(RNase H)不是细菌中的 DNA 聚合酶,而是去除 RNA 引物,然后将其替换为 DNA 核苷酸。 剩下的空隙被DNA连接酶封住。
由于真核生物染色体是线性的,因此人们可能会期望它们的复制会更加简单。 与原核生物一样,真核生物 DNA 聚合酶只能在 5' 到 3' 方向添加核苷酸。 在前导链中,合成一直持续到它到达染色体的末端或另一个朝相反方向发展的复制叉。 在滞后链上,DNA是在短时间内合成的,每个序列都是由单独的引物引发的。 当复制分叉到达线性染色体的末端时,没有地方为要在染色体末端复制的 DNA 片段制作引物。 因此,这些末端保持不成对状态,随着时间的推移,随着细胞的继续分裂,它们可能会逐渐变短。
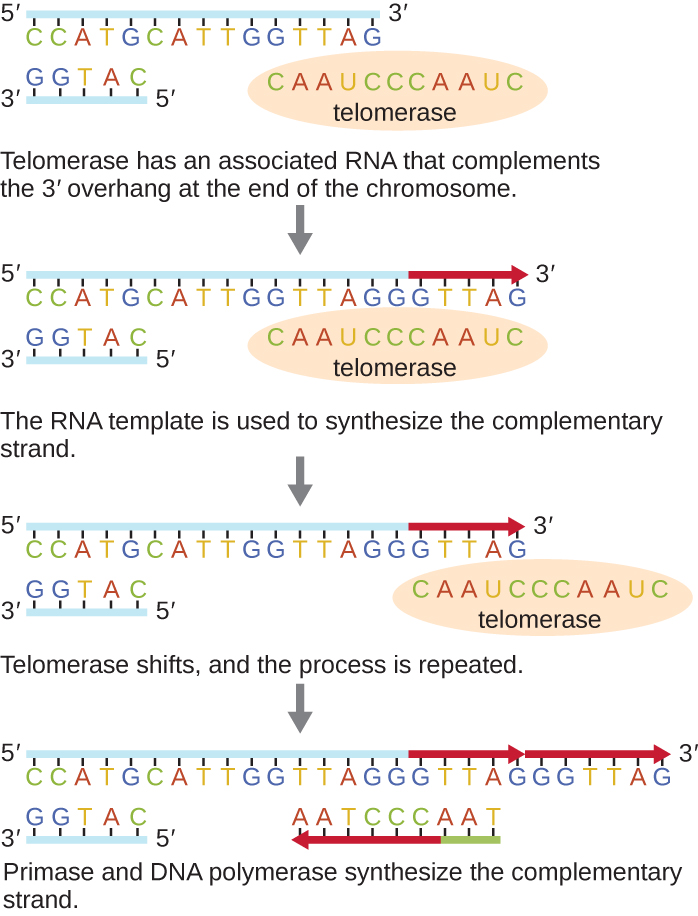
线性染色体的末端被称为端粒,由非编码重复序列组成。 端粒保护编码序列免于在细胞继续分裂时丢失。 在人类中,六碱基对序列 TTAGGG 被重复 100 到 1000 次以形成端粒。 端粒酶的发现(图\(\PageIndex{6}\))阐明了我们对如何维持染色体末端的理解。 端粒酶含有催化部分和内置 RNA 模板。 它附着在染色体的末端,并在DNA链的3'末端添加RNA模板的互补碱基。 一旦滞后链模板的3'末端足够拉长,DNA聚合酶就可以添加与染色体末端互补的核苷酸。 通过这种方式,可以复制染色体的末端。 在人类中,端粒酶通常在生殖细胞和成体干细胞中具有活性;它在成体体细胞中不活跃,可能与这些细胞的衰老有关。 包括真菌和原生动物在内的真核微生物也会产生端粒酶以维持染色体的完整性。 由于发现端粒酶及其作用,伊丽莎白·布莱克本(1948—)于2009年获得了诺贝尔医学或生理学奖。
| 财产 | 细菌 | 真核生物 |
|---|---|---|
| 基因组结构 | 单环染色体 | 多条线性染色体 |
| 每条染色体的起源数 | 单身 | 多个 |
| 复制速率 | 每秒 1000 个核苷酸 | 每秒 100 个核苷酸 |
| 端粒酶 | 不在场 | 当下 |
| 去除 RNA 引物 | DNA pol I | 核糖核酸酶 H |
| 股线伸长率 | DNA pol I | pol β、pol α |
练习\(\PageIndex{3}\)
- 真核生物和原核生物的复制起源有何不同?
- 哪些聚合酶负责真核复制过程中的 DNA 合成?
- 在真核生物的染色体末端发现了什么?为什么?
染色体外元素的 DNA 复制:质粒和病毒
为了复制其核酸,质粒和病毒经常使用原核生物基因组描述的DNA复制模式的变异。 有关各种病毒复制策略的更多信息,请参阅病毒生命周期。
滚动圈复制
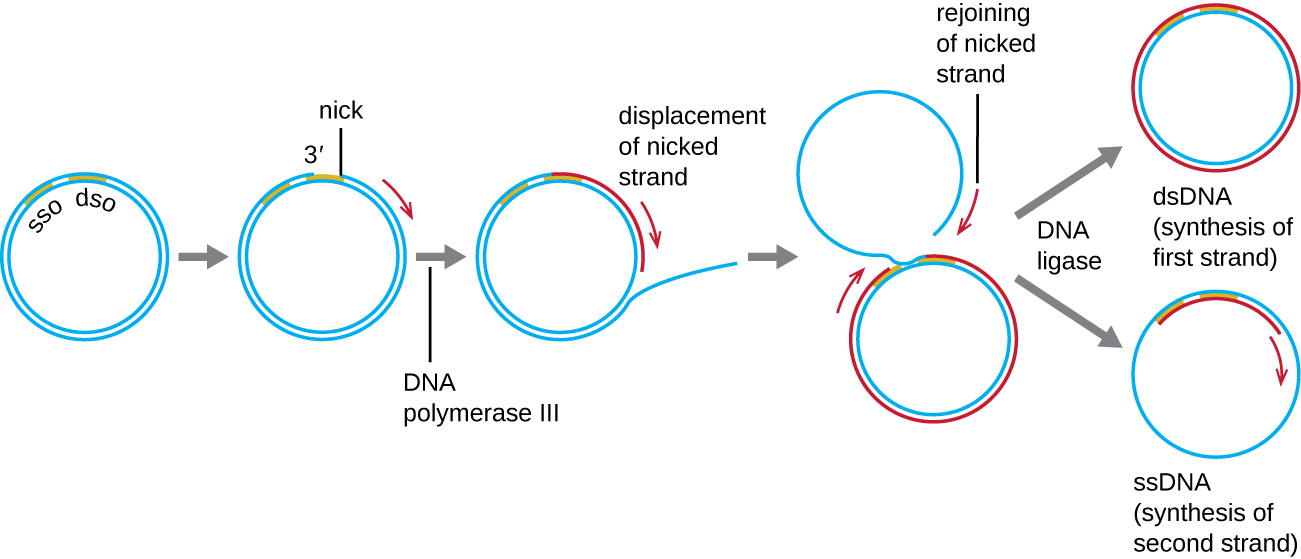
许多细菌质粒(参见原核细胞的独特特征)通过类似于复制细菌染色体的过程进行复制,而其他质粒、几种噬菌体和一些真核生物病毒则使用滚圈复制(图\(\PageIndex{7}\))。 质粒的循环性质和某些病毒基因组在感染时循环化使之成为可能。 滚动圆复制始于在双链起源(dso)位点对一条双链圆分子进行酶切口。 在细菌中,DNA聚合酶III与缺口链的3'-OH基团结合,并开始使用未划痕的链作为模板单向复制DNA,从而取代有缺口的链。 在原始缺口部位完成 DNA 复制会导致缺口链完全移位,然后它可能会再循环成单链 DNA 分子。 然后,RNA primase 合成引物,在单链 DNA(ssDNA)分子的单链起源(ssDNA)位点启动 DNA 复制,从而产生与另一个环形 DNA 分子相同的双链 DNA(dsDNA)分子。
练习\(\PageIndex{4}\)
滚动圈复制中有滞后的链条吗? 为什么或者为什么不呢?
关键概念和摘要
- DNA 复制过程是半保守的,它会产生两个 DNA 分子,每个分子都有一条 DNA 亲本链和一条新合成的链。
- 在细菌中,复制的开始始于复制的起源,其中超线圈的DNA由 DNA回旋酶解开,由解旋酶单链,并通过单链结合蛋白结合以维持其单链滞留状态。 Primase 合成了一个短的 RNA 引物,提供了一个游离的 3'-OH 基团,DNA 聚合酶 III 可以在其中添加 DNA 核苷酸。
- 在伸长过程中,从单个引物中连续合成前导DNA链。 滞后的链条是在短的冈崎碎片中不连续地合成的,每个片段都需要自己的引物。 细菌 DNA 聚合酶 I 会移除 RNA 引物并用 DNA 核苷酸代替,而 DNA 连接酶封住了这些片段之间的空隙。
- 终止细菌的复制涉及通过拓扑异构酶 IV 分解环状的 DNA concatemers 以释放两个环状染色体拷贝。
- 真核生物通常有多个线性染色体,每条染色体都有多个复制来源。 总体而言,真核生物中的复制与原核生物中的复制相似。
- 真核染色体的线性性质要求端粒保护染色体末端的基因。 在某些细胞类型中,端@@ 粒酶可延伸端粒,防止其降解。
- 滚环复制是一种用于复制某些质粒的圆形 DNA 分子的快速单向 DNA 合成。


