14.2: DNA 结构和测序
- Page ID
- 203010
培养技能
- 描述 DNA 的结构
- 解释桑格的 DNA 测序方法
- 讨论真核生物和原核生物 DNA 的异同
DNA的基石是核苷酸。 核苷酸的重要成分是含氮碱、脱氧核糖(5-碳糖)和磷酸基团(图\(\PageIndex{1}\))。 核苷酸根据含氮碱命名。 含氮碱可以是嘌呤,例如腺嘌呤(A)和鸟嘌呤(G),也可以是嘧啶,例如胞嘧啶(C)和胸腺嘧啶(T)。
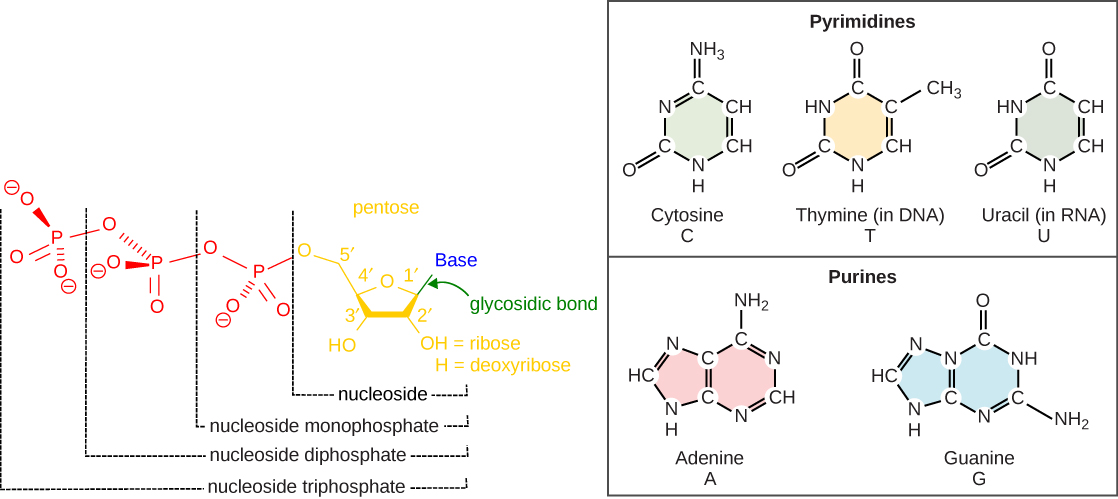
核苷酸通过称为磷酸二酯键或连接的共价键相互结合。 嘌呤具有双环结构,六元环融合到五元环上。 嘧啶体积较小;它们具有单个六元环结构。 五碳糖的碳原子编号为 1'、2'、3'、4' 和 5'(1' 被读为 “一个素数”)。 磷酸盐残基附着在一个核苷酸的一个糖的5'碳的羟基和下一个核苷酸的糖的3'碳的羟基上,从而形成5'-3'磷酸二酯键。
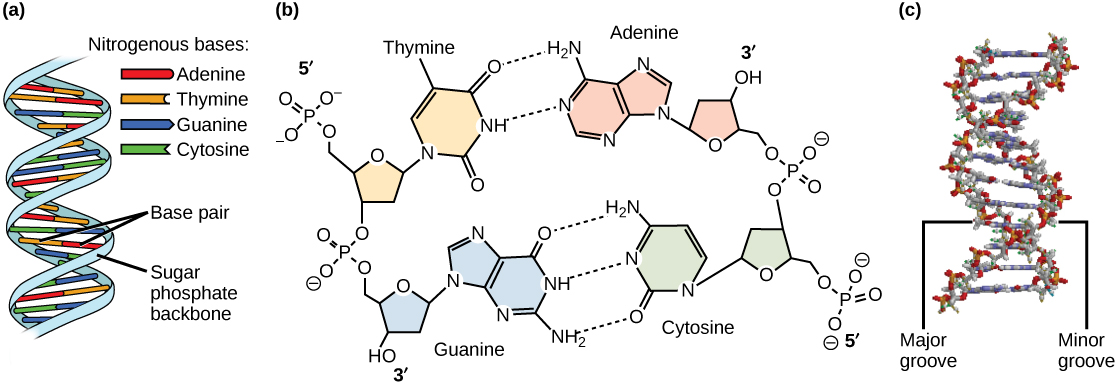
20 世纪 50 年代,弗朗西斯·克里克和詹姆斯·沃森共同确定了英格兰剑桥大学的 DNA 结构。 莱纳斯·鲍林和莫里斯·威尔金斯等其他科学家也在积极探索这个领域。 鲍林利用X射线晶体学发现了蛋白质的二级结构。 在威尔金斯的实验室里,研究人员罗莎琳德·富兰克林正在使用X射线衍射方法来了解DNA的结构。 沃森和克里克能够根据富兰克林的数据拼凑出DNA分子的难题,因为克里克还研究了X射线衍射(图\(\PageIndex{2}\))。 1962 年,詹姆斯·沃森、弗朗西斯·克里克和莫里斯·威尔金斯被授予诺贝尔医学奖。 不幸的是,那时富兰克林已经去世,诺贝尔奖不是死后颁发的。

沃森和克里克提出,DNA由两条链组成,它们相互扭曲形成右手螺旋线。 碱基配对发生在嘌呤和嘧啶之间;也就是说,带有T和G对的A对与C. 腺嘌呤和胸腺嘧啶是互补的碱基对,胞嘧啶和鸟嘌呤也是互补的碱基对。 碱基对通过氢键稳定;腺嘌呤和胸腺嘧啶形成两个氢键,胞嘧啶和鸟嘌呤形成三个氢键。 这两股线本质上是反平行的;也就是说,一条股线的 3' 末端面对另一条股线的 5' 末端。 核苷酸中的糖和磷酸盐构成结构的支柱,而含氮碱则堆叠在里面。 每个碱基对与另一个碱基对相隔 0.34 nm 的距离,螺旋线每圈长 3.4 nm。 因此,每回合螺旋有十个碱基对。 DNA 双螺旋的直径为 2 nm,整个直径均匀。 只有嘌呤和嘧啶的配对才能解释均匀的直径。 两股线相互扭曲导致形成间隔均匀的主槽和小槽(图\(\PageIndex{3}\))。
DNA 测序技术
直到20世纪90年代,DNA测序(读取DNA序列)是一个相对昂贵且漫长的过程。 出于安全考虑,使用放射性标记的核苷酸也使问题更加复杂。 凭借目前可用的技术和自动化机器,该过程既便宜,又更安全,并且可以在几个小时内完成。 弗雷德·桑格开发了用于人类基因组测序项目的测序方法,该方法现已广泛使用(图\(\PageIndex{4}\))。
该方法被称为二脱氧链终止法。 测序方法基于链终止剂,即二脱氧核苷酸(ddNTP)的使用。 二脱氧核苷酸或 ddntpss 与脱氧核苷酸的不同之处在于五碳糖上缺少游离的 3' OH 基团。 如果将 ddnTP 添加到不断增长的 DNTP 链中,则该链不会进一步延伸,因为添加另一个核苷酸所需的游离 3' OH 基团不可用。 通过使用预先确定的脱氧核糖核苷酸与双脱氧核苷酸的比例,可以生成不同大小的DNA片段。
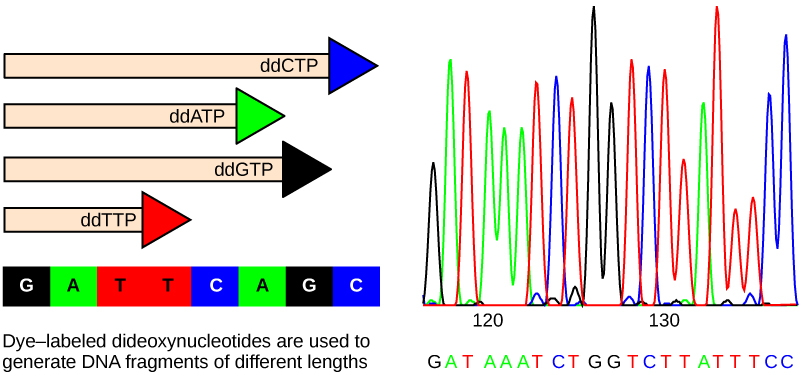
将要测序的DNA样本变性或通过加热至高温分离成两条链。 DNA 分为四个试管,其中添加了引物、DNA 聚合酶和所有四个核苷酸(A、T、G 和 C)。 除了四根试管中的每根外,还向每根试管中分别添加有限数量的四种二脱氧核苷酸中的一种。 根据添加的 ddnTP,这些试管被标记为 A、T、G 和 C。 出于检测目的,四种二脱氧核苷酸中的每一种都带有不同的荧光标签。 链条伸长一直持续到掺入荧光二脱氧核苷酸为止,之后不会发生进一步的伸长。 反应结束后,进行电泳。 甚至可以检测到单个底座的长度差异。 序列是从激光扫描仪读取的。 由于他在DNA测序方面的工作,桑格于1980年获得了诺贝尔化学奖。
链接到学习
桑格的基因组测序引发了一场以快速、低成本对人类基因组进行测序的竞赛,通常被称为一日1000美元的序列。 在此处选择 “快速排序” 动画,了解更多信息。
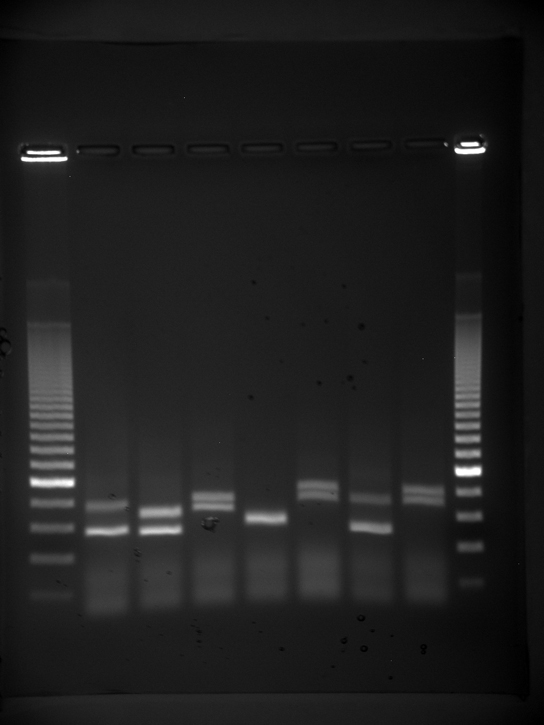
凝胶电泳是一种用于分离不同大小的 DNA 片段的技术。 通常,凝胶是由一种叫做琼脂糖的化学物质制成的。 将琼脂糖粉加入缓冲液中并加热。 冷却后,将凝胶溶液倒入铸造托盘中。 凝胶凝固后,DNA就会加载到凝胶上并施加电流。 DNA 具有净负电荷,从负电极向正电极移动。 施加电流的时间足以让DNA根据大小分离;最小的片段将离井(装载DNA的地方)最远,而分子量较重的片段将离井最近。 DNA 分离后,凝胶会用 DNA 特异性染料进行染色以供观察(图\(\PageIndex{5}\))。
进化连接:尼安德特人基因组——我们之间有何关系?
尼安德特人基因组序列的初稿最近由理查德·格林等人于 2010 年发布。 1 尼安德特人是当今人类最亲密的祖先。 众所周知,他们在大约3万年前从化石记录中消失之前曾生活在欧洲和西亚。 格林的团队研究了从世界各地选出的具有近4万年历史的化石遗迹。 由于骨骼的脆弱性和严重的微生物污染,采用了极其复杂的样本制备和DNA测序方法。 在他们的研究中,科学家们能够对大约40亿个碱基对进行测序。 将尼安德特人的序列与当今来自世界各地的人类序列进行了比较。 在比较了这些序列之后,研究人员发现,尼安德特人基因组与生活在非洲以外的人的相似度比与非洲人的相似度高2%至3%。 尽管当前的理论表明,当今所有人类都可以追溯到非洲的一小部分祖先种群,但来自尼安德特人基因组的数据可能与这种观点相矛盾。 格林和他的同事们还在欧洲和亚洲的人群中发现了与尼安德特人序列比其他当代人类序列更相似的DNA片段。 另一个有趣的观察结果是,尼安德特人与来自巴布亚新几内亚的人和来自中国或法国的人的关系一样密切。 这令人惊讶,因为尼安德特人的化石遗迹仅存在于欧洲和西亚。 最有可能的是,在欧洲人、东亚人和巴布亚新几内亚人出现分歧之前,随着现代人类从非洲崛起,尼安德特人和现代人类之间发生了基因交换。
在当今人类进化过程中,尼安德特人的几个基因似乎发生了变化。 这些基因参与颅骨结构、新陈代谢、皮肤形态和认知发育。 特别令人感兴趣的基因之一是 RUNX2,它在现代人类和尼安德特人中有所不同。 该基因是尼安德特人突出的额骨、钟形肋骨和牙齿差异的原因。 据推测,RUNX2 的进化变化在现代人类的起源中很重要,这影响了颅骨和上半身。
细胞中的 DNA 封装
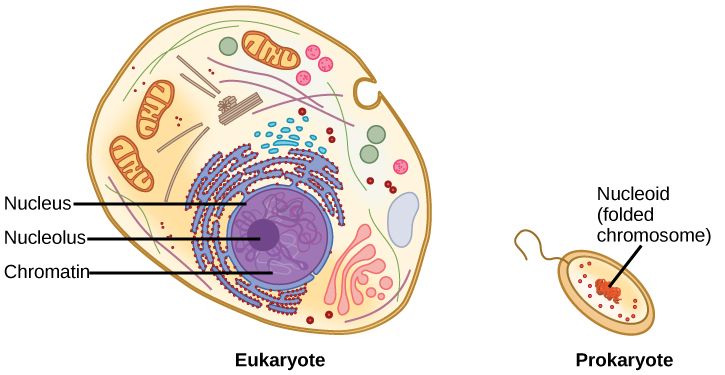
将原核细胞与真核细胞进行比较时,原核生物在许多特征上比真核细胞简单得多(图\(\PageIndex{6}\))。 大多数原核生物都含有单个圆形染色体,该染色体存在于细胞质的一个称为核样体的区域中。
练习\(\PageIndex{1}\)
在真核细胞中,DNA和RNA的合成与蛋白质合成发生在不同的隔室中。 在原核细胞中,这两个过程一起发生。 将流程分开会有什么好处? 将它们一起出现会有什么好处?
- 回答
-
分隔使真核细胞能够将过程分成离散的步骤,从而生成更复杂的蛋白质和RNA产物。 但是单隔室也有一个好处:RNA和蛋白质合成在原核细胞中的发生速度要快得多。
研究最充分的原核生物之一大肠杆菌的基因组大小为460万个碱基对(如果切开并拉伸则约为1.1毫米)。 那么,它如何适合小型细菌细胞呢? DNA被所谓的超级线圈所扭曲。 Supercoiling 意味着 DNA 要么处于下绕状态(每 10 个碱基对的螺旋线少于一圈),要么从其正常的松弛状态中超过 1 圈(每 10 个碱基对超过 1 圈)。 已知有些蛋白质与超级线圈有关;其他蛋白质和酶,例如DNA回旋酶,有助于维持超线圈结构。
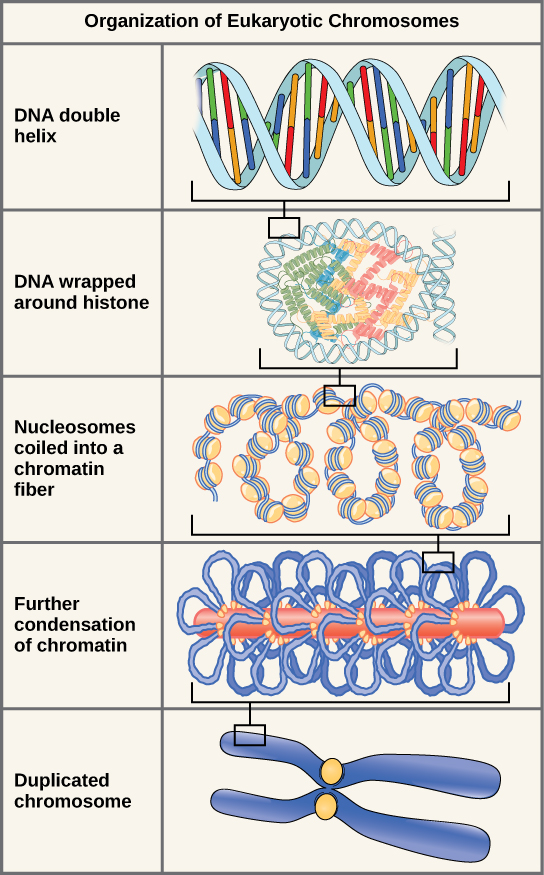
真核生物的染色体均由线性 DNA 分子组成,它们采用不同类型的包装策略将其 DNA 装入细胞核(图\(\PageIndex{7}\))。 在最基本的层面上,DNA被称为组蛋白的蛋白质包裹,形成称为核小体的结构。 组蛋白是进化保守的蛋白质,富含碱性氨基酸并形成八聚体。 DNA(由于磷酸基团而带负电荷)紧紧地包裹在组蛋白核心周围。 这个核小体在连接器 DNA 的帮助下与下一个核小体相连。 这也被称为 “绳子上的珠子” 结构。 它被进一步压实成一个 30 nm 的纤维,这是结构的直径。 在中期阶段,染色体最紧凑,宽度约为700 nm,并且与支架蛋白有关。
在相间,真核生物染色体有两个不同的区域,可以通过染色来区分。 紧密封装的区域被称为异染色质,密度较低的区域被称为真染色质。 Heterochromatin 通常含有未表达的基因,存在于着丝粒和端粒区域。 euchromatin 通常含有经过转录的基因,DNA 封装在核小体周围,但不会进一步压实。
摘要
目前公认的DNA双螺旋结构模型是由沃森和克里克提出的。 一些显著特征是,构成双螺旋的两条线本质上是互补的,是反平行的。 脱氧核糖和磷酸盐构成结构的支柱,含氮碱堆叠在里面。 双螺旋的直径为 2 nm,在整个过程中均匀。 嘌呤总是与嘧啶配对;A 对与 T 配对,G 对与 C 配对。一圈螺旋有十个碱基对。 在细胞分裂过程中,每个子细胞通过称为DNA复制的过程获得一份DNA副本。 原核生物在许多特征上比真核生物简单得多。 大多数原核生物都含有单个圆形染色体。 通常,真核生物染色体包含封装在核小体中的线性 DNA 分子,并且有两个不同的区域,可以通过染色来区分,反映了不同的包装和压实状态。
脚注
- 1 Richard E. Green 等人,《尼安德特人基因组序列草稿》,《科学 328》(2010):710-22。
词汇表
- 电泳
- 用于根据大小分离 DNA 片段的技术




