15.1: O Código Genético
- Page ID
- 182293
Habilidades para desenvolver
- Explique o “dogma central” da síntese protéica
- Descreva o código genético e como a sequência de nucleotídeos prescreve a sequência de aminoácidos e proteínas
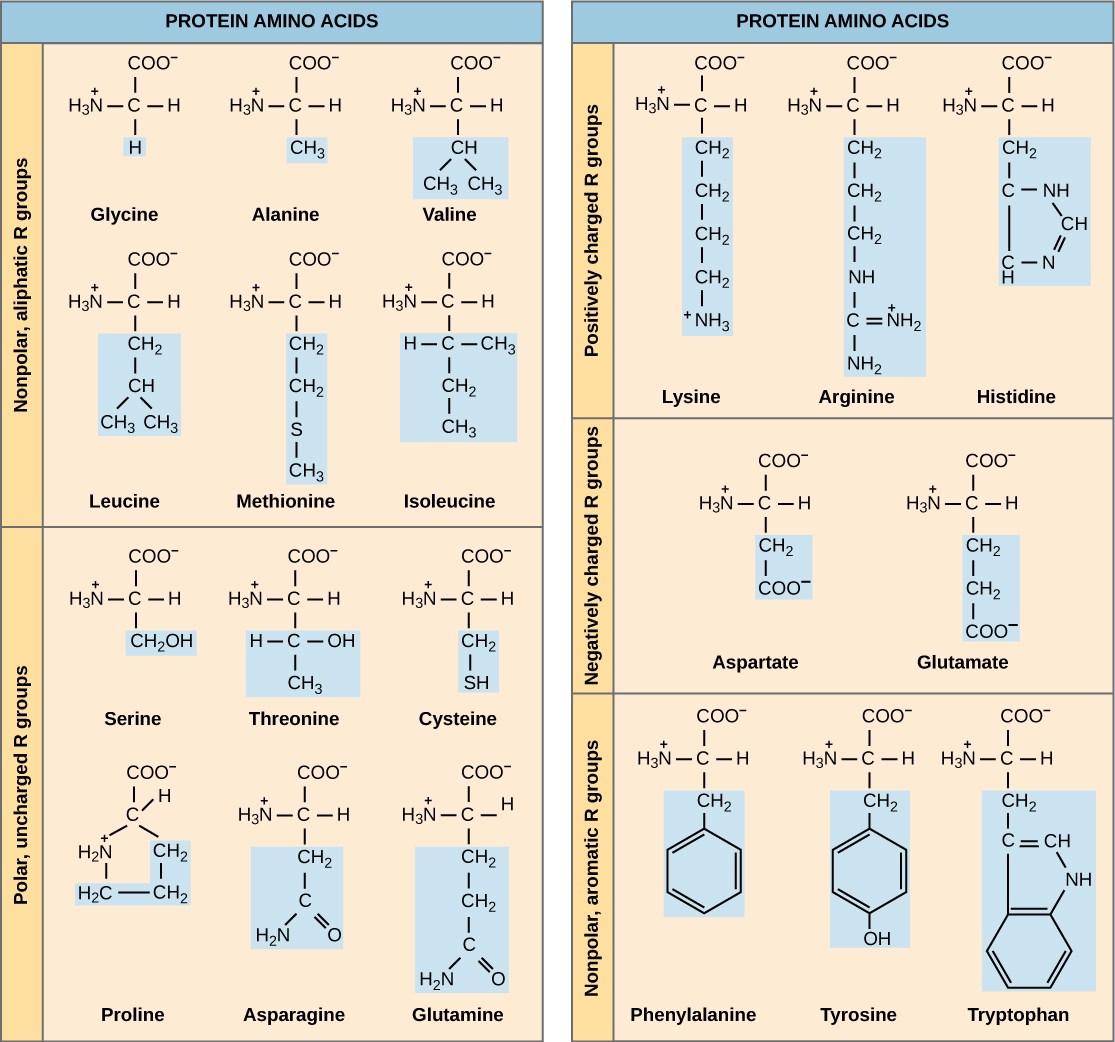
O processo celular de transcrição gera RNA mensageiro (mRNA), uma cópia molecular móvel de um ou mais genes com um alfabeto de A, C, G e uracil (U). A tradução do modelo de mRNA converte informações genéticas baseadas em nucleotídeos em um produto proteico. As sequências de proteínas consistem em 20 aminoácidos comuns; portanto, pode-se dizer que o alfabeto proteico consiste em 20 letras (Figura\(\PageIndex{1}\)). Cada aminoácido é definido por uma sequência de três nucleotídeos chamada códon trigêmeo. Aminoácidos diferentes têm diferentes químicas (como ácido versus básico, ou polar e não polar) e diferentes restrições estruturais. A variação na sequência de aminoácidos dá origem a uma enorme variação na estrutura e função das proteínas.
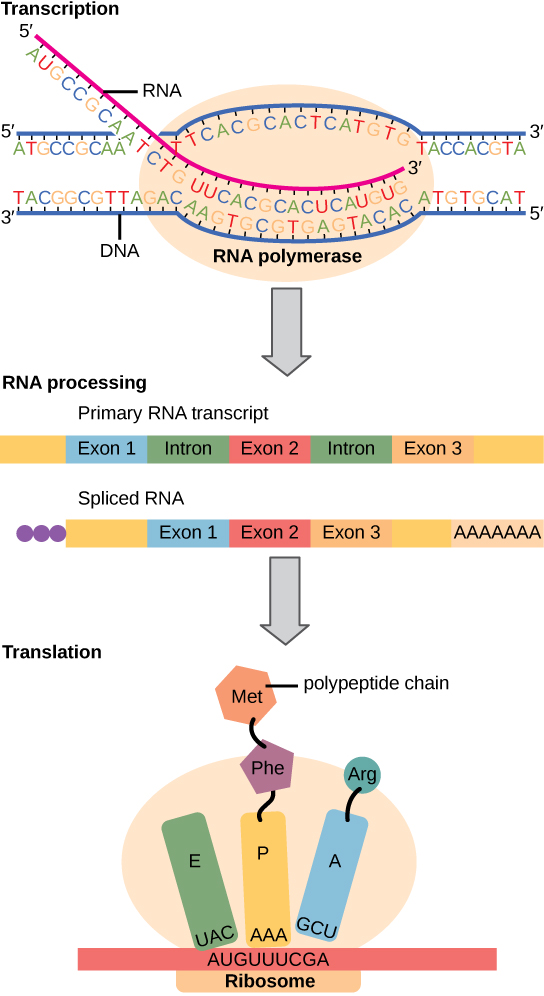
O dogma central: o DNA codifica o RNA; o RNA codifica a proteína
O fluxo de informação genética nas células do DNA ao mRNA e à proteína é descrito pelo Dogma Central (Figura\(\PageIndex{2}\)), que afirma que os genes especificam a sequência de mRNAs, que por sua vez especificam a sequência das proteínas. A decodificação de uma molécula para outra é realizada por proteínas e RNAs específicos. Como as informações armazenadas no DNA são tão centrais para a função celular, faz sentido intuitivo que a célula faça cópias de mRNA dessas informações para síntese de proteínas, mantendo o próprio DNA intacto e protegido. A cópia do DNA para o RNA é relativamente simples, com um nucleotídeo sendo adicionado à fita de mRNA para cada nucleotídeo lido na fita de DNA. A tradução para proteína é um pouco mais complexa porque três nucleotídeos de mRNA correspondem a um aminoácido na sequência polipeptídica. No entanto, a tradução para proteína ainda é sistemática e colinear, de forma que os nucleotídeos 1 a 3 correspondem ao aminoácido 1, os nucleotídeos 4 a 6 correspondem ao aminoácido 2 e assim por diante.
O código genético é degenerado e universal
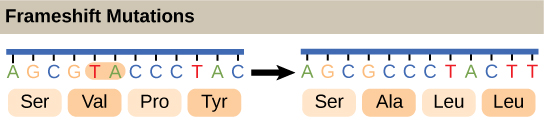
Dados os diferentes números de “letras” nos “alfabetos” de mRNA e proteínas, os cientistas teorizaram que as combinações de nucleotídeos correspondiam a aminoácidos únicos. Dupletos de nucleotídeos não seriam suficientes para especificar cada aminoácido porque existem apenas 16 combinações possíveis de dois nucleotídeos (4 2). Em contraste, existem 64 possíveis trigêmeos de nucleotídeos (4 3), o que é muito mais do que o número de aminoácidos. Os cientistas teorizaram que os aminoácidos eram codificados por trigêmeos de nucleotídeos e que o código genético era degenerado. Em outras palavras, um determinado aminoácido pode ser codificado por mais de um trigêmeo nucleotídico. Posteriormente, isso foi confirmado experimentalmente; Francis Crick e Sydney Brenner usaram a proflavina mutagênica química para inserir um, dois ou três nucleotídeos no gene de um vírus. Quando um ou dois nucleotídeos foram inseridos, a síntese protéica foi completamente abolida. Quando três nucleotídeos foram inseridos, a proteína foi sintetizada e funcional. Isso demonstrou que três nucleotídeos especificam cada aminoácido. Esses trigêmeos de nucleotídeos são chamados de códons. A inserção de um ou dois nucleotídeos alterou completamente o quadro de leitura do trigêmeo, alterando assim a mensagem para cada aminoácido subsequente (Figura\(\PageIndex{4}\)). Embora a inserção de três nucleotídeos tenha causado a inserção de um aminoácido extra durante a tradução, a integridade do resto da proteína foi mantida.
Os cientistas resolveram meticulosamente o código genético traduzindo mRNAs sintéticos in vitro e sequenciando as proteínas que eles especificaram (Figura\(\PageIndex{3}\)).
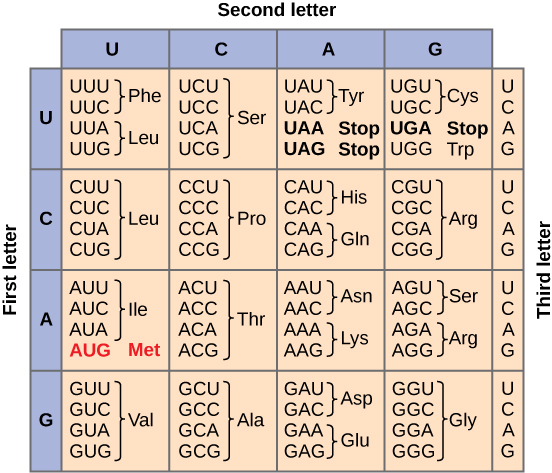
Além de instruir a adição de um aminoácido específico a uma cadeia polipeptídica, três dos 64 códons encerram a síntese protéica e liberam o polipeptídeo da máquina de tradução. Esses trigêmeos são chamados de códons sem sentido, ou códons de parada. Outro códon, AUG, também tem uma função especial. Além de especificar o aminoácido metionina, ele também serve como códon inicial para iniciar a tradução. O quadro de leitura para tradução é definido pelo códon inicial do AUG próximo à extremidade 5' do mRNA.
O código genético é universal. Com algumas exceções, praticamente todas as espécies usam o mesmo código genético para a síntese de proteínas. A conservação dos códons significa que um mRNA purificado que codifica a proteína globina em cavalos poderia ser transferido para uma célula de tulipa, e a tulipa sintetizaria a globina de cavalo. O fato de haver apenas um código genético é uma evidência poderosa de que toda a vida na Terra compartilha uma origem comum, especialmente considerando que existem cerca de 10.84 combinações possíveis de 20 aminoácidos e 64 códons trigêmeos.
Link para o aprendizado

Transcreva um gene e traduza em proteína usando o emparelhamento complementar e o código genético neste local.
Acredita-se que a degeneração seja um mecanismo celular para reduzir o impacto negativo de mutações aleatórias. Os códons que especificam o mesmo aminoácido normalmente diferem apenas em um nucleotídeo. Além disso, aminoácidos com cadeias laterais quimicamente similares são codificados por códons similares. Essa nuance do código genético garante que uma mutação de substituição de nucleotídeo único possa especificar o mesmo aminoácido, mas não ter efeito, ou especificar um aminoácido similar, impedindo que a proteína se torne completamente não funcional.
Conexão de método científico: o que tem mais DNA: um kiwi ou um morango?

Pergunta: Um kiwi e um morango com aproximadamente o mesmo tamanho (Figura\(\PageIndex{5}\)) também teriam aproximadamente a mesma quantidade de DNA?
Antecedentes: Os genes são transportados nos cromossomos e são feitos de DNA. Todos os mamíferos são diplóides, o que significa que eles têm duas cópias de cada cromossomo. No entanto, nem todas as plantas são diplóides. O morango comum é octoplóide (8 n) e o kiwi cultivado é hexaplóide (6 n). Pesquise o número total de cromossomos nas células de cada uma dessas frutas e pense em como isso pode corresponder à quantidade de DNA nos núcleos celulares dessas frutas. Leia sobre a técnica de isolamento de DNA para entender como cada etapa do protocolo de isolamento ajuda a liberar e precipitar o DNA.
Hipótese: Imagine se você seria capaz de detectar uma diferença na quantidade de DNA de morangos e kiwis de tamanhos semelhantes. Qual fruta você acha que produziria mais DNA?
Teste sua hipótese: isole o DNA de um morango e um kiwi de tamanho semelhante. Realize o experimento em pelo menos triplicado para cada fruta.
- Prepare um frasco de tampão de extração de DNA com 900 mL de água, 50 mL de detergente de louça e duas colheres de chá de sal de cozinha. Misture por inversão (tampe e vire de cabeça para baixo algumas vezes).
- Moa um morango e um kiwi manualmente em um saco plástico, ou usando um almofariz e pilão, ou com uma tigela de metal e a ponta de um instrumento sem corte. Moa por pelo menos dois minutos por fruta.
- Adicione 10 mL do tampão de extração de DNA a cada fruta e misture bem por pelo menos um minuto.
- Remova os detritos celulares filtrando cada mistura de frutas com gaze ou pano poroso e em um funil colocado em um tubo de ensaio ou recipiente apropriado.
- Despeje etanol gelado ou isopropanol (álcool isopropílico) no tubo de ensaio. Você deve observar o DNA branco precipitado.
- Reúna o DNA de cada fruta enrolando-o em torno de varetas de vidro separadas.
Registre suas observações: Como você não está medindo quantitativamente o volume de DNA, você pode registrar para cada ensaio se as duas frutas produziram quantidades iguais ou diferentes de DNA observadas a olho nu. Se uma ou outra fruta produziu visivelmente mais DNA, registre isso também. Determine se suas observações são consistentes com vários pedaços de cada fruta.
Analise seus dados: Você notou uma diferença óbvia na quantidade de DNA produzida por cada fruta? Seus resultados foram reproduzíveis?
Faça uma conclusão: Considerando o que você sabe sobre o número de cromossomos em cada fruta, você pode concluir que o número de cromossomos necessariamente se correlaciona com a quantidade de DNA? Você consegue identificar alguma desvantagem desse procedimento? Se você tivesse acesso a um laboratório, como você poderia padronizar sua comparação e torná-la mais quantitativa?
Resumo
O código genético se refere ao alfabeto de DNA (A, T, C, G), ao alfabeto de RNA (A, U, C, G) e ao alfabeto polipeptídico (20 aminoácidos). O Dogma Central descreve o fluxo de informação genética na célula, dos genes ao mRNA e às proteínas. Os genes são usados para produzir mRNA pelo processo de transcrição; o mRNA é usado para sintetizar proteínas pelo processo de tradução. O código genético é degenerado porque 64 códons trigêmeos no mRNA especificam apenas 20 aminoácidos e três códons sem sentido. Quase todas as espécies do planeta usam o mesmo código genético.
Glossário
- Dogma central
- afirma que os genes especificam a sequência de mRNAs, que por sua vez especificam a sequência de proteínas
- códon
- três nucleotídeos consecutivos no mRNA que especificam a inserção de um aminoácido ou a liberação de uma cadeia polipeptídica durante a tradução
- colinear
- em termos de RNA e proteína, três “unidades” de RNA (nucleotídeos) especificam uma “unidade” de proteína (aminoácido) de forma consecutiva
- degeneração
- (do código genético) descreve que um determinado aminoácido pode ser codificado por mais de um trigêmeo de nucleotídeo; o código é degenerado, mas não ambíguo
- códon sem sentido
- um dos três códons de mRNA que especifica o término da tradução
- quadro de leitura
- sequência de códons trigêmeos no mRNA que especificam uma proteína específica; uma mudança no ribossomo de um ou dois nucleotídeos em qualquer direção abole completamente a síntese dessa proteína


