14.4: Replicação de DNA em procariontes
- Page ID
- 182260
Habilidades para desenvolver
- Explicar o processo de replicação do DNA em procariontes
- Discuta o papel de diferentes enzimas e proteínas no apoio a esse processo
A replicação do DNA tem sido extremamente bem estudada em procariontes, principalmente por causa do pequeno tamanho do genoma e dos mutantes disponíveis. A E. coli tem 4,6 milhões de pares de bases em um único cromossomo circular e tudo isso é replicado em aproximadamente 42 minutos, partindo de uma única origem de replicação e prosseguindo ao redor do círculo em ambas as direções. Isso significa que aproximadamente 1000 nucleotídeos são adicionados por segundo. O processo é bastante rápido e ocorre sem muitos erros.
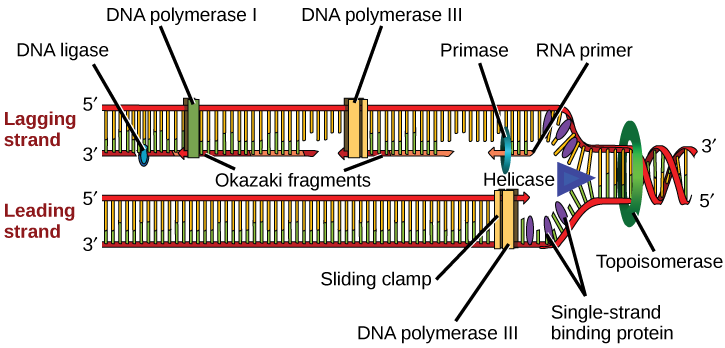
A replicação do DNA emprega um grande número de proteínas e enzimas, cada uma das quais desempenha um papel fundamental durante o processo. Um dos principais participantes é a enzima DNA polimerase, também conhecida como DNA pol, que adiciona nucleotídeos um a um à crescente cadeia de DNA que são complementares à fita modelo. A adição de nucleotídeos requer energia; essa energia é obtida dos nucleotídeos que têm três fosfatos ligados a eles, semelhante ao ATP, que tem três grupos fosfato ligados. Quando a ligação entre os fosfatos é quebrada, a energia liberada é usada para formar a ligação fosfodiéster entre o nucleotídeo de entrada e a cadeia de crescimento. Em procariontes, três tipos principais de polimerases são conhecidos: DNA pol I, DNA pol II e DNA pol III. Sabe-se agora que o DNA pol III é a enzima necessária para a síntese de DNA; DNA pol I e DNA pol II são necessários principalmente para reparo.
Como a máquina de replicação sabe por onde começar? Acontece que existem sequências específicas de nucleotídeos chamadas origens da replicação, onde a replicação começa. Em E. coli, que tem uma única origem de replicação em seu único cromossomo (assim como a maioria dos procariontes), tem aproximadamente 245 pares de bases e é rica em sequências AT. A origem da replicação é reconhecida por certas proteínas que se ligam a esse local. Uma enzima chamada helicase desenrola o DNA quebrando as ligações de hidrogênio entre os pares de bases nitrogenadas. A hidrólise de ATP é necessária para esse processo. À medida que o DNA se abre, estruturas em forma de Y chamadas garfos de replicação são formadas. Duas bifurcações de replicação são formadas na origem da replicação e elas são estendidas bidirecionalmente à medida que a replicação prossegue. As proteínas de ligação de fita única revestem as fitas simples de DNA próximas ao garfo de replicação para evitar que o DNA de fita simples volte para uma dupla hélice. A DNA polimerase é capaz de adicionar nucleotídeos somente na direção 5' a 3' (uma nova fita de DNA só pode ser estendida nessa direção). Também requer um grupo 3'-OH livre ao qual possa adicionar nucleotídeos formando uma ligação fosfodiéster entre a extremidade 3'-OH e o fosfato 5' do próximo nucleotídeo. Isso significa essencialmente que ele não pode adicionar nucleotídeos se um grupo 3'-OH livre não estiver disponível. Então, como ele adiciona o primeiro nucleotídeo? O problema é resolvido com a ajuda de um primer que fornece a extremidade 3'-OH gratuita. Outra enzima, a RNA primase, sintetiza um primer de RNA com cerca de cinco a dez nucleotídeos de comprimento e complementar ao DNA. Como essa sequência prepara a síntese de DNA, ela é apropriadamente chamada de primer. A DNA polimerase agora pode estender esse primer de RNA, adicionando nucleotídeos um a um que são complementares à fita modelo (Figura\(\PageIndex{1}\)).
Exercício\(\PageIndex{1}\)
Você isola uma cepa celular na qual a união de fragmentos de Okazaki está comprometida e suspeita que uma mutação tenha ocorrido em uma enzima encontrada na bifurcação de replicação. Qual enzima tem maior probabilidade de sofrer mutação?
- Resposta
-
DNA ligase, à medida que essa enzima une fragmentos de Okazaki.
O garfo de replicação se move à taxa de 1000 nucleotídeos por segundo. A DNA polimerase só pode se estender na direção de 5' a 3', o que representa um pequeno problema na bifurcação de replicação. Como sabemos, a dupla hélice do DNA é anti-paralela; ou seja, uma fita está na direção 5' a 3' e a outra é orientada na direção 3' a 5'. Uma fita, que é complementar à fita de DNA parental de 3' a 5', é sintetizada continuamente em direção ao garfo de replicação porque a polimerase pode adicionar nucleotídeos nessa direção. Essa fita sintetizada continuamente é conhecida como a fita principal. A outra fita, complementar ao DNA parental de 5' a 3', é estendida para longe do garfo de replicação, em pequenos fragmentos conhecidos como fragmentos de Okazaki, cada um exigindo um primer para iniciar a síntese. Os fragmentos de Okazaki têm o nome do cientista japonês que os descobriu pela primeira vez. O fio com os fragmentos de Okazaki é conhecido como fio defasado.
O fio principal pode ser estendido apenas com um primer, enquanto o fio atrasado precisa de um novo primer para cada um dos fragmentos curtos de Okazaki. A direção geral do fio defasado será de 3' a 5', e a do fio principal de 5' a 3'. Uma proteína chamada pinça deslizante mantém a DNA polimerase no lugar enquanto continua adicionando nucleotídeos. A pinça deslizante é uma proteína em forma de anel que se liga ao DNA e mantém a polimerase no lugar. A topoisomerase evita o enrolamento excessivo da dupla hélice do DNA à frente do garfo de replicação quando o DNA se abre; ela faz isso causando cortes temporários na hélice do DNA e, em seguida, selando-a novamente. Conforme a síntese prossegue, os primers de RNA são substituídos pelo DNA. Os primers são removidos pela atividade da exonuclease do DNA pol I, e as lacunas são preenchidas por desoxirribonucleotídeos. Os cortes que permanecem entre o DNA recém-sintetizado (que substituiu o primer de RNA) e o DNA sintetizado anteriormente são selados pela enzima DNA ligase que catalisa a formação da ligação fosfodiéster entre a extremidade 3'-OH de um nucleotídeo e a extremidade 5' de fosfato do outro fragmento.
Depois que o cromossomo é completamente replicado, as duas cópias de DNA se movem para duas células diferentes durante a divisão celular. O processo de replicação do DNA pode ser resumido da seguinte forma.
etapas de replicação do DNA
- O DNA se desenrola na origem da replicação.
- O Helicase abre os garfos de replicação formadores de DNA; eles são estendidos bidirecionalmente.
- As proteínas de ligação de fita única revestem o DNA ao redor do garfo de replicação para evitar o rebobinamento do DNA.
- A topoisomerase se liga na região à frente da bifurcação de replicação para evitar o superenrolamento.
- A Primase sintetiza primers de RNA complementares à fita de DNA.
- A DNA polimerase começa a adicionar nucleotídeos à extremidade 3'-OH do primer.
- O alongamento do fio atrasado e do fio dianteiro continua.
- Os primers de RNA são removidos pela atividade da exonuclease.
- As lacunas são preenchidas pelo DNA pol adicionando DNTPs.
- A lacuna entre os dois fragmentos de DNA é selada pela DNA ligase, que ajuda na formação de ligações fosfodiéster.
A tabela\(\PageIndex{1}\) resume as enzimas envolvidas na replicação do DNA procariótico e as funções de cada uma.
| Enzima/proteína | Função específica |
|---|---|
| Pesquisa de DNA I | A atividade da exonuclease remove o primer de RNA e substitui pelo DNA recém-sintetizado |
| Pesquisa de DNA II | Função de reparo |
| Pesquisa de DNA III | Enzima principal que adiciona nucleotídeos na direção 5'-3' |
| Helicase | Abre a hélice do DNA quebrando as ligações de hidrogênio entre as bases nitrogenadas |
| Ligase | Sela as lacunas entre os fragmentos de Okazaki para criar uma fita contínua de DNA |
| Primase | Sintetiza os primers de RNA necessários para iniciar a replicação |
| Braçadeira deslizante | Ajuda a manter a DNA polimerase no lugar quando os nucleotídeos estão sendo adicionados |
| Topoisomerase | Ajuda a aliviar o estresse no DNA ao se desenrolar, causando quebras e, em seguida, selando novamente o DNA |
| Proteínas de ligação de fita única (SSB) | Liga-se ao DNA de fita simples para evitar que o DNA se recupere. |
Link para o aprendizado
Revise o processo completo de replicação do DNA aqui.
Resumo
A replicação em procariontes começa a partir de uma sequência encontrada no cromossomo chamada origem da replicação - o ponto em que o DNA se abre. A helicase abre a dupla hélice do DNA, resultando na formação do garfo de replicação. As proteínas de ligação de fita única se ligam ao DNA de fita simples próximo ao garfo de replicação para manter o garfo aberto. A Primase sintetiza um primer de RNA para iniciar a síntese pela DNA polimerase, que pode adicionar nucleotídeos somente na direção 5' a 3'. Um fio é sintetizado continuamente na direção do garfo de replicação; isso é chamado de fio principal. A outra fita é sintetizada em uma direção distante da bifurcação de replicação, em pequenos trechos de DNA conhecidos como fragmentos de Okazaki. Esse fio é conhecido como fio atrasado. Uma vez concluída a replicação, os primers de RNA são substituídos por nucleotídeos de DNA e o DNA é selado com DNA ligase, que cria ligações de fosfodiéster entre o 3'-OH de uma extremidade e o 5' fosfato da outra fita.
Glossário
- helicase
- durante a replicação, essa enzima ajuda a abrir a hélice do DNA quebrando as ligações de hidrogênio
- fio atrasado
- durante a replicação, a fita que é replicada em fragmentos curtos e longe da bifurcação de replicação
- vertente principal
- fio que é sintetizado continuamente na direção 5'-3' que é sintetizado na direção do garfo de replicação
- ligase
- enzima que catalisa a formação de uma ligação fosfodiéster entre as extremidades 3' OH e 5' fosfato do DNA
- Fragmento de Okazaki
- Fragmento de DNA que é sintetizado em trechos curtos na fita defasada
- primase
- enzima que sintetiza o primer de RNA; o primer é necessário para que o DNA pol inicie a síntese de uma nova fita de DNA
- primer
- pequeno trecho de nucleotídeos que é necessário para iniciar a replicação; no caso da replicação, o primer tem nucleotídeos de RNA
- garfo de replicação
- Estrutura em forma de Y formada durante o início da replicação
- proteína de ligação de fita única
- durante a replicação, proteína que se liga ao DNA de fita simples; isso ajuda a manter as duas fitas de DNA separadas para que possam servir como modelos
- braçadeira deslizante
- Proteína em forma de anel que retém o pólo de DNA na fita de DNA
- topoisomerase
- enzima que causa o enrolamento ou o enrolamento excessivo do DNA quando a replicação do DNA está ocorrendo


