14.2: Estrutura e sequenciamento do DNA
- Page ID
- 182276
Habilidades para desenvolver
- Descreva a estrutura do DNA
- Explicar o método Sanger de sequenciamento de DNA
- Discuta as semelhanças e diferenças entre DNA eucariótico e procariótico
Os blocos de construção do DNA são os nucleotídeos. Os componentes importantes do nucleotídeo são uma base nitrogenada, a desoxirribose (açúcar de 5 carbonos) e um grupo fosfato (Figura\(\PageIndex{1}\)). O nucleotídeo é nomeado dependendo da base nitrogenada. A base nitrogenada pode ser uma purina, como adenina (A) e guanina (G), ou uma pirimidina, como citosina (C) e timina (T).
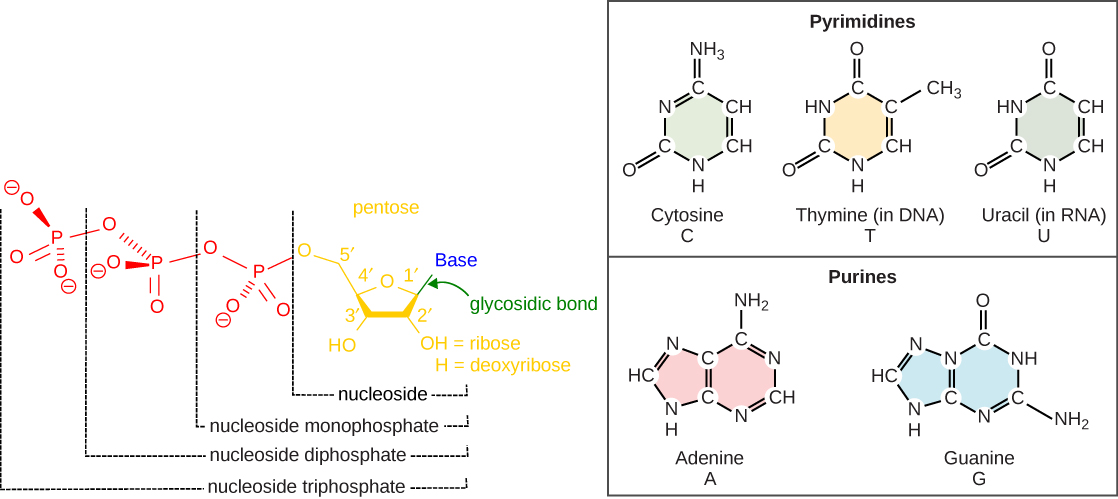
Os nucleotídeos se combinam entre si por ligações covalentes conhecidas como ligações ou ligações fosfodiéster. As purinas têm uma estrutura de anel duplo com um anel de seis membros fundido a um anel de cinco membros. As pirimidinas são menores em tamanho; elas têm uma única estrutura em anel de seis membros. Os átomos de carbono do açúcar de cinco carbonos são numerados 1', 2', 3', 4' e 5' (1' é lido como “um primo”). O resíduo de fosfato está ligado ao grupo hidroxila do carbono 5' de um açúcar de um nucleotídeo e ao grupo hidroxila do carbono 3' do açúcar do próximo nucleotídeo, formando assim uma ligação fosfodiéster 5'-3'.
Na década de 1950, Francis Crick e James Watson trabalharam juntos para determinar a estrutura do DNA na Universidade de Cambridge, Inglaterra. Outros cientistas como Linus Pauling e Maurice Wilkins também estavam explorando ativamente esse campo. Pauling descobriu a estrutura secundária das proteínas usando a cristalografia de raios-X. No laboratório de Wilkins, a pesquisadora Rosalind Franklin estava usando métodos de difração de raios-X para entender a estrutura do DNA. Watson e Crick conseguiram montar o quebra-cabeça da molécula de DNA com base nos dados de Franklin porque Crick também havia estudado a difração de raios-X (Figura\(\PageIndex{2}\)). Em 1962, James Watson, Francis Crick e Maurice Wilkins receberam o Prêmio Nobel de Medicina. Infelizmente, até então Franklin havia morrido e os prêmios Nobel não foram concedidos postumamente.

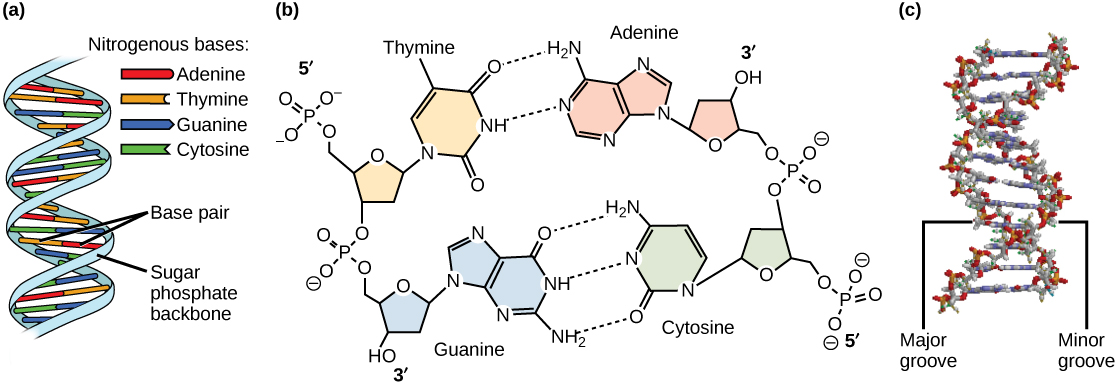
Watson e Crick propuseram que o DNA é feito de duas fitas que são torcidas uma em volta da outra para formar uma hélice destra. O emparelhamento de bases ocorre entre uma purina e pirimidina; ou seja, pares A com T e G pares com C. Adenina e timina são pares de bases complementares, e citosina e guanina também são pares de bases complementares. Os pares de bases são estabilizados por ligações de hidrogênio; adenina e timina formam duas ligações de hidrogênio e citosina e guanina formam três ligações de hidrogênio. Os dois fios são de natureza anti-paralela; ou seja, a extremidade de 3 pés de um fio está voltada para a extremidade 5 pés do outro fio. O açúcar e o fosfato dos nucleotídeos formam a espinha dorsal da estrutura, enquanto as bases nitrogenadas são empilhadas em seu interior. Cada par de bases é separado do outro par de bases por uma distância de 0,34 nm, e cada volta da hélice mede 3,4 nm. Portanto, dez pares de bases estão presentes por volta da hélice. O diâmetro da dupla hélice do DNA é de 2 nm e é uniforme por toda parte. Somente o emparelhamento entre uma purina e uma pirimidina pode explicar o diâmetro uniforme. A torção dos dois fios em torno um do outro resulta na formação de ranhuras maiores e menores uniformemente espaçadas (Figura\(\PageIndex{3}\)).
Técnicas de Sequenciamento
Até a década de 1990, o sequenciamento do DNA (leitura da sequência do DNA) era um processo relativamente caro e longo. O uso de nucleotídeos radiomarcados também agravou o problema por meio de questões de segurança. Com a tecnologia atualmente disponível e as máquinas automatizadas, o processo é barato, mais seguro e pode ser concluído em questão de horas. Fred Sanger desenvolveu o método de sequenciamento usado para o projeto de sequenciamento do genoma humano, que é amplamente usado atualmente (Figura\(\PageIndex{4}\)).
Link para o aprendizado

Visite este site para assistir a um vídeo explicando a técnica de leitura de sequências de DNA resultante do trabalho de Sanger.
O método é conhecido como método de terminação da cadeia de dideoxi. O método de sequenciamento é baseado no uso de terminadores de cadeia, os didesoxinucleotídeos (DDNTPs). Os didesoxinucleotídeos, ou DDNTPSs, diferem dos desoxinucleotídeos pela falta de um grupo 3' OH livre no açúcar de cinco carbonos. Se um dDNTP for adicionado a uma fita de DNA em crescimento, a cadeia não será mais estendida porque o grupo 3' OH livre necessário para adicionar outro nucleotídeo não está disponível. Usando uma proporção predeterminada de desoxirribonucleotídeos para didesoxinucleotídeos, é possível gerar fragmentos de DNA de tamanhos diferentes.
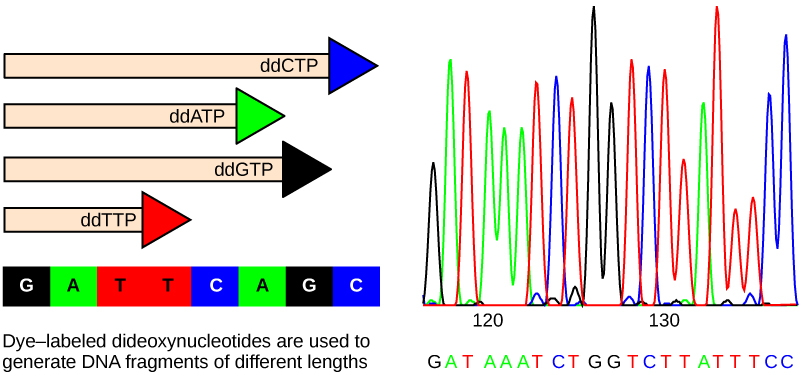
A amostra de DNA a ser sequenciada é desnaturada ou separada em duas fitas por aquecimento a altas temperaturas. O DNA é dividido em quatro tubos nos quais um primer, DNA polimerase e todos os quatro nucleotídeos (A, T, G e C) são adicionados. Além de cada um dos quatro tubos, quantidades limitadas de um dos quatro didesoxinucleotídeos são adicionadas a cada tubo, respectivamente. Os tubos são rotulados como A, T, G e C de acordo com o dDNTP adicionado. Para fins de detecção, cada um dos quatro didesoxinucleotídeos carrega um rótulo fluorescente diferente. O alongamento da cadeia continua até que um nucleotídeo dideoxi fluorescente seja incorporado, após o qual nenhum alongamento adicional ocorre. Após o término da reação, a eletroforese é realizada. Até mesmo uma diferença no comprimento de uma única base pode ser detectada. A sequência é lida em um scanner a laser. Por seu trabalho em sequenciamento de DNA, Sanger recebeu o Prêmio Nobel de Química em 1980.
Link para o aprendizado
O sequenciamento do genoma de Sanger levou a uma corrida para sequenciar genomas humanos em alta velocidade e baixo custo, muitas vezes chamados de $1000 em uma sequência de um dia. Saiba mais selecionando a animação Sequencing at Speed aqui.
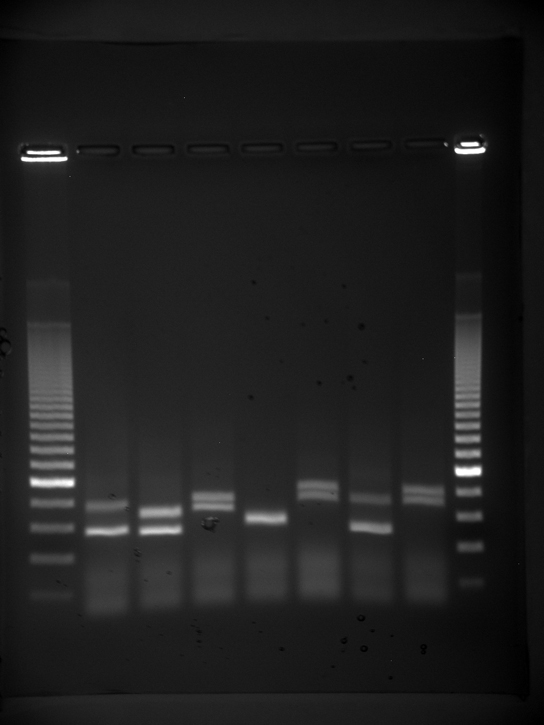
A eletroforese em gel é uma técnica usada para separar fragmentos de DNA de tamanhos diferentes. Normalmente, o gel é feito de uma substância química chamada agarose. O pó de agarose é adicionado a um tampão e aquecido. Após o resfriamento, a solução de gel é despejada em uma bandeja de fundição. Depois que o gel se solidifica, o DNA é carregado no gel e a corrente elétrica é aplicada. O DNA tem uma carga negativa líquida e se move do eletrodo negativo em direção ao eletrodo positivo. A corrente elétrica é aplicada por tempo suficiente para permitir que o DNA se separe de acordo com o tamanho; os fragmentos menores estarão mais distantes do poço (onde o DNA foi carregado) e os fragmentos de maior peso molecular estarão mais próximos do poço. Depois que o DNA é separado, o gel é corado com um corante específico de DNA para visualização (Figura\(\PageIndex{5}\)).
Conexão Evolutiva: Genoma Neandertal - Como estamos relacionados?
O primeiro rascunho da sequência do genoma do Neandertal foi publicado recentemente por Richard E. Green et al. em 2010. 1 Os neandertais são os ancestrais mais próximos dos humanos atuais. Eles eram conhecidos por terem vivido na Europa e na Ásia Ocidental antes de desaparecerem dos registros fósseis há aproximadamente 30.000 anos. A equipe de Green estudou restos fósseis de quase 40.000 anos que foram selecionados de locais em todo o mundo. Meios extremamente sofisticados de preparação de amostras e sequenciamento de DNA foram empregados devido à natureza frágil dos ossos e à forte contaminação microbiana. Em seu estudo, os cientistas conseguiram sequenciar cerca de quatro bilhões de pares de bases. A sequência de Neandertal foi comparada com a dos humanos atuais de todo o mundo. Depois de comparar as sequências, os pesquisadores descobriram que o genoma do Neandertal tinha 2 a 3 por cento maior semelhança com pessoas que viviam fora da África do que com pessoas na África. Embora as teorias atuais tenham sugerido que todos os humanos atuais podem ser atribuídos a uma pequena população ancestral na África, os dados do genoma do Neandertal podem contradizer essa visão. Green e seus colegas também descobriram segmentos de DNA entre pessoas na Europa e na Ásia que são mais semelhantes às sequências de Neandertal do que a outras sequências humanas contemporâneas. Outra observação interessante foi que os neandertais são tão intimamente relacionados com as pessoas de Papua Nova Guiné quanto com as da China ou da França. Isso é surpreendente porque restos fósseis de Neandertal foram localizados apenas na Europa e na Ásia Ocidental. Muito provavelmente, o intercâmbio genético ocorreu entre neandertais e humanos modernos quando os humanos modernos emergiram da África, antes da divergência de europeus, asiáticos orientais e papua-nova-guineenses.
Vários genes parecem ter sofrido mudanças dos neandertais durante a evolução dos humanos atuais. Esses genes estão envolvidos na estrutura craniana, no metabolismo, na morfologia da pele e no desenvolvimento cognitivo. Um dos genes que é de particular interesse é o RUNX2, que é diferente nos humanos e nos neandertais modernos. Esse gene é responsável pelo osso frontal proeminente, pela caixa torácica em forma de sino e pelas diferenças dentárias observadas nos neandertais. Especula-se que uma mudança evolutiva no RUNX2 tenha sido importante na origem dos humanos modernos, e isso afetou o crânio e a parte superior do corpo.
Link para o aprendizado

Assista à palestra de Svante Pääbo explicando a pesquisa do genoma neandertal na conferência anual TED (Tecnologia, Entretenimento, Design) de 2011.
Embalagem de DNA em células
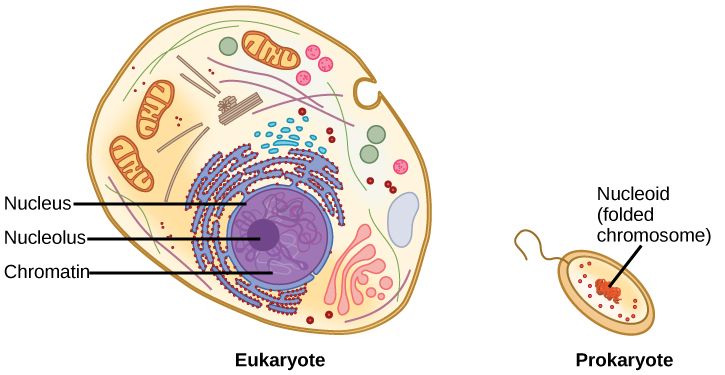
Ao comparar células procarióticas com células eucarióticas, os procariontes são muito mais simples do que os eucariotos em muitas de suas características (Figura\(\PageIndex{6}\)). A maioria dos procariontes contém um único cromossomo circular que é encontrado em uma área do citoplasma chamada nucleóide.
Exercício\(\PageIndex{1}\)
Nas células eucarióticas, a síntese de DNA e RNA ocorre em um compartimento separado da síntese protéica. Nas células procarióticas, os dois processos ocorrem juntos. Quais as vantagens de separar os processos? Quais são as vantagens de fazê-los ocorrer juntos?
- Responda
-
A compartimentalização permite que uma célula eucariótica divida os processos em etapas discretas para que possa construir produtos de proteína e RNA mais complexos. Mas há uma vantagem em ter um único compartimento: a síntese de RNA e proteína ocorre muito mais rapidamente em uma célula procariótica.
O tamanho do genoma em um dos procariontes mais bem estudados, E.coli, é de 4,6 milhões de pares de bases (aproximadamente 1,1 mm, se cortado e esticado). Então, como isso se encaixa dentro de uma pequena célula bacteriana? O DNA é distorcido pelo que é conhecido como superenrolamento. Superenrolamento significa que o DNA está sub-enrolado (menos de uma volta da hélice por 10 pares de bases) ou enrolado (mais de 1 volta por 10 pares de bases) de seu estado normal de relaxamento. Sabe-se que algumas proteínas estão envolvidas no superenrolamento; outras proteínas e enzimas, como a DNA girase, ajudam na manutenção da estrutura superenrolada.
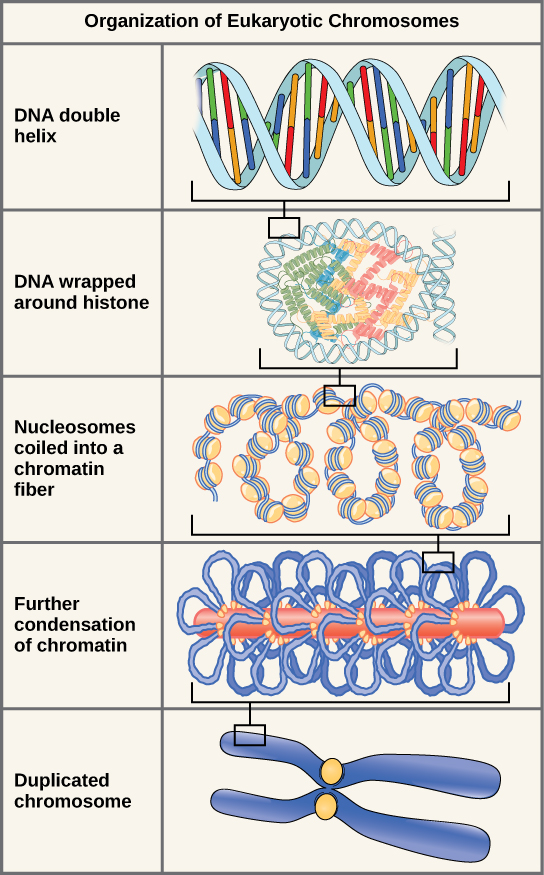
Os eucariotos, cujos cromossomos consistem cada um em uma molécula linear de DNA, empregam um tipo diferente de estratégia de empacotamento para encaixar seu DNA dentro do núcleo (Figura\(\PageIndex{7}\)). No nível mais básico, o DNA é envolvido em proteínas conhecidas como histonas para formar estruturas chamadas nucleossomos. As histonas são proteínas evolutivamente conservadas que são ricas em aminoácidos básicos e formam um octâmero. O DNA (que é carregado negativamente por causa dos grupos fosfato) é enrolado firmemente ao redor do núcleo da histona. Esse nucleossomo é ligado ao próximo com a ajuda de um DNA ligante. Isso também é conhecido como estrutura de “contas em uma corda”. Isso é ainda compactado em uma fibra de 30 nm, que é o diâmetro da estrutura. No estágio de metáfase, os cromossomos são mais compactos, têm aproximadamente 700 nm de largura e são encontrados em associação com proteínas de suporte.
Na interfase, os cromossomos eucarióticos têm duas regiões distintas que podem ser distinguidas pela coloração. A região altamente compactada é conhecida como heterocromatina, e a região menos densa é conhecida como eucromatina. A heterocromatina geralmente contém genes que não são expressos e é encontrada nas regiões do centrômero e dos telômeros. A eucromatina geralmente contém genes que são transcritos, com DNA empacotado em torno dos nucleossomos, mas não compactado posteriormente.
Resumo
O modelo atualmente aceito da estrutura de dupla hélice do DNA foi proposto por Watson e Crick. Algumas das principais características são que os dois fios que compõem a dupla hélice são de natureza complementar e antiparalela. Açúcares e fosfatos de desoxirribose formam a espinha dorsal da estrutura e as bases nitrogenadas são empilhadas em seu interior. O diâmetro da dupla hélice, 2 nm, é uniforme por toda parte. Uma purina sempre emparelha com uma pirimidina; A emparelha com T e G emparelha com C. Uma volta da hélice tem dez pares de bases. Durante a divisão celular, cada célula filha recebe uma cópia do DNA por um processo conhecido como replicação do DNA. Os procariontes são muito mais simples do que os eucariotos em muitas de suas características. A maioria dos procariontes contém um único cromossomo circular. Em geral, os cromossomos eucarióticos contêm uma molécula linear de DNA empacotada em nucleossomos e têm duas regiões distintas que podem ser distinguidas por coloração, refletindo diferentes estados de embalagem e compactação.
Notas de pé
- 1 Richard E. Green et al., “A Rascunho da Sequência do Genoma Neandertal”, Science 328 (2010): 710-22.
Glossário
- eletroforese
- técnica usada para separar fragmentos de DNA de acordo com o tamanho


