10.3: Genômica e Proteômica
- Page ID
- 179086
O estudo dos ácidos nucléicos começou com a descoberta do DNA, progrediu para o estudo de genes e pequenos fragmentos e agora explodiu no campo da genômica. Genômica é o estudo de genomas inteiros, incluindo o conjunto completo de genes, sua sequência e organização de nucleotídeos e suas interações dentro de uma espécie e com outras espécies. Os avanços na genômica foram possíveis graças à tecnologia de sequenciamento de DNA. Assim como a tecnologia da informação levou ao Google Maps, que nos permite obter informações detalhadas sobre locais ao redor do mundo, as informações genômicas são usadas para criar mapas semelhantes do DNA de diferentes organismos.
Mapeando genomas
O mapeamento do genoma é o processo de encontrar a localização dos genes em cada cromossomo. Os mapas criados são comparáveis aos mapas que usamos para navegar pelas ruas. Um mapa genético é uma ilustração que lista genes e sua localização em um cromossomo. Os mapas genéticos fornecem uma visão geral (semelhante a um mapa de rodovias interestaduais) e usam marcadores genéticos (semelhantes aos pontos turísticos). Um marcador genético é um gene ou sequência em um cromossomo que mostra uma ligação genética com uma característica de interesse. O marcador genético tende a ser herdado com o gene de interesse, e uma medida da distância entre eles é a frequência de recombinação durante a meiose. Os primeiros geneticistas chamaram isso de análise de ligação.
Os mapas físicos abordam os detalhes íntimos de regiões menores dos cromossomos (semelhante a um roteiro detalhado) (Figura\(\PageIndex{1}\)). Um mapa físico é uma representação da distância física, em nucleotídeos, entre genes ou marcadores genéticos. Tanto mapas de ligação genética quanto mapas físicos são necessários para construir uma imagem completa do genoma. Ter um mapa completo do genoma torna mais fácil para os pesquisadores estudarem genes individuais. Os mapas do genoma humano ajudam os pesquisadores em seus esforços para identificar genes causadores de doenças humanas relacionados a doenças como câncer, doenças cardíacas e fibrose cística, para citar alguns. Além disso, o mapeamento do genoma pode ser usado para ajudar a identificar organismos com características benéficas, como micróbios com a capacidade de limpar poluentes ou até mesmo prevenir a poluição. Pesquisas envolvendo mapeamento do genoma vegetal podem levar a métodos que produzem maiores rendimentos agrícolas ou ao desenvolvimento de plantas que se adaptam melhor às mudanças climáticas.
![Um diagrama mostrando um cromossomo humano com bandas reveladas com uma coloração de Giemsa. As bandas são rotuladas com Xp e um número no braço curto e Xq e um número no braço longo. Certos genes são encontrados em algumas das bandas. Esses genes estão rotulados à direita: anemia de Fanconi B, síndrome de Wiskott-Aldrich, doença de Pelizaeus-Merzbacher, síndrome do X frágil e deficiência de G6PD [0].](https://bio.libretexts.org/@api/deki/files/9384/Figure_10_03_01.jpg)
Os mapas genéticos fornecem o esboço e os mapas físicos fornecem os detalhes. É fácil entender por que os dois tipos de técnicas de mapeamento genômico são importantes para mostrar o panorama geral. As informações obtidas de cada técnica são usadas em combinação para estudar o genoma. O mapeamento genômico é usado com diferentes organismos modelo usados para pesquisas. O mapeamento do genoma ainda é um processo contínuo e, à medida que técnicas mais avançadas são desenvolvidas, mais avanços são esperados. O mapeamento do genoma é semelhante a completar um quebra-cabeça complicado usando todos os dados disponíveis. As informações de mapeamento geradas em laboratórios de todo o mundo são inseridas em bancos de dados centrais, como o National Center for Biotechnology Information (NCBI). Esforços são feitos para tornar as informações mais facilmente acessíveis aos pesquisadores e ao público em geral. Assim como usamos sistemas de posicionamento global em vez de mapas em papel para navegar pelas estradas, o NCBI nos permite usar uma ferramenta de visualização de genoma para simplificar o processo de mineração de dados.
CONCEITO EM AÇÃO

A Herança Mendeliana Online no Homem (OMIM) é um catálogo on-line pesquisável de genes humanos e doenças genéticas. Este site mostra o mapeamento do genoma e também detalha a história e a pesquisa de cada característica e distúrbio. Clique no link para pesquisar características (como destreza manual) e doenças genéticas (como diabetes).
Sequenciamento do genoma completo
Embora tenha havido avanços significativos nas ciências médicas nos últimos anos, os médicos ainda estão confusos com muitas doenças e os pesquisadores estão usando o sequenciamento do genoma completo para resolver o problema. O sequenciamento do genoma completo é um processo que determina a sequência de DNA de um genoma inteiro. O sequenciamento do genoma completo é uma abordagem de força bruta para resolver problemas quando há uma base genética no centro de uma doença. Vários laboratórios agora oferecem serviços para sequenciar, analisar e interpretar genomas inteiros.
Em 2010, o sequenciamento do genoma completo foi usado para salvar um menino cujos intestinos tinham vários abscessos misteriosos. A criança passou por várias operações no cólon sem alívio. Finalmente, uma sequência completa do genoma revelou um defeito em uma via que controla a apoptose (morte celular programada). Um transplante de medula óssea foi usado para superar esse distúrbio genético, levando à cura do menino. Ele foi a primeira pessoa a ser diagnosticada com sucesso usando o sequenciamento do genoma completo.
Os primeiros genomas a serem sequenciados, como aqueles pertencentes a vírus, bactérias e leveduras, eram menores em termos de número de nucleotídeos do que os genomas de organismos multicelulares. Os genomas de outros organismos modelo, como o camundongo (Mus musculus), a mosca da fruta (Drosophila melanogaster) e o nematóide (Caenorhabditis elegans) são agora conhecidos. Muitas pesquisas básicas são realizadas em organismos modelo porque as informações podem ser aplicadas a outros organismos. Um organismo modelo é uma espécie estudada como modelo para entender os processos biológicos em outras espécies que podem ser representadas pelo organismo modelo. Por exemplo, as moscas da fruta são capazes de metabolizar o álcool como os humanos, então os genes que afetam a sensibilidade ao álcool foram estudados nas moscas-das-frutas em um esforço para entender a variação na sensibilidade ao álcool em humanos. Ter genomas inteiros sequenciados ajuda nos esforços de pesquisa nesses organismos modelo (Figura\(\PageIndex{2}\)).

A primeira sequência do genoma humano foi publicada em 2003. O número de genomas inteiros que foram sequenciados aumenta constantemente e agora inclui centenas de espécies e milhares de genomas humanos individuais.
Aplicando genômica
A introdução de projetos de sequenciamento de DNA e sequenciamento do genoma completo, particularmente o Projeto Genoma Humano, expandiu a aplicabilidade das informações da sequência de DNA. A genômica agora está sendo usada em uma ampla variedade de campos, como metagenômica, farmacogenômica e genômica mitocondrial. A aplicação mais conhecida da genômica é entender e encontrar curas para doenças.
Prevendo o risco de doenças no nível individual
Prever o risco de doenças envolve o rastreamento e a identificação de indivíduos atualmente saudáveis por meio da análise do genoma em nível individual. A intervenção com mudanças no estilo de vida e medicamentos pode ser recomendada antes do início da doença. No entanto, essa abordagem é mais aplicável quando o problema surge de uma única mutação genética. Esses defeitos representam apenas cerca de 5% das doenças encontradas nos países desenvolvidos. A maioria das doenças comuns, como doenças cardíacas, são multifatoriais ou poligênicas, o que se refere a uma característica fenotípica determinada por dois ou mais genes e também a fatores ambientais, como a dieta. Em abril de 2010, cientistas da Universidade de Stanford publicaram a análise do genoma de um indivíduo saudável (Stephen Quake, cientista da Universidade de Stanford, que teve seu genoma sequenciado); a análise previu sua propensão a adquirir várias doenças. Uma avaliação de risco foi feita para analisar a porcentagem de risco de Quake para 55 condições médicas diferentes. Foi encontrada uma mutação genética rara que mostrou que ele corria risco de ataque cardíaco repentino. Também foi previsto que ele tenha um risco de 23% de desenvolver câncer de próstata e um risco de 1,4% de desenvolver a doença de Alzheimer. Os cientistas usaram bancos de dados e várias publicações para analisar os dados genômicos. Embora o sequenciamento genômico esteja se tornando mais acessível e as ferramentas analíticas estejam se tornando mais confiáveis, as questões éticas que envolvem a análise genômica em nível populacional ainda precisam ser abordadas. Por exemplo, esses dados poderiam ser usados legitimamente para cobrar mais ou menos pelo seguro ou para afetar as classificações de crédito?
Estudos de associação de todo o genoma
Desde 2005, é possível realizar um tipo de estudo chamado estudo de associação genômica ampla, ou GWAS. Um GWAS é um método que identifica diferenças entre indivíduos em polimorfismos de nucleotídeo único (SNPs) que podem estar envolvidos na causa de doenças. O método é particularmente adequado para doenças que podem ser afetadas por uma ou várias alterações genéticas em todo o genoma. É muito difícil identificar os genes envolvidos em tal doença usando informações da história da família. O método GWAS se baseia em um banco de dados genético que está em desenvolvimento desde 2002 chamado Projeto Internacional HapMap. O Projeto HapMap sequenciou os genomas de várias centenas de indivíduos de todo o mundo e identificou grupos de SNPs. Os grupos incluem SNPs que estão localizados próximos uns dos outros nos cromossomos, então eles tendem a permanecer juntos por meio de recombinação. O fato de o grupo permanecer junto significa que identificar um marcador SNP é tudo o que é necessário para identificar todos os SNPs no grupo. Existem vários milhões de SNPs identificados, mas identificá-los em outros indivíduos que não tiveram seu genoma completo sequenciado é muito mais fácil porque somente os SNPs marcadores precisam ser identificados.
Em um desenho comum para um GWAS, dois grupos de indivíduos são escolhidos; um grupo tem a doença e o outro grupo não. Os indivíduos de cada grupo são combinados em outras características para reduzir o efeito das variáveis de confusão que causam diferenças entre os dois grupos. Por exemplo, os genótipos podem ser diferentes porque os dois grupos são, em sua maioria, retirados de diferentes partes do mundo. Uma vez escolhidos os indivíduos, e normalmente seus números são de mil ou mais para que o estudo funcione, amostras de seu DNA são obtidas. O DNA é analisado usando sistemas automatizados para identificar grandes diferenças na porcentagem de SNPs específicos entre os dois grupos. Freqüentemente, o estudo examina um milhão ou mais de SNPs no DNA. Os resultados do GWAS podem ser usados de duas maneiras: as diferenças genéticas podem ser usadas como marcadores de suscetibilidade à doença em indivíduos não diagnosticados, e os genes específicos identificados podem ser alvos para pesquisas sobre a via molecular da doença e possíveis terapias. Um desdobramento da descoberta de associações genéticas com doenças foi a formação de empresas que fornecem a chamada “genômica pessoal”, que identificarão os níveis de risco para várias doenças com base no complemento SNP de um indivíduo. A ciência por trás desses serviços é controversa.
Como o GWAS busca associações entre genes e doenças, esses estudos fornecem dados para outras pesquisas sobre causas, em vez de responder a perguntas específicas por si mesmos. Uma associação entre uma diferença genética e uma doença não significa necessariamente que haja uma relação de causa e efeito. No entanto, alguns estudos forneceram informações úteis sobre as causas genéticas das doenças. Por exemplo, três estudos diferentes em 2005 identificaram um gene para uma proteína envolvida na regulação da inflamação no corpo que está associada a uma cegueira causadora de doenças chamada degeneração macular relacionada à idade. Isso abriu novas possibilidades para pesquisas sobre a causa dessa doença. Um grande número de genes foi identificado como associado à doença de Crohn usando o GWAS, e alguns deles sugeriram novos mecanismos hipotéticos para a causa da doença.
Farmacogenômica
A farmacogenômica envolve a avaliação da eficácia e segurança dos medicamentos com base nas informações da sequência genômica de um indivíduo. As informações pessoais da sequência do genoma podem ser usadas para prescrever medicamentos que sejam mais eficazes e menos tóxicos com base no genótipo de cada paciente. O estudo de mudanças na expressão gênica pode fornecer informações sobre o perfil de transcrição gênica na presença do medicamento, que pode ser usado como um indicador precoce do potencial de efeitos tóxicos. Por exemplo, genes envolvidos no crescimento celular e na morte celular controlada, quando perturbados, podem levar ao crescimento de células cancerosas. Estudos de todo o genoma também podem ajudar a encontrar novos genes envolvidos na toxicidade de medicamentos. As assinaturas genéticas podem não ser totalmente precisas, mas podem ser testadas mais detalhadamente antes que os sintomas patológicos surjam.
Metagenômica
Tradicionalmente, a microbiologia tem sido ensinada com a visão de que os microrganismos são melhor estudados em condições puras de cultura, o que envolve isolar um único tipo de célula e cultivá-la em laboratório. Como os microrganismos podem passar por várias gerações em questão de horas, seus perfis de expressão gênica se adaptam ao novo ambiente de laboratório muito rapidamente. Por outro lado, muitas espécies resistem a serem cultivadas isoladamente. A maioria dos microrganismos não vive como entidades isoladas, mas em comunidades microbianas conhecidas como biofilmes. Por todas essas razões, a cultura pura nem sempre é a melhor forma de estudar microrganismos. Metagenômica é o estudo dos genomas coletivos de várias espécies que crescem e interagem em um nicho ambiental. A metagenômica pode ser usada para identificar novas espécies mais rapidamente e analisar o efeito dos poluentes no meio ambiente (Figura\(\PageIndex{3}\)). As técnicas de metagenômica agora também podem ser aplicadas a comunidades de eucariotos superiores, como peixes.
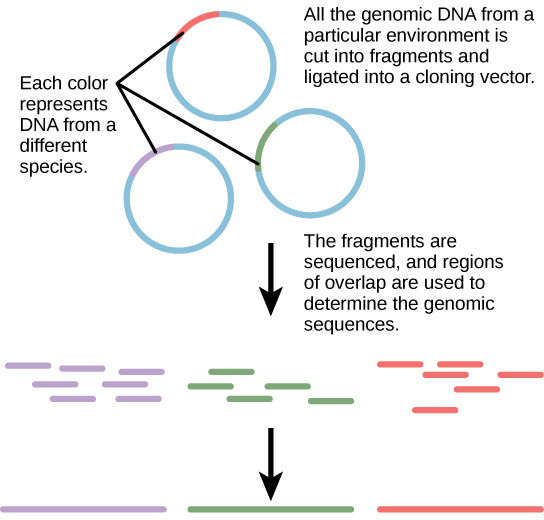
Criação de novos biocombustíveis
O conhecimento da genômica de microrganismos está sendo usado para encontrar melhores maneiras de aproveitar os biocombustíveis de algas e cianobactérias. As principais fontes de combustível atualmente são carvão, petróleo, madeira e outros produtos vegetais, como o etanol. Embora as usinas sejam recursos renováveis, ainda é necessário encontrar mais fontes alternativas de energia renovável para atender às demandas de energia de nossa população. O mundo microbiano é um dos maiores recursos para genes que codificam novas enzimas e produzem novos compostos orgânicos, e permanece praticamente inexplorado. Esse vasto recurso genético tem o potencial de fornecer novas fontes de biocombustíveis (Figura\(\PageIndex{4}\)).

Genômica mitocondrial
As mitocôndrias são organelas intracelulares que contêm seu próprio DNA. O DNA mitocondrial sofre mutações rapidamente e é frequentemente usado para estudar relações evolutivas. Outra característica que torna interessante o estudo do genoma mitocondrial é que, na maioria dos organismos multicelulares, o DNA mitocondrial é transmitido pela mãe durante o processo de fertilização. Por esse motivo, a genômica mitocondrial é frequentemente usada para rastrear a genealogia.
Genômica na análise forense
Informações e pistas obtidas de amostras de DNA encontradas em cenas de crimes foram usadas como prova em processos judiciais, e marcadores genéticos foram usados em análises forenses. A análise genômica também se tornou útil nesse campo. Em 2001, foi publicado o primeiro uso da genômica na ciência forense. Foi um esforço colaborativo entre instituições de pesquisa acadêmica e o FBI para resolver os misteriosos casos de antraz (Figura\(\PageIndex{5}\)) que foram transportados pelo Serviço Postal dos EUA. A bactéria antraz foi transformada em pó infeccioso e enviada para a mídia e dois senadores dos EUA. O pó infectou a equipe administrativa e os funcionários dos correios que abriram ou manipularam as cartas. Cinco pessoas morreram e 17 ficaram doentes com a bactéria. Usando a genômica microbiana, os pesquisadores determinaram que uma cepa específica de antraz era usada em todas as correspondências; eventualmente, a fonte foi rastreada até um cientista em um laboratório nacional de biodefesa em Maryland.
Genômica na Agricultura
A genômica pode reduzir os ensaios e falhas envolvidos na pesquisa científica até certo ponto, o que poderia melhorar a qualidade e a quantidade dos rendimentos das culturas na agricultura (Figura\(\PageIndex{6}\)). Vincular características a genes ou assinaturas genéticas ajuda a melhorar a criação de culturas para gerar híbridos com as qualidades mais desejáveis. Os cientistas usam dados genômicos para identificar características desejáveis e, em seguida, transferem essas características para um organismo diferente para criar um novo organismo geneticamente modificado, conforme descrito no módulo anterior. Os cientistas estão descobrindo como a genômica pode melhorar a qualidade e a quantidade da produção agrícola. Por exemplo, cientistas poderiam usar características desejáveis para criar um produto útil ou aprimorar um produto existente, como tornar uma cultura sensível à seca mais tolerante à estação seca.

Proteômica
As proteínas são os produtos finais dos genes que desempenham a função codificada pelo gene. As proteínas são compostas por aminoácidos e desempenham papéis importantes na célula. Todas as enzimas (exceto as ribozimas) são proteínas e atuam como catalisadores que afetam a taxa de reações. As proteínas também são moléculas reguladoras e algumas são hormônios. As proteínas transportadoras, como a hemoglobina, ajudam a transportar oxigênio para vários órgãos. Anticorpos que se defendem contra partículas estranhas também são proteínas. No estado doente, a função da proteína pode ser prejudicada devido a mudanças no nível genético ou devido ao impacto direto em uma proteína específica.
Um proteoma é o conjunto completo de proteínas produzidas por um tipo de célula. Os proteomas podem ser estudados usando o conhecimento dos genomas porque os genes codificam mRNAs e os mRNAs codificam proteínas. O estudo da função dos proteomas é chamado de proteômica. A proteômica complementa a genômica e é útil quando os cientistas querem testar suas hipóteses baseadas em genes. Embora todas as células de um organismo multicelular tenham o mesmo conjunto de genes, o conjunto de proteínas produzidas em diferentes tecidos é diferente e depende da expressão gênica. Assim, o genoma é constante, mas o proteoma varia e é dinâmico dentro de um organismo. Além disso, os RNAs podem ser alternativamente emendados (cortados e colados para criar novas combinações e novas proteínas), e muitas proteínas são modificadas após a tradução. Embora o genoma forneça um plano, a arquitetura final depende de vários fatores que podem mudar a progressão dos eventos que geram o proteoma.
Genomas e proteomas de pacientes que sofrem de doenças específicas estão sendo estudados para entender a base genética da doença. A doença mais proeminente que está sendo estudada com abordagens proteômicas é o câncer (Figura\(\PageIndex{7}\)). Abordagens proteômicas estão sendo usadas para melhorar o rastreamento e a detecção precoce do câncer; isso é obtido identificando proteínas cuja expressão é afetada pelo processo da doença. Uma proteína individual é chamada de biomarcador, enquanto um conjunto de proteínas com níveis de expressão alterados é chamado de assinatura proteica. Para que um biomarcador ou assinatura proteica seja útil como candidato ao rastreamento e detecção precoces de um câncer, ele deve ser secretado em fluidos corporais, como suor, sangue ou urina, para que exames em grande escala possam ser realizados de forma não invasiva. O problema atual do uso de biomarcadores para a detecção precoce do câncer é a alta taxa de resultados falso-negativos. Um resultado falso negativo é um resultado de teste negativo que deveria ter sido positivo. Em outras palavras, muitos casos de câncer não são detectados, o que torna os biomarcadores não confiáveis. Alguns exemplos de biomarcadores proteicos usados na detecção do câncer são CA-125 para câncer de ovário e PSA para câncer de próstata. As assinaturas de proteínas podem ser mais confiáveis do que os biomarcadores para detectar células cancerosas. A proteômica também está sendo usada para desenvolver planos de tratamento individualizados, que envolvem a previsão de se um indivíduo responderá ou não a medicamentos específicos e os efeitos colaterais que o indivíduo pode ter. A proteômica também está sendo usada para prever a possibilidade de recorrência da doença.

O Instituto Nacional do Câncer desenvolveu programas para melhorar a detecção e o tratamento do câncer. As Tecnologias Proteômicas Clínicas para Câncer e a Rede de Pesquisa de Detecção Precoce são esforços para identificar assinaturas de proteínas específicas para diferentes tipos de câncer. O Programa de Proteômica Biomédica foi desenvolvido para identificar assinaturas de proteínas e projetar terapias eficazes para pacientes com câncer.
Resumo
O mapeamento do genoma é semelhante a resolver um quebra-cabeça grande e complicado com informações provenientes de laboratórios de todo o mundo. Os mapas genéticos fornecem um esboço da localização dos genes dentro de um genoma e estimam a distância entre genes e marcadores genéticos com base na frequência de recombinação durante a meiose. Os mapas físicos fornecem informações detalhadas sobre a distância física entre os genes. As informações mais detalhadas estão disponíveis por meio do mapeamento de sequências. As informações de todas as fontes de mapeamento e sequenciamento são combinadas para estudar um genoma inteiro.
O sequenciamento do genoma completo é o recurso mais recente disponível para tratar doenças genéticas. Alguns médicos estão usando o sequenciamento completo do genoma para salvar vidas. A genômica tem muitas aplicações industriais, incluindo desenvolvimento de biocombustíveis, agricultura, produtos farmacêuticos e controle de poluição.
A imaginação é a única barreira para a aplicabilidade da genômica. A genômica está sendo aplicada à maioria dos campos da biologia; ela pode ser usada para medicina personalizada, previsão de riscos de doenças em nível individual, estudo de interações medicamentosas antes da realização de ensaios clínicos e estudo de microrganismos no meio ambiente, em oposição ao laboratório. Também está sendo aplicado à geração de novos biocombustíveis, avaliação genealógica usando mitocôndrias, avanços na ciência forense e melhorias na agricultura.
Proteômica é o estudo de todo o conjunto de proteínas expressas por um determinado tipo de célula sob certas condições ambientais. Em um organismo multicelular, diferentes tipos de células terão proteomas diferentes, e estes variarão com as mudanças no ambiente. Ao contrário de um genoma, um proteoma é dinâmico e está sob constante fluxo, o que o torna mais complicado e útil do que apenas o conhecimento dos genomas.
Glossário
- biomarcador
- uma proteína individual que é produzida exclusivamente em um estado doente
- mapa genético
- um esboço dos genes e sua localização em um cromossomo baseado em frequências de recombinação entre marcadores
- genômica
- o estudo de genomas inteiros, incluindo o conjunto completo de genes, sua sequência e organização de nucleotídeos e suas interações dentro de uma espécie e com outras espécies
- metagenômica
- o estudo dos genomas coletivos de várias espécies que crescem e interagem em um nicho ambiental
- organismo modelo
- uma espécie que é estudada e usada como modelo para entender os processos biológicos em outras espécies representadas pelo organismo modelo
- farmacogenômica
- o estudo das interações medicamentosas com o genoma ou proteoma; também chamado de toxicogenômica
- mapa físico
- uma representação da distância física entre genes ou marcadores genéticos
- assinatura de proteína
- um conjunto de proteínas sobreexpressas ou subexpressas características das células em um determinado tecido doente
- proteômica
- estudo da função dos proteomas
- sequenciamento do genoma completo
- um processo que determina a sequência de nucleotídeos de um genoma inteiro


