10.1: Clonagem e engenharia genética
- Page ID
- 179085
Biotecnologia é o uso de métodos artificiais para modificar o material genético de organismos vivos ou células para produzir novos compostos ou realizar novas funções. A biotecnologia tem sido usada para melhorar a pecuária e as plantações desde o início da agricultura por meio da criação seletiva. Desde a descoberta da estrutura do DNA em 1953, e particularmente desde o desenvolvimento de ferramentas e métodos para manipular o DNA na década de 1970, a biotecnologia se tornou sinônimo da manipulação do DNA dos organismos no nível molecular. As principais aplicações dessa tecnologia são na medicina (para a produção de vacinas e antibióticos) e na agricultura (para a modificação genética das culturas). A biotecnologia também tem muitas aplicações industriais, como fermentação, tratamento de derramamentos de óleo e produção de biocombustíveis, bem como muitas aplicações domésticas, como o uso de enzimas em detergentes para a roupa.
Manipulando material genético
Para realizar as aplicações descritas acima, os biotecnólogos devem ser capazes de extrair, manipular e analisar ácidos nucléicos.
Revisão da estrutura do ácido nucléico
Para entender as técnicas básicas usadas para trabalhar com ácidos nucléicos, lembre-se de que os ácidos nucléicos são macromoléculas feitas de nucleotídeos (açúcar, fosfato e base nitrogenada). Cada um dos grupos fosfato dessas moléculas tem uma carga negativa líquida. Um conjunto completo de moléculas de DNA no núcleo de organismos eucarióticos é chamado de genoma. O DNA tem duas fitas complementares ligadas por ligações de hidrogênio entre as bases emparelhadas.
Ao contrário do DNA nas células eucarióticas, as moléculas de RNA saem do núcleo. O RNA mensageiro (mRNA) é analisado com mais frequência porque representa os genes codificadores de proteínas que estão sendo expressos na célula.
Isolamento de ácidos nucleicos
Para estudar ou manipular ácidos nucléicos, o DNA deve primeiro ser extraído das células. Várias técnicas são usadas para extrair diferentes tipos de DNA (Figura\(\PageIndex{1}\)). A maioria das técnicas de extração de ácido nucléico envolve etapas para abrir a célula e, em seguida, o uso de reações enzimáticas para destruir todas as macromoléculas indesejadas. As células são abertas usando uma solução de detergente contendo compostos tamponantes. Para evitar a degradação e a contaminação, macromoléculas como proteínas e RNA são inativadas usando enzimas. O DNA é então retirado da solução usando álcool. O DNA resultante, por ser composto por polímeros longos, forma uma massa gelatinosa.
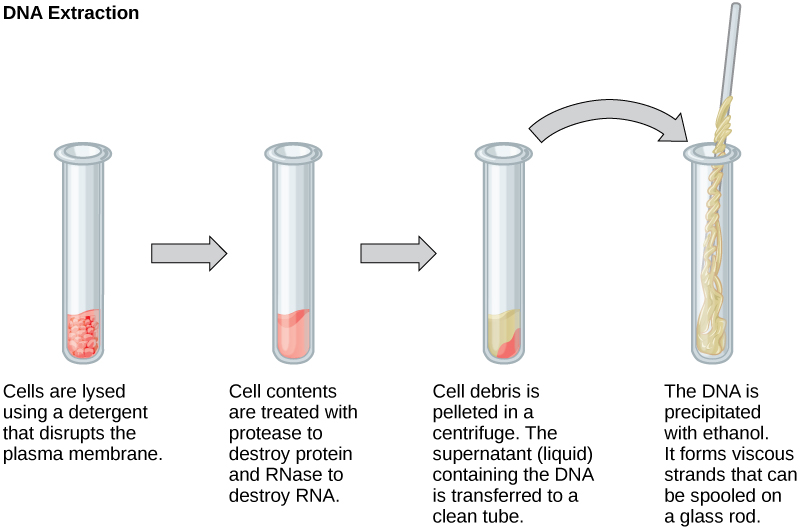
O RNA é estudado para entender os padrões de expressão gênica nas células. O RNA é naturalmente muito instável porque as enzimas que decompõem o RNA estão comumente presentes na natureza. Alguns são até secretados pela nossa própria pele e são muito difíceis de inativar. Semelhante à extração de DNA, a extração de RNA envolve o uso de vários tampões e enzimas para inativar outras macromoléculas e preservar apenas o RNA.
Eletroforese em gel
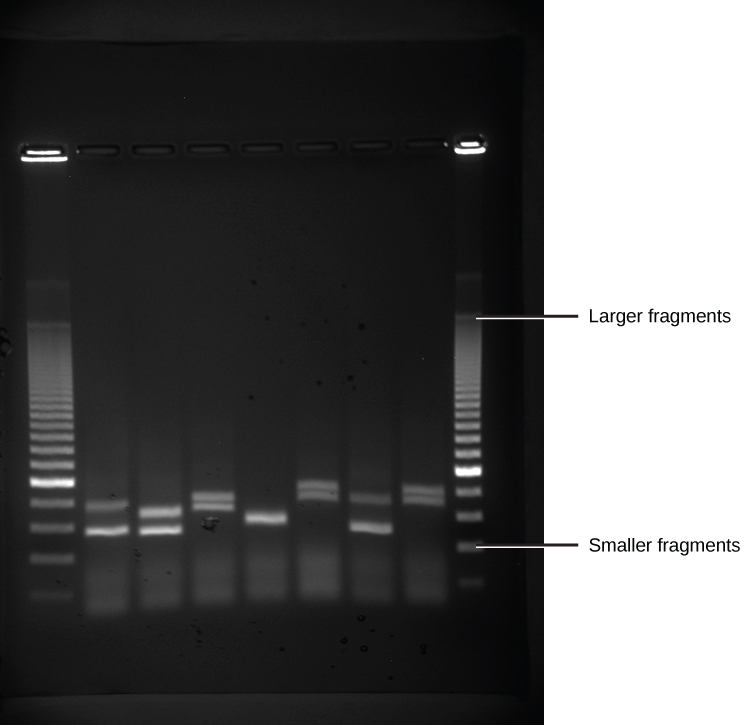
Como os ácidos nucléicos são íons carregados negativamente em pH neutro ou alcalino em um ambiente aquoso, eles podem ser movidos por um campo elétrico. A eletroforese em gel é uma técnica usada para separar moléculas carregadas com base no tamanho e na carga. Os ácidos nucléicos podem ser separados como cromossomos inteiros ou fragmentos. Os ácidos nucléicos são carregados em uma fenda em uma extremidade de uma matriz de gel, uma corrente elétrica é aplicada e as moléculas carregadas negativamente são puxadas para a extremidade oposta do gel (a extremidade com o eletrodo positivo). Moléculas menores se movem pelos poros do gel mais rapidamente do que moléculas maiores; essa diferença na taxa de migração separa os fragmentos com base no tamanho. Os ácidos nucléicos em uma matriz de gel são invisíveis até serem corados com um composto que permita sua visualização, como um corante. Fragmentos distintos de ácidos nucléicos aparecem como bandas a distâncias específicas da parte superior do gel (a extremidade negativa do eletrodo) que são baseadas em seu tamanho (Figura\(\PageIndex{2}\)). Uma mistura de muitos fragmentos de tamanhos variados aparece como uma mancha longa, enquanto o DNA genômico não cortado geralmente é muito grande para passar pelo gel e forma uma única faixa grande na parte superior do gel.
Reação em cadeia da polimerase
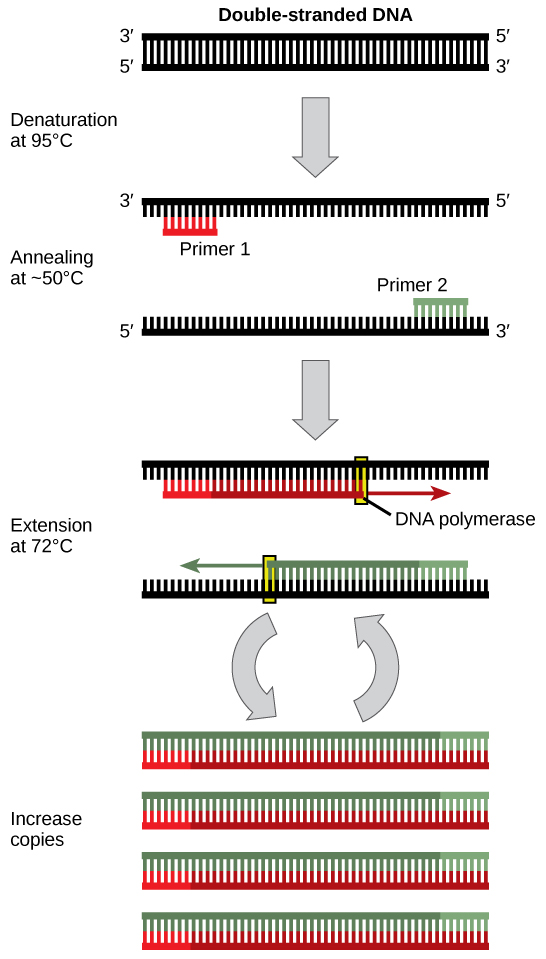
A análise de DNA geralmente requer o foco em uma ou mais regiões específicas do genoma. Também envolve frequentemente situações em que apenas uma ou algumas cópias de uma molécula de DNA estão disponíveis para análise posterior. Esses valores são insuficientes para a maioria dos procedimentos, como a eletroforese em gel. A reação em cadeia da polimerase (PCR) é uma técnica usada para aumentar rapidamente o número de cópias de regiões específicas do DNA para análises posteriores (Figura\(\PageIndex{3}\)). A PCR usa uma forma especial de DNA polimerase, a enzima que replica o DNA, e outras sequências curtas de nucleotídeos chamadas primers, que se unem a uma porção específica do DNA que está sendo replicada. O PCR é usado para muitos propósitos em laboratórios. Isso inclui: 1) a identificação do proprietário de uma amostra de DNA deixada na cena do crime; 2) análise de paternidade; 3) a comparação de pequenas quantidades de DNA antigo com organismos modernos; e 4) determinar a sequência de nucleotídeos em uma região específica.
Clonagem
Em geral, clonagem significa a criação de uma réplica perfeita. Normalmente, a palavra é usada para descrever a criação de uma cópia geneticamente idêntica. Em biologia, a recriação de um organismo inteiro é chamada de “clonagem reprodutiva”. Muito antes das tentativas de clonar um organismo inteiro, os pesquisadores aprenderam a copiar pequenos trechos de DNA, um processo conhecido como clonagem molecular.
Clonagem molecular
A clonagem permite a criação de várias cópias de genes, a expressão de genes e o estudo de genes específicos. Para colocar o fragmento de DNA em uma célula bacteriana em uma forma que será copiada ou expressa, o fragmento é primeiro inserido em um plasmídeo. Um plasmídeo (também chamado de vetor nesse contexto) é uma pequena molécula circular de DNA que se replica independentemente do DNA cromossômico das bactérias. Na clonagem, as moléculas de plasmídeo podem ser usadas para fornecer um “veículo” no qual inserir um fragmento de DNA desejado. Os plasmídeos modificados geralmente são reintroduzidos em um hospedeiro bacteriano para replicação. Conforme as bactérias se dividem, elas copiam seu próprio DNA (incluindo os plasmídeos). O fragmento de DNA inserido é copiado junto com o restante do DNA bacteriano. Em uma célula bacteriana, o fragmento de DNA do genoma humano (ou outro organismo que está sendo estudado) é chamado de DNA estranho para diferenciá-lo do DNA da bactéria (o DNA do hospedeiro).
Os plasmídeos ocorrem naturalmente em populações bacterianas (como a Escherichia coli) e têm genes que podem contribuir com características favoráveis ao organismo, como a resistência a antibióticos (a capacidade de não serem afetados por antibióticos). Os plasmídeos foram altamente projetados como vetores para clonagem molecular e para a subsequente produção em larga escala de moléculas importantes, como a insulina. Uma característica valiosa dos vetores de plasmídeo é a facilidade com que um fragmento estranho de DNA pode ser introduzido. Esses vetores de plasmídeo contêm muitas sequências curtas de DNA que podem ser cortadas com diferentes enzimas de restrição comumente disponíveis. As enzimas de restrição (também chamadas de endonucleases de restrição) reconhecem sequências específicas de DNA e as cortam de maneira previsível; elas são produzidas naturalmente pelas bactérias como um mecanismo de defesa contra DNA estranho. Muitas enzimas de restrição fazem cortes escalonados nas duas fitas do DNA, de modo que as extremidades cortadas tenham uma saliência de fita simples de 2 a 4 nucleotídeos. A sequência reconhecida pela enzima de restrição é uma sequência de quatro a oito nucleotídeos que é um palíndromo. Como com uma palavra palíndromo, isso significa que a sequência tem a mesma leitura para frente e para trás. Na maioria dos casos, a sequência tem a mesma leitura para frente em um fio e para trás no fio complementar. Quando um corte escalonado é feito em uma sequência como essa, as saliências são complementares (Figura\(\PageIndex{4}\)).
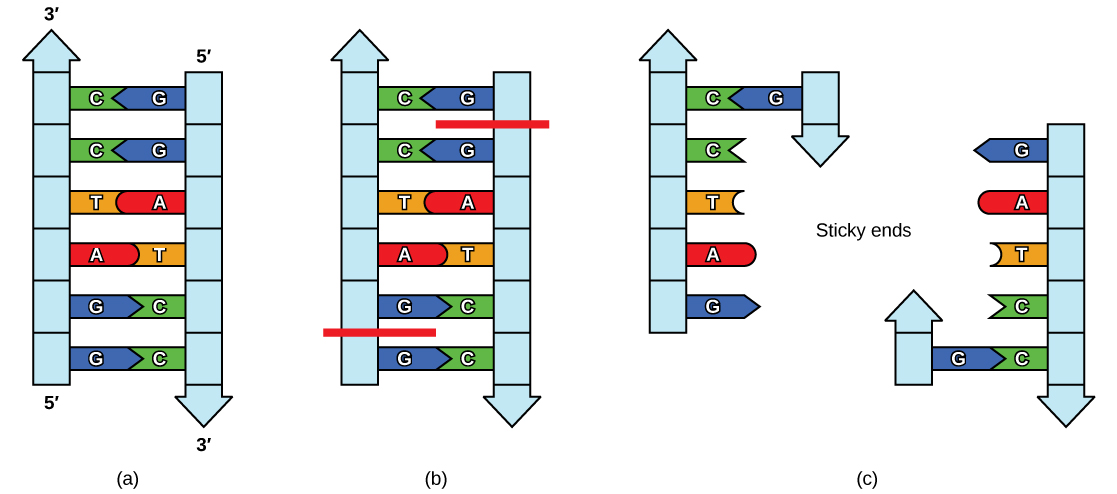
Como essas saliências são capazes de se unir por ligação de hidrogênio com saliências complementares em um pedaço de DNA cortado com a mesma enzima de restrição, elas são chamadas de “pontas pegajosas”. O processo de formação de ligações de hidrogênio entre sequências complementares em fitas simples para formar DNA de fita dupla é chamado de recozimento. A adição de uma enzima chamada DNA ligase, que participa da replicação do DNA nas células, une permanentemente os fragmentos de DNA quando as pontas adesivas se juntam. Dessa forma, qualquer fragmento de DNA pode ser emendado entre as duas extremidades de um DNA plasmídico que foi cortado com a mesma enzima de restrição (Figura\(\PageIndex{5}\)).
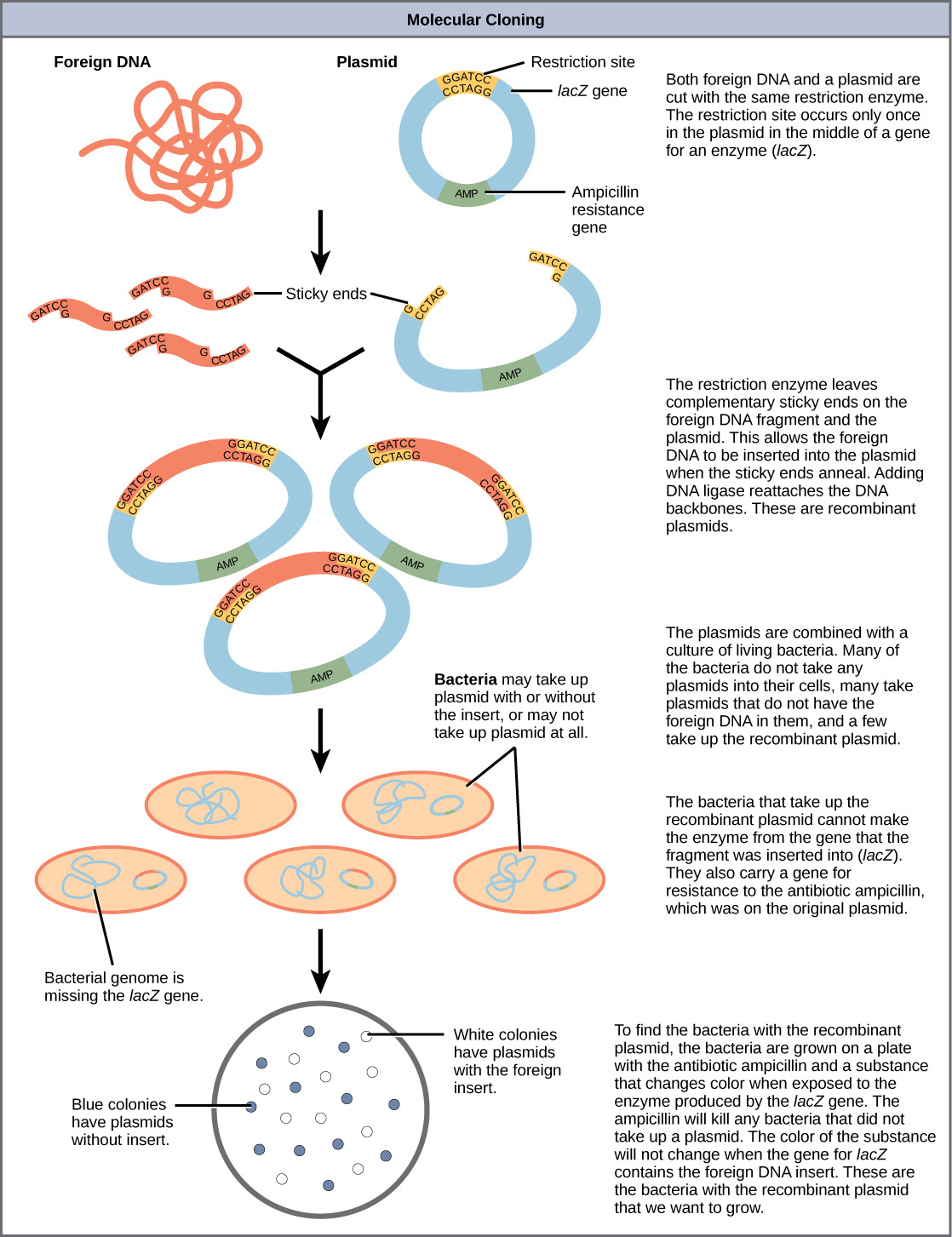
Plasmídeos com DNA estranho inserido neles são chamados de moléculas de DNA recombinante porque contêm novas combinações de material genético. As proteínas produzidas a partir de moléculas de DNA recombinante são chamadas de proteínas recombinantes. Nem todos os plasmídeos recombinantes são capazes de expressar genes. Os plasmídeos também podem ser projetados para expressar proteínas somente quando estimulados por certos fatores ambientais, para que os cientistas possam controlar a expressão das proteínas recombinantes.
Clonagem reprodutiva
A clonagem reprodutiva é um método usado para fazer um clone ou uma cópia idêntica de um organismo multicelular inteiro. A maioria dos organismos multicelulares se reproduz por meio sexual, o que envolve a contribuição do DNA de dois indivíduos (pais), impossibilitando a geração de uma cópia idêntica ou um clone de qualquer um dos pais. Avanços recentes na biotecnologia possibilitaram a clonagem reprodutiva de mamíferos em laboratório.
A reprodução sexual natural envolve a união, durante a fertilização, de um espermatozóide e um óvulo. Cada um desses gametas é haplóide, o que significa que eles contêm um conjunto de cromossomos em seus núcleos. A célula resultante, ou zigoto, é então diploide e contém dois conjuntos de cromossomos. Essa célula se divide mitoticamente para produzir um organismo multicelular. No entanto, a união de apenas duas células não pode produzir um zigoto viável; existem componentes no citoplasma do óvulo que são essenciais para o desenvolvimento precoce do embrião durante suas primeiras divisões celulares. Sem essas disposições, não haveria desenvolvimento posterior. Portanto, para produzir um novo indivíduo, é necessário um complemento genético diploide e um citoplasma de óvulo. A abordagem para produzir um indivíduo clonado artificialmente é pegar o óvulo de um indivíduo e remover o núcleo haplóide. Em seguida, um núcleo diploide de uma célula corporal de um segundo indivíduo, o doador, é colocado no óvulo. O ovo é então estimulado a se dividir para que o desenvolvimento prossiga. Isso parece simples, mas, na verdade, são necessárias muitas tentativas até que cada uma das etapas seja concluída com êxito.
O primeiro animal agrícola clonado foi Dolly, uma ovelha que nasceu em 1996. A taxa de sucesso da clonagem reprodutiva na época era muito baixa. Dolly viveu por seis anos e morreu de um tumor no pulmão (Figura\(\PageIndex{6}\)). Houve especulação de que, como o DNA celular que deu origem a Dolly veio de um indivíduo mais velho, a idade do DNA pode ter afetado sua expectativa de vida. Desde Dolly, várias espécies de animais (como cavalos, touros e cabras) foram clonadas com sucesso.
Houve tentativas de produzir embriões humanos clonados como fontes de células-tronco embrionárias. No procedimento, o DNA de um humano adulto é introduzido em um óvulo humano, que é então estimulado a se dividir. A tecnologia é semelhante à usada para produzir Dolly, mas o embrião nunca é implantado em uma mãe substituta. As células produzidas são chamadas de células-tronco embrionárias porque têm a capacidade de se desenvolver em muitos tipos diferentes de células, como células musculares ou nervosas. As células-tronco poderiam ser usadas para pesquisar e, finalmente, fornecer aplicações terapêuticas, como a substituição de tecidos danificados. O benefício da clonagem nesse caso é que as células usadas para regenerar novos tecidos seriam uma combinação perfeita com o doador do DNA original. Por exemplo, um paciente com leucemia não precisaria de um irmão com um tecido compatível para um transplante de medula óssea.
CONEXÃO ARTÍSTICA
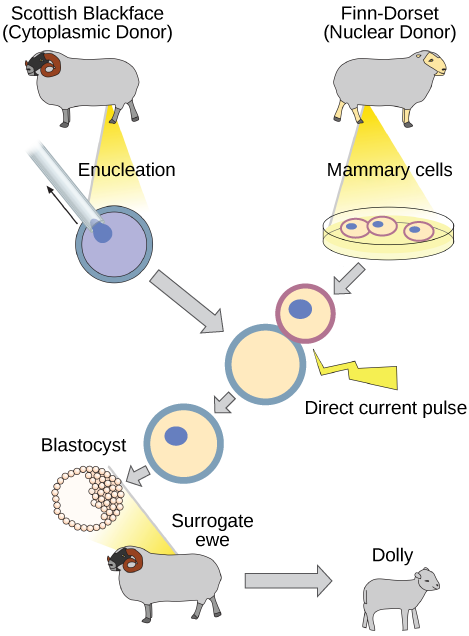
Por que Dolly era uma ovelha Finn-Dorset e não uma ovelha escocesa Blackface?
Engenharia Genética
Usar a tecnologia de DNA recombinante para modificar o DNA de um organismo e obter características desejáveis é chamado de engenharia genética. A adição de DNA estranho na forma de vetores de DNA recombinante que são gerados pela clonagem molecular é o método mais comum de engenharia genética. Um organismo que recebe o DNA recombinante é chamado de organismo geneticamente modificado (OGM). Se o DNA estranho que é introduzido vem de uma espécie diferente, o organismo hospedeiro é chamado de transgênico. Bactérias, plantas e animais foram geneticamente modificados desde o início dos anos 1970 para fins acadêmicos, médicos, agrícolas e industriais. Esses aplicativos serão examinados com mais detalhes no próximo módulo.
CONCEITO EM AÇÃO

Assista a este pequeno vídeo explicando como os cientistas criam um animal transgênico.
Embora os métodos clássicos de estudo da função dos genes tenham começado com um determinado fenótipo e determinem a base genética desse fenótipo, as técnicas modernas permitem que os pesquisadores comecem no nível da sequência de DNA e perguntem: “O que esse gene ou elemento de DNA faz?” Essa técnica, chamada de genética reversa, resultou na reversão da metodologia genética clássica. Um exemplo desse método é análogo a danificar uma parte do corpo para determinar sua função. Um inseto que perde uma asa não pode voar, o que significa que a função da asa é voar. O método genético clássico compara insetos que não podem voar com insetos que podem voar e observa que os insetos não voadores perderam asas. Da mesma forma, em uma abordagem genética reversa, a mutação ou exclusão de genes fornece aos pesquisadores pistas sobre a função gênica. Alternativamente, a genética reversa pode ser usada para fazer com que um gene se expresse demais para determinar quais efeitos fenotípicos podem ocorrer.
Resumo
Os ácidos nucléicos podem ser isolados das células para fins de análise posterior, abrindo as células e destruindo enzimaticamente todas as outras macromoléculas principais. Cromossomos fragmentados ou inteiros podem ser separados com base no tamanho por eletroforese em gel. Pequenos trechos de DNA podem ser amplificados por PCR. O DNA pode ser cortado (e posteriormente reagrupado) usando enzimas de restrição. As técnicas moleculares e celulares da biotecnologia permitem que os pesquisadores criem organismos geneticamente, modificando-os para obter características desejáveis.
A clonagem pode envolver a clonagem de pequenos fragmentos de DNA (clonagem molecular) ou a clonagem de organismos inteiros (clonagem reprodutiva). Na clonagem molecular com bactérias, um fragmento de DNA desejado é inserido em um plasmídeo bacteriano usando enzimas de restrição e o plasmídeo é absorvido por uma bactéria, que então expressará o DNA estranho. Usando outras técnicas, genes estranhos podem ser inseridos em organismos eucarióticos. Em cada caso, os organismos são chamados de organismos transgênicos. Na clonagem reprodutiva, um núcleo doador é colocado em um óvulo enucleado, que é então estimulado a se dividir e se transformar em um organismo.
Nos métodos de genética reversa, um gene é mutado ou removido de alguma forma para identificar seu efeito no fenótipo de todo o organismo como forma de determinar sua função.
Conexões artísticas
Figura\(\PageIndex{6}\): Por que Dolly era uma ovelha Finn-Dorset e não uma ovelha escocesa Blackface?
- Responda
-
Porque embora a célula original tenha vindo de uma ovelha escocesa Blackface e a mãe substituta fosse uma Scottish Blackface, o DNA veio de um Finn-Dorset.
Glossário
- recozimento
- em biologia molecular, o processo pelo qual duas fitas simples de DNA hidrogênio se ligam a nucleotídeos complementares para formar uma molécula de fita dupla
- biotecnologia
- o uso de métodos artificiais para modificar o material genético de organismos vivos ou células para produzir novos compostos ou realizar novas funções
- clonagem
- a produção de uma cópia exata — especificamente, uma cópia genética exata — de um gene, célula ou organismo
- eletroforese em gel
- uma técnica usada para separar moléculas com base em sua capacidade de migrar através de um gel semissólido em resposta a uma corrente elétrica
- engenharia genética
- alteração da composição genética de um organismo usando os métodos moleculares da biotecnologia
- organismo geneticamente modificado (OGM)
- um organismo cujo genoma foi alterado artificialmente
- plasmídeo
- uma pequena molécula circular de DNA encontrada em bactérias que se replica independentemente do cromossomo bacteriano principal; plasmídeos codificam algumas características importantes para bactérias e podem ser usados como vetores para transportar DNA para bactérias em aplicações de engenharia genética
- reação em cadeia da polimerase (PCR)
- uma técnica usada para fazer várias cópias do DNA
- DNA recombinante
- uma combinação de fragmentos de DNA gerados por clonagem molecular que não existe na natureza
- proteína recombinante
- uma proteína que é expressa a partir de moléculas de DNA recombinante
- enzima de restrição
- uma enzima que reconhece uma sequência específica de nucleotídeos no DNA e corta a fita dupla de DNA nesse local de reconhecimento, geralmente com um corte escalonado deixando fitas simples curtas ou pontas “pegajosas”
- genética reversa
- uma forma de análise genética que manipula o DNA para interromper ou afetar o produto de um gene para analisar a função do gene
- clonagem reprodutiva
- clonagem de organismos inteiros
- transgênico
- descrevendo um organismo que recebe DNA de uma espécie diferente


