11.4 : Synthèse des protéines (traduction)
- Page ID
- 187668
Objectifs d'apprentissage
- Décrivez le code génétique et expliquez pourquoi il est considéré comme presque universel
- Expliquer le processus de traduction et les fonctions de la machinerie moléculaire de traduction
- Comparez la traduction chez les eucaryotes et les procaryotes
La synthèse des protéines consomme plus d'énergie cellulaire que tout autre processus métabolique. À leur tour, les protéines représentent plus de masse que toute autre macromolécule des organismes vivants. Ils remplissent pratiquement toutes les fonctions d'une cellule, en tant qu'éléments fonctionnels (par exemple, enzymes) et structuraux. Le processus de traduction, ou synthèse des protéines, qui constitue la deuxième partie de l'expression des gènes, implique le décodage par un ribosome d'un message d'ARNm en un produit polypeptidique.
Le code génétique
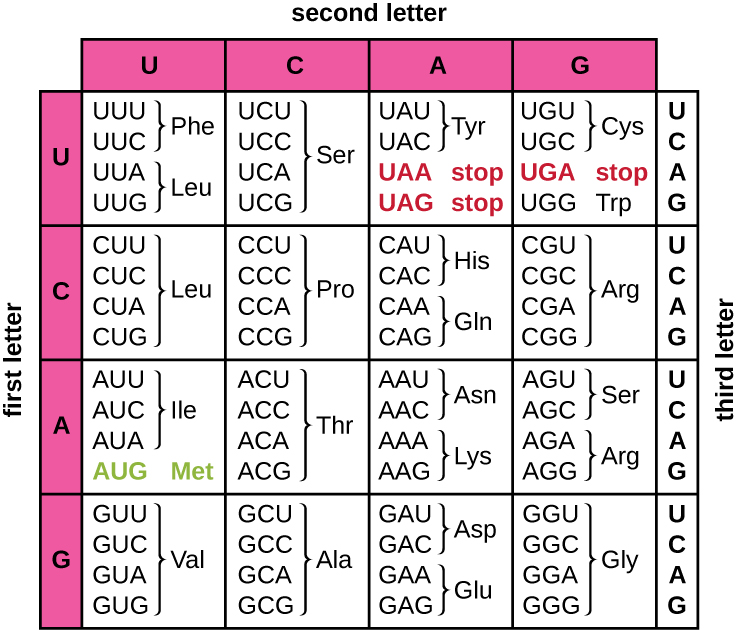
La traduction du modèle d'ARNm convertit les informations génétiques basées sur les nucléotides en « langage » des acides aminés pour créer un produit protéique. Une séquence protéique est constituée de 20 acides aminés courants. Chaque acide aminé est défini dans l'ARNm par un triplet de nucléotides appelé codon. La relation entre un codon d'ARNm et l'acide aminé correspondant est appelée code génétique.
Le code à trois nucléotides signifie qu'il existe un total de 64 combinaisons possibles (4 3, avec quatre nucléotides différents possibles à chacune des trois positions différentes au sein du codon). Ce nombre est supérieur au nombre d'acides aminés et un acide aminé donné est codé par plusieurs codons (Figure\(\PageIndex{1}\)). Cette redondance du code génétique est appelée dégénérescence. Généralement, alors que les deux premières positions d'un codon sont importantes pour déterminer quel acide aminé sera incorporé dans un polypeptide en croissance, la troisième position, appelée position d'oscillation, est moins critique. Dans certains cas, si le nucléotide en troisième position est modifié, le même acide aminé est toujours incorporé.
Alors que 61 des 64 triplets possibles codent pour des acides aminés, trois des 64 codons ne codent pas pour un acide aminé ; ils interrompent la synthèse des protéines, libérant ainsi le polypeptide de la machinerie de traduction. Ils sont appelés codon stop ou codon s absurde. Un autre codon, AUG, possède également une fonction spéciale. En plus de spécifier l'acide aminé méthionine, il sert généralement de codon de départ pour initier la traduction. Le cadre de lecture, c'est-à-dire la façon dont les nucléotides de l'ARNm sont regroupés en codons, pour la traduction, est défini par le codon AUG start près de l'extrémité 5' de l'ARNm. Chaque ensemble de trois nucléotides suivant ce codon de départ est un codon dans le message de l'ARNm.
Le code génétique est quasiment universel. À quelques exceptions près, pratiquement toutes les espèces utilisent le même code génétique pour la synthèse des protéines, ce qui prouve clairement que toutes les formes de vie existantes sur Terre ont une origine commune. Cependant, des acides aminés inhabituels tels que la sélénocystéine et la pyrrolysine ont été observés chez les archées et les bactéries. Dans le cas de la sélénocystéine, le codon utilisé est l'UGA (normalement un codon stop). Cependant, l'UGA peut coder pour la sélénocystéine à l'aide d'une structure tige-boucle (connue sous le nom de séquence d'insertion de la sélénocystéine ou élément SECIS), qui se trouve dans la région 3' non traduite de l'ARNm. La pyrrolysine utilise un codon stop différent, l'UAG. L'incorporation de pyrrolysine nécessite le gène pyLS et un ARN de transfert (ARNt) unique avec un anticodon CUA.
Exercice\(\PageIndex{1}\)
- Combien de bases se trouvent dans chaque codon ?
- Quel acide aminé est codé par le codon AAU ?
- Que se passe-t-il lorsqu'un codon stop est atteint ?
La machine de synthèse des protéines
Outre le modèle d'ARNm, de nombreuses molécules et macromolécules contribuent au processus de traduction. La composition de chaque composant varie selon les taxons ; par exemple, les ribosomes peuvent être composés d'un nombre différent d'ARN ribosomaux (ARNr) et de polypeptides selon l'organisme. Cependant, les structures générales et les fonctions de la machinerie de synthèse des protéines sont comparables des bactéries aux cellules humaines. La traduction nécessite la saisie d'un modèle d'ARNm, de ribosomes, d'ARNt et de divers facteurs enzymatiques.
Ribosomes
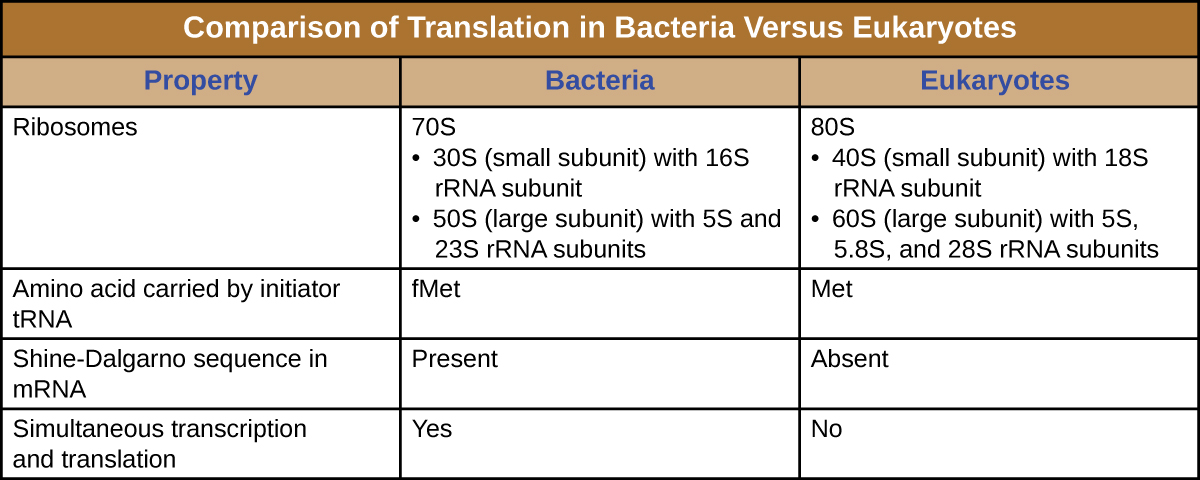
Un ribosome est une macromolécule complexe composée d'ARNr catalytiques (appelés ribozymes) et d'ARNr structuraux, ainsi que de nombreux polypeptides distincts. Les ARNr matures constituent environ 50 % de chaque ribosome. Les procaryotes possèdent des ribosomes 70S, tandis que les eucaryotes ont des ribosomes 80S dans le cytoplasme et le réticulum endoplasmique rugueux, et des ribosomes 70S dans les mitochondries et les chloroplastes. Les ribosomes se dissocient en grandes et petites sous-unités lorsqu'ils ne synthétisent pas de protéines et se réassocient au début de la traduction. Chez E. coli, la petite sous-unité est décrite comme 30S (qui contient la sous-unité de l'ARNr 16S) et la grande sous-unité est 50S (qui contient les sous-unités d'ARNr 5S et 23S), pour un total de 70S (les unités de Svedberg ne sont pas additives). Les ribosomes eucaryotes possèdent une petite sous-unité 40S (qui contient la sous-unité de l'ARNr 18S) et une grande sous-unité 60S (qui contient les sous-unités d'ARNr 5S, 5.8S et 28S), soit un total de 80S. La petite sous-unité est responsable de la liaison au modèle d'ARNm, tandis que la grande sous-unité lie les ARNt (voir la sous-section suivante).
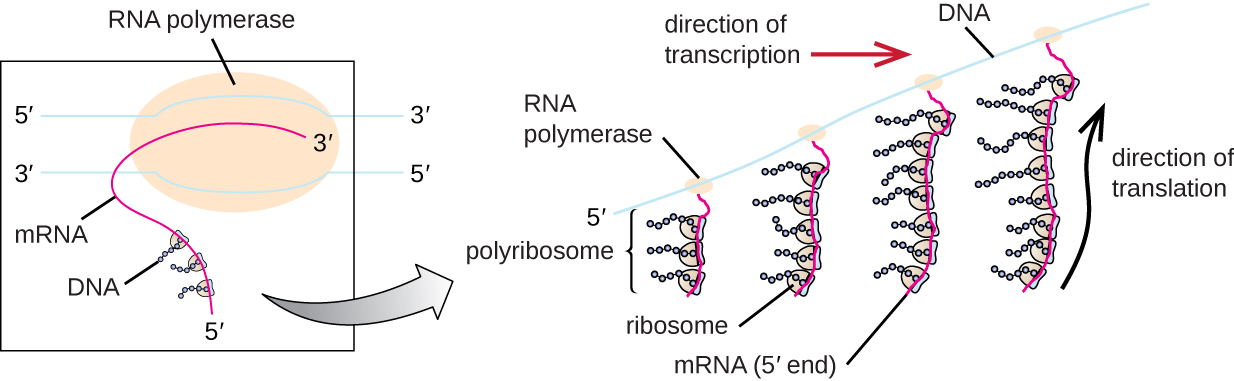
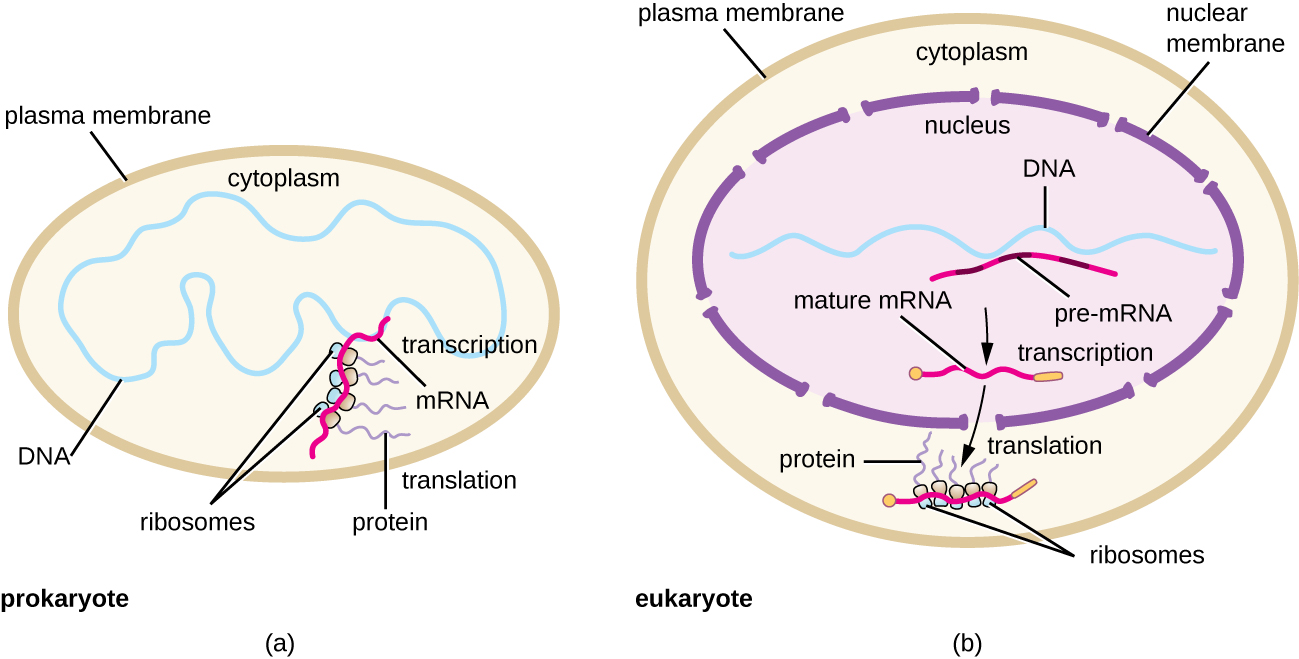
Chaque molécule d'ARNm est traduite simultanément par de nombreux ribosomes, tous synthétisant des protéines dans la même direction : lecture de l'ARNm de 5' à 3' et synthèse du polypeptide de l'extrémité N à l'extrémité C. La structure complète contenant un ARNm et de multiples ribosomes associés est appelée polyribosome (ou polysome). Tant chez les bactéries que chez les archées, avant la fin de la transcription, chaque transcrit codant pour une protéine est déjà utilisé pour commencer la synthèse de nombreuses copies du ou des polypeptides codés, car les processus de transcription et de traduction peuvent se produire simultanément, formant des polyribosomes (Figure \(\PageIndex{2}\)). La transcription et la traduction peuvent se produire simultanément parce que ces deux processus se produisent dans la même direction de 5' à 3', ils se produisent tous deux dans le cytoplasme de la cellule et parce que le transcrit d'ARN n'est pas traité une fois qu'il est transcrit. Cela permet à une cellule procaryote de répondre très rapidement à un signal environnemental nécessitant de nouvelles protéines. En revanche, dans les cellules eucaryotes, la transcription et la traduction simultanées ne sont pas possibles. Bien que des polyribosomes se forment également chez les eucaryotes, ils ne peuvent pas le faire tant que la synthèse de l'ARN n'est pas terminée et que la molécule d'ARN n'a pas été modifiée et transportée hors du noyau.
Transférer des ARN
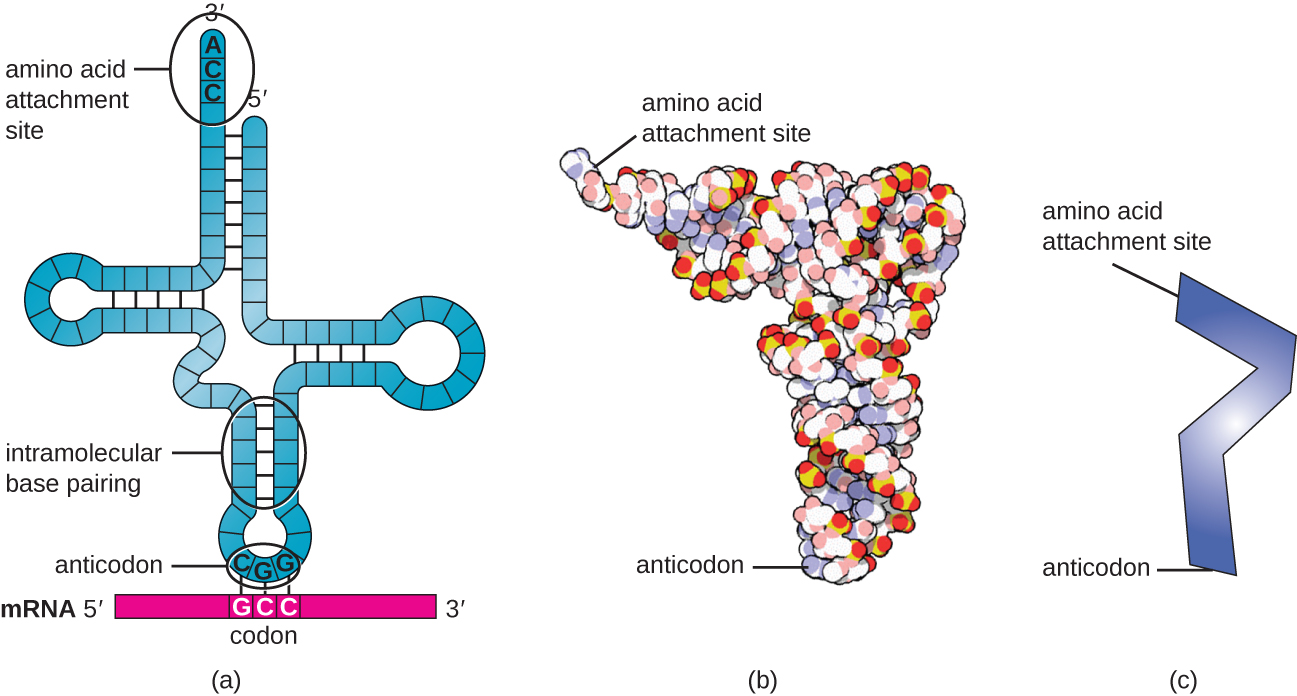
Les ARN de transfert (ARNt) sont des molécules d'ARN structurales et, selon les espèces, de nombreux types d'ARNt existent dans le cytoplasme. Les espèces bactériennes ont généralement entre 60 et 90 types. En tant qu'adaptateur, chaque type d'ARNt se lie à un codon spécifique sur la matrice d'ARNm et ajoute l'acide aminé correspondant à la chaîne polypeptidique. Par conséquent, les ARNt sont les molécules qui « traduisent » réellement le langage de l'ARN dans le langage des protéines. En tant que molécules adaptatrices de la traduction, il est surprenant que les ARNt puissent intégrer autant de spécificité dans un boîtier aussi petit. La molécule d'ARNt interagit avec trois facteurs : les aminoacyl tRNA synthétases, les ribosomes et l'ARNm.
Les ARNt matures prennent une structure tridimensionnelle lorsque des bases complémentaires exposées dans la molécule d'ARN monocaténaire se lient entre elles par hydrogène (Figure\(\PageIndex{3}\)). Cette forme positionne le site de liaison aux acides aminés, appelé extrémité de liaison aux acides aminés CCA, qui est une séquence cytosine-cytosine-adénine à l'extrémité 3' de l'ARNt, et l'anticodonat à l'autre extrémité. L'anticodon est une séquence de trois nucléotides qui se lie à un codon d'ARNm par un appariement de bases complémentaires.
Un acide aminé est ajouté à l'extrémité d'une molécule d'ARNt par le biais du processus de « charge » d'ARNt, au cours duquel chaque molécule d'ARNt est liée à son acide aminé correct ou apparenté par un groupe d'enzymes appelées aminoacyl tRNA synthétases. Au moins un type d'aminoacyl tRNA synthétase existe pour chacun des 20 acides aminés. Au cours de ce processus, l'acide aminé est d'abord activé par l'ajout d'adénosine monophosphate (AMP), puis transféré à l'ARNt, ce qui en fait un tRNA chargé, et l'AMP est libéré.
Exercice\(\PageIndex{2}\)
- Décrire la structure et la composition du ribosome procaryote.
- Dans quelle direction est lu le modèle d'ARNm ?
- Décrire la structure et la fonction d'un ARNt.
Le mécanisme de synthèse des protéines
La traduction est similaire chez les procaryotes et les eucaryotes. Nous allons explorer ici comment se produit la traduction chez E. coli, un procaryote représentatif, et préciser toute différence entre la traduction bactérienne et la traduction eucaryote.
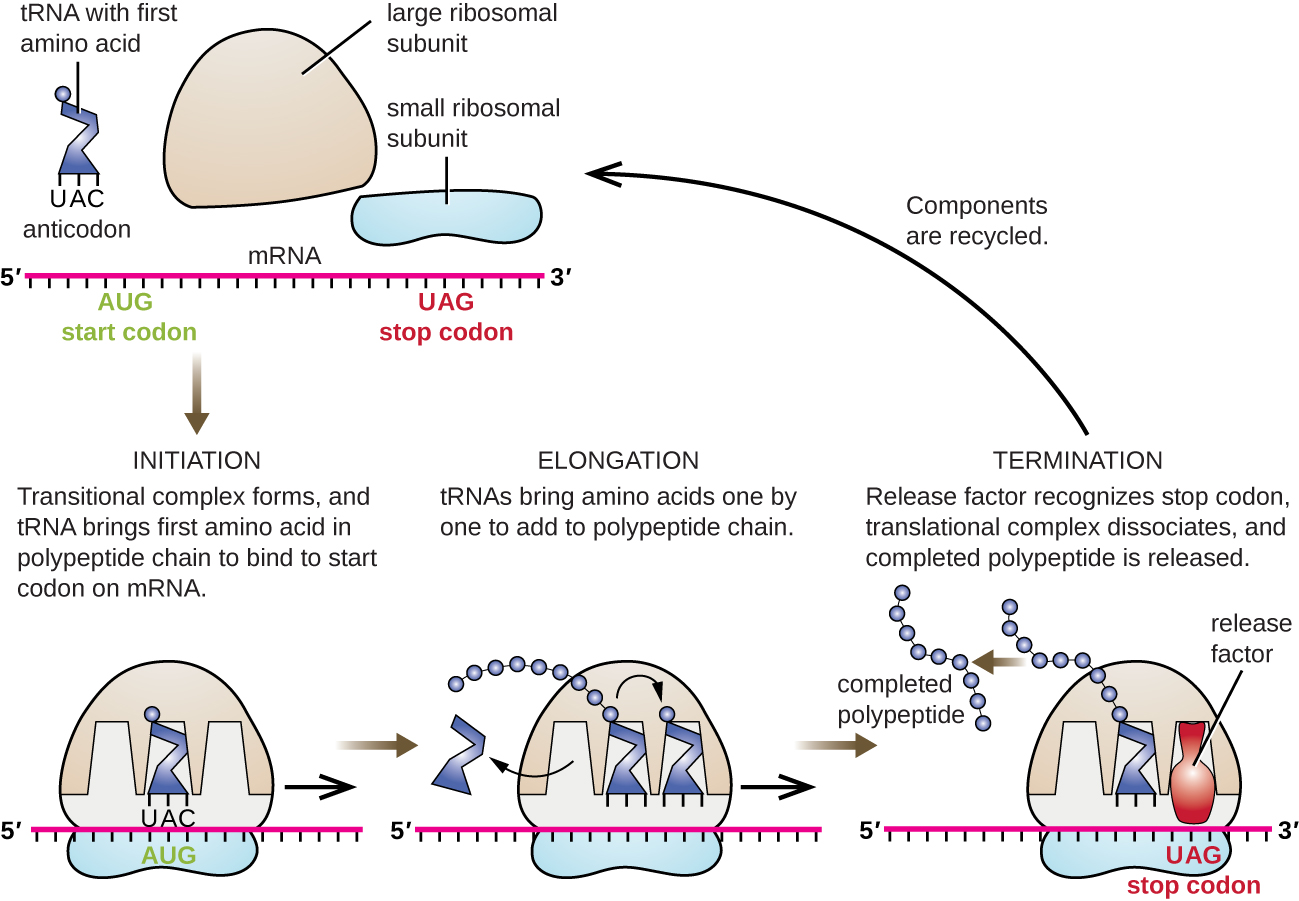
Initiation
L'initiation de la synthèse des protéines commence par la formation d'un complexe d'initiation. Chez E. coli, ce complexe implique le petit ribosome 30S, la matrice d'ARNm, trois facteurs d'initiation qui aident le ribosome à s'assembler correctement, le guanosine triphosphate (GTP) qui agit comme une source d'énergie, et un ARNt initiateur spécial transportant de la N-formyl-méthionine (fMET-tRNA fMET) ) (Figurine\(\PageIndex{4}\)). L'ARNt initiateur interagit avec le codon de départ AUG de l'ARNm et transporte une méthionine formylée (fMet). En raison de son implication dans l'initiation, le fMet est inséré au début (terminus N) de chaque chaîne polypeptidique synthétisée par E. coli. Dans l'ARNm d'E. coli, une séquence principale en amont du premier codon AUG, appelée séquence Shine-Dalgarno (également connue sous le nom de site de liaison ribosomique AGGAGG), interagit par appariement de bases complémentaires avec les molécules d'ARNr qui composent le ribosome. Cette interaction ancre la sous-unité ribosomale 30S au bon endroit sur le modèle d'ARNm. À ce stade, la sous-unité ribosomale 50S se lie ensuite au complexe d'initiation, formant un ribosome intact.
Chez les eucaryotes, la formation du complexe d'initiation est similaire, avec les différences suivantes :
- L'ARNt initiateur est un ARNt spécialisé différent transportant de la méthionine, appelé met-tRNAi
- Au lieu de se lier à l'ARNm au niveau de la séquence Shine-Dalgarno, le complexe d'initiation eucaryote reconnaît la coiffe 5' de l'ARNm eucaryote, puis suit le long de l'ARNm dans la direction 5' à 3' jusqu'à ce que le codon AUG start soit reconnu. À ce stade, la sous-unité 60S se lie au complexe du met-tRNAi, de l'ARNm et de la sous-unité 40S.
Allongement
Chez les procaryotes et les eucaryotes, les bases de l'allongement de la translation sont les mêmes. Chez E. coli, la liaison de la sous-unité ribosomale 50S pour produire le ribosome intact forme trois sites ribosomiques importants sur le plan fonctionnel : le site A (aminoacyl) lie les ARNt aminoacylés chargés entrants. Le site P (peptidyl) lie les ARNt chargés porteurs d'acides aminés qui ont formé des liaisons peptidiques avec la chaîne polypeptidique croissante mais qui ne se sont pas encore dissociés de leur ARNt correspondant. Le site E (exit) libère des ARNt dissociés afin qu'ils puissent être rechargés en acides aminés libres. Il existe une exception notable à cette chaîne d'assemblage d'ARNt : lors de la formation initiale du complexe, le fMET bactérien − ARNt fMET ou Met-ARNt eucaryote pénètre directement dans le site P sans entrer d'abord dans le site A, fournissant ainsi un site A libre prêt à accepter l'ARNt correspondant au premier codon après l'AUG.
L'élongation se produit par des mouvements du ribosome à un seul codon, chacun appelé événement de translocation. Lors de chaque événement de translocation, les ARNt chargés entrent sur le site A, puis se déplacent vers le site P, puis enfin vers le site E pour être retirés. Les mouvements ribosomiques, ou pas, sont induits par des changements conformationnels qui font avancer le ribosome de trois bases dans la direction 3'. Des liaisons peptidiques se forment entre le groupe amino de l'acide aminé attaché à l'ARNt du site A et le groupe carboxyle de l'acide aminé attaché à l'ARNt du site P. La formation de chaque liaison peptidique est catalysée par la peptidyl transférase, un ribozyme à base d'ARN intégré à la sous-unité ribosomale 50S. L'acide aminé lié à l'ARNt du site P est également lié à la chaîne polypeptidique croissante. Lorsque le ribosome traverse l'ARNm, l'ancien ARNt du site P entre dans le site E, se détache de l'acide aminé et est expulsé. Plusieurs des étapes de l'élongation, y compris la liaison d'un tRNA aminoacyle chargé au site A et la translocation, nécessitent de l'énergie dérivée de l'hydrolyse du GTP, qui est catalysée par des facteurs d'élongation spécifiques. Étonnamment, l'appareil de traduction d'E. coli ne prend que 0,05 seconde pour ajouter chaque acide aminé, ce qui signifie qu'une protéine de 200 acides aminés peut être traduite en seulement 10 secondes.
Résiliation
La fin de la traduction se produit lorsqu'un codon absurde (UAA, UAG ou UGA) est rencontré pour lequel il n'existe aucun ARNt complémentaire. En s'alignant sur le site A, ces codons absurdes sont reconnus par des facteurs de libération chez les procaryotes et les eucaryotes qui entraînent le détachement de l'acide aminé du site P de son ARNt, libérant ainsi le polypeptide nouvellement créé. Les petites et grandes sous-unités ribosomales se dissocient de l'ARNm et l'une de l'autre ; elles sont recrutées presque immédiatement dans un autre complexe d'initialisation de traduction.
En résumé, plusieurs caractéristiques clés distinguent l'expression des gènes procaryotes de celle observée chez les eucaryotes. Elles sont illustrées dans la figure\(\PageIndex{5}\) et répertoriées dans la figure\(\PageIndex{6}\).
Ciblage, repliement et modification des protéines
Pendant et après la traduction, les polypeptides peuvent avoir besoin d'être modifiés avant d'être biologiquement actifs. Les modifications post-traductionnelles incluent :
- élimination des séquences de signaux traduits, c'est-à-dire des séquences courtes d'acides aminés qui aident à diriger une protéine vers un compartiment cellulaire spécifique
- « repliement » approprié du polypeptide et association de plusieurs sous-unités polypeptidiques, souvent facilitées par des protéines chaperons, en une structure tridimensionnelle distincte
- traitement protéolytique d'un polypeptide inactif pour libérer un composant protéique actif, et
- diverses modifications chimiques (par exemple, phosphorylation, méthylation ou glycosylation) des acides aminés individuels.
Exercice\(\PageIndex{3}\)
- Quels sont les composants du complexe d'initiation à la traduction chez les procaryotes ?
- Quelles sont les deux différences entre l'initiation de la traduction procaryote et eucaryote ?
- Que se passe-t-il à chacun des trois sites actifs du ribosome ?
- Qu'est-ce qui cause l'arrêt de la traduction ?
Concepts clés et résumé
- Lors de la traduction, les polypeptides sont synthétisés à l'aide de séquences d'ARNm et de machines cellulaires, y compris des ARNt qui associent des codons d'ARNm à des acides aminés spécifiques et à des ribosomes composés d'ARN et de protéines qui catalysent la réaction.
- Le code génétique est dégénéré en ce sens que plusieurs codons d'ARNm codent pour les mêmes acides aminés. Le code génétique est presque universel chez les organismes vivants.
- Les ribosomes procaryotes (70S) et eucaryotes cytoplasmiques (80S) sont composés chacun d'une grande sous-unité et d'une petite sous-unité de tailles différentes entre les deux groupes. Chaque sous-unité est composée d'ARNr et de protéines. Les ribosomes organites des cellules eucaryotes ressemblent aux ribosomes procaryotes.
- De 60 à 90 espèces d'ARNt existent dans les bactéries. Chaque ARNt possède un anticodon à trois nucléotides ainsi qu'un site de liaison pour un acide aminé apparenté. Tous les ARNt dotés d'un anticodon spécifique porteront le même acide aminé.
- L'initiation de la traduction se produit lorsque la petite sous-unité ribosomale se lie à des facteurs d'initiation et à un ARNt initiateur au niveau du codon initial d'un ARNm, suivie de la liaison au complexe d'initiation de la grande sous-unité ribosomale.
- Dans les cellules procaryotes, le codon de départ code pour la N-formyl-méthionine transportée par un ARNt initiateur spécial. Dans les cellules eucaryotes, le codon de départ code la méthionine transportée par un ARNt initiateur spécial. De plus, alors que la liaison ribosomique de l'ARNm des procaryotes est facilitée par la séquence Shine-Dalgarno au sein de l'ARNm, les ribosomes eucaryotes se lient à la coiffe 5' de l'ARNm.
- Au cours de la phase d'élongation de la traduction, un ARNt chargé se lie à l'ARNm dans le site A du ribosome ; une liaison peptidique est catalysée entre les deux acides aminés adjacents, rompant la liaison entre le premier acide aminé et son ARNt ; le ribosome déplace un codon le long du ARNm ; et le premier ARNt est déplacé du site P du ribosome vers le site E et quitte le complexe ribosomal.
- La traduction se termine lorsque le ribosome rencontre un codon stop, qui ne code pas pour un ARNt. Les facteurs de libération provoquent la libération du polypeptide et la dissociation du complexe ribosomal.
- Chez les procaryotes, la transcription et la traduction peuvent être couplées, la traduction d'une molécule d'ARNm commençant dès que la transcription permet une exposition suffisante à l'ARNm pour la fixation d'un ribosome, avant la fin de la transcription. La transcription et la traduction ne sont pas couplées chez les eucaryotes parce que la transcription a lieu dans le noyau, alors que la traduction a lieu dans le cytoplasme ou en association avec le réticulum endoplasmique rugueux.
- Les polypeptides nécessitent souvent une ou plusieurs modifications post-traductionnelles pour devenir biologiquement actifs.


