11.3 : Transcription de l'ARN
- Page ID
- 187681
Objectifs d'apprentissage
- Expliquer comment l'ARN est synthétisé en utilisant l'ADN comme matrice
- Distinguer la transcription chez les procaryotes et chez les eucaryotes
Au cours du processus de transcription, les informations codées dans la séquence d'ADN d'un ou de plusieurs gènes sont transcrites dans un brin d'ARN, également appelé transcrit d'ARN. La molécule d'ARN monocaténaire qui en résulte, composée de ribonucléotides contenant les bases adénine (A), cytosine (C), guanine (G) et uracile (U), agit comme une copie moléculaire mobile de la séquence d'ADN d'origine. La transcription chez les procaryotes et les eucaryotes nécessite que la double hélice de l'ADN se déroule partiellement dans la région de synthèse de l'ARN. La région déroulée est appelée bulle de transcription. La transcription d'un gène particulier se fait toujours à partir de l'un des deux brins d'ADN qui servent de matrice, ce que l'on appelle le brin antisens. Le produit d'ARN est complémentaire au brin matrice de l'ADN et est presque identique au brin d'ADN non matrice, ou brin sensoriel. La seule différence est que dans l'ARN, tous les nucléotides T sont remplacés par des nucléotides U ; lors de la synthèse de l'ARN, U est incorporé lorsqu'il y a un A dans le brin antisens complémentaire.
Transcription chez les bactéries
Les bactéries utilisent la même ARN polymérase pour transcrire tous leurs gènes. Comme l'ADN polymérase, l'ARN polymérase ajoute des nucléotides un par un au groupe 3'-OH de la chaîne nucléotidique en croissance. L'une des principales différences d'activité entre l'ADN polymérase et l'ARN polymérase est la nécessité de disposer d'un 3'-OH sur lequel ajouter des nucléotides : l'ADN polymérase a besoin d'un tel groupe 3'-OH, nécessitant donc une amorce, alors que l'ARN polymérase n'en a pas besoin. Au cours de la transcription, un ribonucléotide complémentaire au brin matrice d'ADN est ajouté au brin d'ARN en croissance et une liaison phosphodiester covalente est formée par synthèse par déshydratation entre le nouveau nucléotide et le dernier nucléotide ajouté. Chez E. coli, l'ARN polymérase comprend six sous-unités polypeptidiques, dont cinq constituent l'enzyme centrale de la polymérase responsable de l'ajout de nucléotides d'ARN à un brin en croissance. La sixième sous-unité est connue sous le nom de sigma (σ). Le facteur σ permet à l'ARN polymérase de se lier à un promoteur spécifique, permettant ainsi la transcription de divers gènes. Il existe différents facteurs σ qui permettent la transcription de divers gènes.
Initiation
L'initiation de la transcription commence au niveau d'un promoteur, une séquence d'ADN sur laquelle le mécanisme de transcription se lie et initie la transcription. La paire de nucléotides de la double hélice de l'ADN qui correspond au site à partir duquel le premier nucléotide d'ARN 5' est transcrit est le site d'initiation. Les nucléotides précédant le site d'initiation sont désignés « en amont », tandis que les nucléotides suivant le site d'initiation sont appelés nucléotides « en aval ». Dans la plupart des cas, les promoteurs sont situés juste en amont des gènes qu'ils régulent. Bien que les séquences promotrices varient d'un génome bactérien à l'autre, quelques éléments sont conservés. Aux positions —10 et —35 dans l'ADN avant le site d'initiation (désigné +1), il existe deux séquences consensuelles de promoteurs, ou régions similaires chez tous les promoteurs et chez diverses espèces bactériennes. La séquence consensuelle —10, appelée boîte TATA, est TATAAT. La séquence —35 est reconnue et liée par σ.
Allongement
L'allongement de la phase de transcription commence lorsque la sous-unité σ se dissocie de la polymérase, ce qui permet à l'enzyme centrale de synthétiser de l'ARN complémentaire à la matrice d'ADN dans une direction 5' à 3' à un rythme d'environ 40 nucléotides par seconde. Au fur et à mesure que l'élongation se poursuit, l'ADN est continuellement déroulé devant l'enzyme centrale et enroulé à nouveau derrière elle (Figure\(\PageIndex{1}\)).
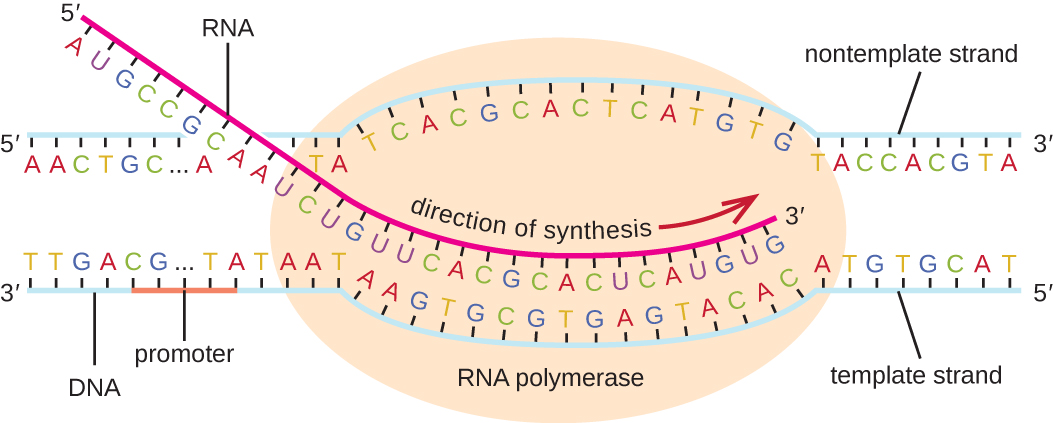
Résiliation
Une fois qu'un gène est transcrit, la polymérase bactérienne doit se dissocier de la matrice d'ADN et libérer l'ARN nouvellement créé. C'est ce que l'on appelle la fin de la transcription. La matrice d'ADN comprend des séquences nucléotidiques répétées qui agissent comme des signaux de terminaison, provoquant le blocage et la libération de l'ARN polymérase de la matrice d'ADN, libérant ainsi le transcrit d'ARN.
Exercice\(\PageIndex{1}\)
- Où le facteur σ de l'ARN polymérase lie-t-il l'ADN pour démarrer la transcription ?
- Que se passe-t-il pour initier l'activité de polymérisation de l'ARN polymérase ?
- D'où vient le signal de fin de transcription ?
Transcription chez les eucaryotes
Les procaryotes et les eucaryotes effectuent fondamentalement le même processus de transcription, avec quelques différences significatives (voir Tableau\(\PageIndex{1}\)). Les eucaryotes utilisent trois polymérases différentes, les ARN polymérases I, II et III, toutes structurellement distinctes de l'ARN polymérase bactérienne. Chacun transcrit un sous-ensemble différent de gènes. Il est intéressant de noter que les archées contiennent une seule ARN polymérase qui est plus étroitement apparentée à l'ARN polymérase II eucaryote qu'à son homologue bactérienne. Les ARNm des eucaryotes sont également généralement monocistroniques, c'est-à-dire qu'ils ne codent chacun qu'un seul polypeptide, tandis que les ARNm procaryotes des bactéries et des archées sont généralement polycistroniques, c'est-à-dire qu'ils codent pour de multiples polypeptides.
La différence la plus importante entre les procaryotes et les eucaryotes est le noyau membranaire de ces derniers, qui influence la facilité d'utilisation des molécules d'ARN pour la synthèse des protéines. Les gènes étant liés dans un noyau, la cellule eucaryote doit transporter les molécules d'ARN codant pour les protéines vers le cytoplasme pour être traduites. Les transcrits primaires codant pour les protéines, les molécules d'ARN directement synthétisées par l'ARN polymérase, doivent subir plusieurs étapes de traitement afin de protéger ces molécules d'ARN de la dégradation pendant leur transfert du noyau vers le cytoplasme et leur traduction en protéine. Par exemple, les ARNm eucaryotes peuvent durer plusieurs heures, alors que l'ARNm procaryote typique ne dure pas plus de 5 secondes.
Le transcrit primaire (également appelé pré-ARNm) est d'abord recouvert de protéines stabilisant l'ARN afin de le protéger de la dégradation pendant qu'il est traité et exporté hors du noyau. Le premier type de traitement commence alors que le transcrit primaire est encore en cours de synthèse ; un nucléotide spécial de 7-méthylguanosine, appelé cap 5', est ajouté à l'extrémité 5' du transcrit en croissance. En plus de prévenir la dégradation, les facteurs impliqués dans la synthèse protéique ultérieure reconnaissent le bouchon, ce qui aide à initier la traduction par les ribosomes. Une fois l'élongation terminée, une autre enzyme de traitement ajoute une chaîne d'environ 200 nucléotides d'adénine à l'extrémité 3', appelée queue poly-A. Cette modification protège davantage le pré-ARNm de la dégradation et indique aux facteurs cellulaires que le transcrit doit être exporté vers le cytoplasme.
Les gènes eucaryotes qui codent pour des polypeptides sont composés de séquences codantes appelées exons (ex -on signifie qu'ils sont exprimés) et de séquences intermédiaires appelées introns (int -ron indique leur rôle int ervenant). Les séquences d'ARN transcrites correspondant aux introns ne codent pas pour des régions du polypeptide fonctionnel et sont retirées du pré-ARNm pendant le traitement. Il est essentiel que toutes les séquences d'ARN codées par des introns soient complètement et précisément éliminées d'un pré-ARNm avant la synthèse des protéines afin que les séquences d'ARN codées par des exons soient correctement réunies pour coder un polypeptide fonctionnel. Si le processus échoue ne serait-ce que par un seul nucléotide, les séquences des exons réunis seraient décalées et le polypeptide résultant serait non fonctionnel. Le processus d'élimination des séquences d'ARN codées par des introns et de reconnexion de celles codées par des exons est appelé épissage de l'ARN et est facilité par l'action d'un épissage contenant de petites protéines ribonucléaires nucléaires (SNRNP). Les séquences d'ARN codées par des introns sont retirées du pré-ARNm alors que celui-ci se trouve encore dans le noyau. Bien qu'ils ne soient pas traduits, les introns semblent avoir diverses fonctions, notamment la régulation des gènes et le transport des ARNm. À la fin de ces modifications, le transcrit mature, l'ARNm qui code pour un polypeptide, est transporté hors du noyau, destiné au cytoplasme pour être traduit. Les introns peuvent être épissés différemment, ce qui entraîne l'inclusion ou l'exclusion de divers exons du produit final d'ARNm. Ce processus est connu sous le nom d'épissage alternatif. L'avantage de l'épissage alternatif est que différents types de transcrits d'ARNm peuvent être générés, tous dérivés de la même séquence d'ADN. Ces dernières années, il a été démontré que certaines archées ont également la capacité d'épisser leur pré-ARNm.
| Propriété | Des bactéries | Eucaryotes |
|---|---|---|
| Nombre de polypeptides codés par ARNm | Monocistronique ou polycistronique | Exclusivement monocistronique |
| Allongation du fil | noyau + σ = holoenzyme | ARN polymérases I, II ou III |
| Ajout d'une casquette de 5 pieds | Non | Oui |
| Ajout d'un empennage en Poly-A de 3 pieds | Non | Oui |
| Épissage du pré-ARNm | Non | Oui |
Visualisez comment se produit l'épissage de l'ARNm en regardant le processus en action dans cette vidéo. Découvrez comment les introns sont éliminés lors de l'épissage de l'ARN ici.
Exercice\(\PageIndex{2}\)
- Dans les cellules eucaryotes, comment le transcrit d'ARN d'un gène d'une protéine est-il modifié après sa transcription ?
- Les exons ou les introns contiennent-ils des informations sur les séquences protéiques ?
Orientation clinique : partie 2
Au service des urgences, une infirmière a dit à Mark qu'il avait pris la bonne décision de se rendre à l'hôpital parce que ses symptômes indiquaient une infection devenue incontrôlable. Les symptômes de Mark s'étaient aggravés, la zone de peau affectée et l'ampleur de l'enflure augmentait. Dans la zone touchée, une éruption cutanée avait commencé, des cloques et de petites poches de gaz se sont formées sous la couche externe de la peau, et une partie de la peau devenait grise. En raison de l'odeur putride du pus s'écoulant de l'une des cloques, de la progression rapide de l'infection et de l'apparence visuelle de la peau atteinte, le médecin a immédiatement commencé le traitement de la fasciite nécrosante. Le médecin de Mark a ordonné une culture du liquide s'écoulant de la plaquette thermoformée et a également ordonné des analyses sanguines, y compris une numération des globules blancs.
Mark a été admis à l'unité de soins intensifs et a commencé à administrer par voie intraveineuse un antibiotique à large spectre afin de minimiser la propagation de l'infection. Malgré l'antibiothérapie, l'état de Mark s'est rapidement détérioré. Mark est devenu confus et étourdi. Quelques heures après son admission à l'hôpital, sa tension artérielle a chuté de façon significative et sa respiration est devenue moins profonde et plus rapide. De plus, les cloques ont augmenté, leur couleur s'intensifiant pour devenir d'un noir violacé, et la blessure elle-même semblait progresser rapidement le long de la jambe de Mark.
Exercice\(\PageIndex{3}\)
- Quels sont les agents responsables possibles de la fasciite nécrosante de Mark ?
- Quelles sont les raisons possibles pour lesquelles le traitement antibiotique ne semble pas fonctionner ?
Concepts clés et résumé
- Lors de la transcription, les informations codées dans l'ADN sont utilisées pour fabriquer de l'ARN.
- L'ARN polymérase synthétise l'ARN en utilisant le brin antisens de l'ADN comme matrice en ajoutant des nucléotides d'ARN complémentaires à l'extrémité 3' du brin en croissance.
- L'ARN polymérase se lie à l'ADN au niveau d'une séquence appelée promoteur lors du début de la transcription.
- Les gènes codant pour des protéines ayant des fonctions apparentées sont fréquemment transcrits sous le contrôle d'un seul promoteur chez les procaryotes, ce qui entraîne la formation d'une molécule d'ARNm polycistronique codant pour de multiples polypeptides.
- Contrairement à l'ADN polymérase, l'ARN polymérase ne nécessite pas de groupe 3'-OH pour ajouter des nucléotides, de sorte qu'une amorce n'est pas nécessaire lors de l'initiation.
- La fin de la transcription chez les bactéries se produit lorsque l'ARN polymérase rencontre des séquences d'ADN spécifiques qui entraînent le blocage de la polymérase. Cela entraîne la libération de l'ARN polymérase à partir du brin de matrice d'ADN, libérant ainsi le transcrit d'ARN.
- Les eucaryotes possèdent trois ARN polymérases différentes. Les eucaryotes possèdent également un ARNm monocistronique, chacun ne codant qu'un seul polypeptide.
- Les transcrits primaires eucaryotes sont traités de plusieurs manières, notamment par l'ajout d'une coiffe 5' et d'une queue 3′- poly-A, ainsi que par épissage, afin de générer une molécule d'ARNm mature qui peut être transportée hors du noyau et qui est protégée contre la dégradation.


