7.5 : Utilisation de la biochimie pour identifier les microorganismes
- Page ID
- 187796
Objectifs d'apprentissage
- Décrire des exemples de produits de biosynthèse au sein d'une cellule qui peuvent être détectés pour identifier les bactéries
L'identification précise des isolats bactériens est essentielle dans un laboratoire de microbiologie clinique, car les résultats éclairent souvent les décisions relatives au traitement qui influent directement sur les résultats pour les patients. Par exemple, les cas d'intoxication alimentaire nécessitent une identification précise de l'agent causal afin que les médecins puissent prescrire un traitement approprié. De même, il est important d'identifier avec précision l'agent pathogène responsable lors d'une épidémie afin que des stratégies appropriées puissent être utilisées pour contenir l'épidémie.
Il existe de nombreuses façons de détecter, de caractériser et d'identifier les microorganismes. Certaines méthodes s'appuient sur des caractéristiques biochimiques phénotypiques, tandis que d'autres font appel à l'identification génotypique. Les caractéristiques biochimiques d'une bactérie fournissent de nombreux caractères utiles pour la classification et l'identification. L'analyse des capacités nutritionnelles et métaboliques de l'isolat bactérien est une approche courante pour déterminer le genre et l'espèce de la bactérie. Certaines des voies métaboliques les plus importantes que les bactéries utilisent pour survivre seront abordées dans Métabolisme microbien. Dans cette section, nous allons discuter de quelques méthodes qui utilisent des caractéristiques biochimiques pour identifier les microorganismes.
Certains microorganismes stockent certains composés sous forme de granulés dans leur cytoplasme, et le contenu de ces granules peut être utilisé à des fins d'identification. Par exemple, le poly-β-hydroxybutyrate (PHB) est un composé accumulateur de carbone et d'énergie présent dans certaines bactéries non fluorescentes du genre Pseudomonas. Les différentes espèces de ce genre peuvent être classées selon la présence ou l'absence de PHB et de pigments fluorescents. Le pathogène humain P. aeruginosa et le phytopathogène P. syringae sont deux exemples d'espèces fluorescentes de Pseudomonas qui n'accumulent pas de granules de PHB.
D'autres systèmes s'appuient sur des caractéristiques biochimiques pour identifier les microorganismes par leurs réactions biochimiques, telles que l'utilisation du carbone et d'autres tests métaboliques. Dans de petits laboratoires ou dans des laboratoires d'enseignement, ces essais sont réalisés à l'aide d'un nombre limité de tubes à essai. Cependant, des systèmes plus modernes, tels que celui développé par Biolog, Inc., sont basés sur des panels de réactions biochimiques effectuées simultanément et analysées par un logiciel. Le système de Biolog identifie les cellules en fonction de leur capacité à métaboliser certaines substances biochimiques et de leurs propriétés physiologiques, notamment le pH et la sensibilité chimique. Il utilise toutes les principales classes de produits biochimiques dans ses analyses. Les identifications peuvent être effectuées manuellement ou à l'aide d'instruments semi-automatisés ou entièrement automatisés.
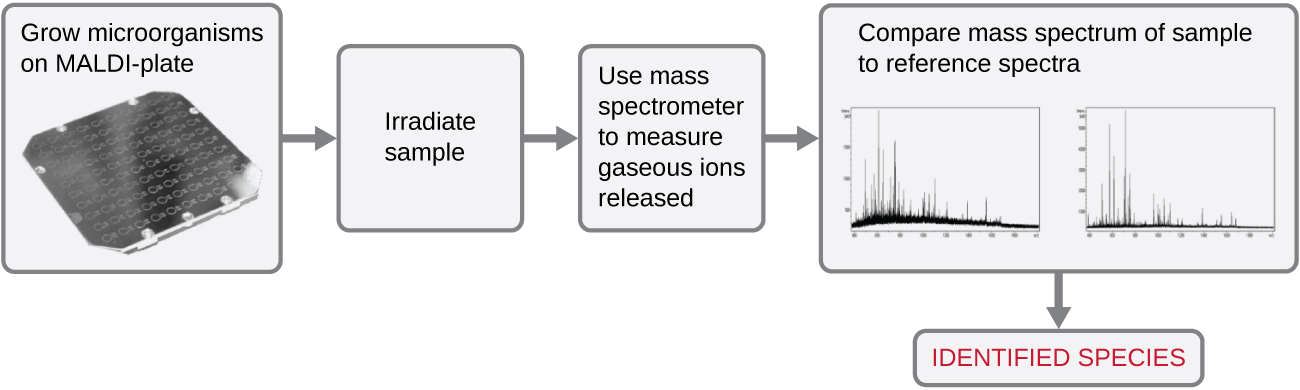
Un autre système automatisé identifie les microorganismes en déterminant le spectre de masse de l'échantillon, puis en le comparant à une base de données contenant les spectres de masse connus de milliers de microorganismes. Cette méthode est basée sur la spectrométrie de masse à temps de vol à désorption/ionisation laser assistée par matrice (MALDI-TOF) et utilise des plaques MALDI jetables sur lesquelles le microorganisme est mélangé à un réactif matriciel spécialisé (Figure\(\PageIndex{1}\)). Le mélange échantillon/réactif est irradié avec un laser ultraviolet pulsé de haute intensité, ce qui entraîne l'éjection d'ions gazeux générés par les différents constituants chimiques du microorganisme. Ces ions gazeux sont collectés et accélérés par le spectromètre de masse, les ions se déplaçant à une vitesse déterminée par leur rapport masse/charge (m/z), atteignant ainsi le détecteur à différents moments. Un diagramme du signal du détecteur par rapport à m/z donne un spectre de masse pour l'organisme qui est uniquement lié à sa composition biochimique. La comparaison du spectre de masse avec une bibliothèque de spectres de référence obtenus à partir d'analyses identiques de microorganismes connus permet d'identifier le microbe inconnu.
Les microbes peuvent également être identifiés en mesurant leurs profils lipidiques uniques. Comme nous l'avons appris, les acides gras des lipides peuvent varier en termes de longueur de chaîne, de présence ou d'absence de doubles liaisons et de nombre de doubles liaisons, de groupes hydroxyles, de branches et de cycles. Pour identifier un microbe par sa composition lipidique, les acides gras présents dans leurs membranes sont analysés. Une analyse biochimique couramment utilisée à cette fin est une technique utilisée dans les laboratoires cliniques, de santé publique et alimentaires. Elle repose sur la détection de différences uniques entre les acides gras et est appelée analyse des esters méthyliques d'acides gras (FAME). Lors d'une analyse FAME, les acides gras sont extraits des membranes de microorganismes, modifiés chimiquement pour former des esters méthyliques volatils et analysés par chromatographie en phase gazeuse (GC). Le chromatogramme GC obtenu est comparé aux chromatogrammes de référence dans une base de données contenant des données sur des milliers d'isolats bactériens afin d'identifier le microorganisme inconnu (Figure\(\PageIndex{2}\)).
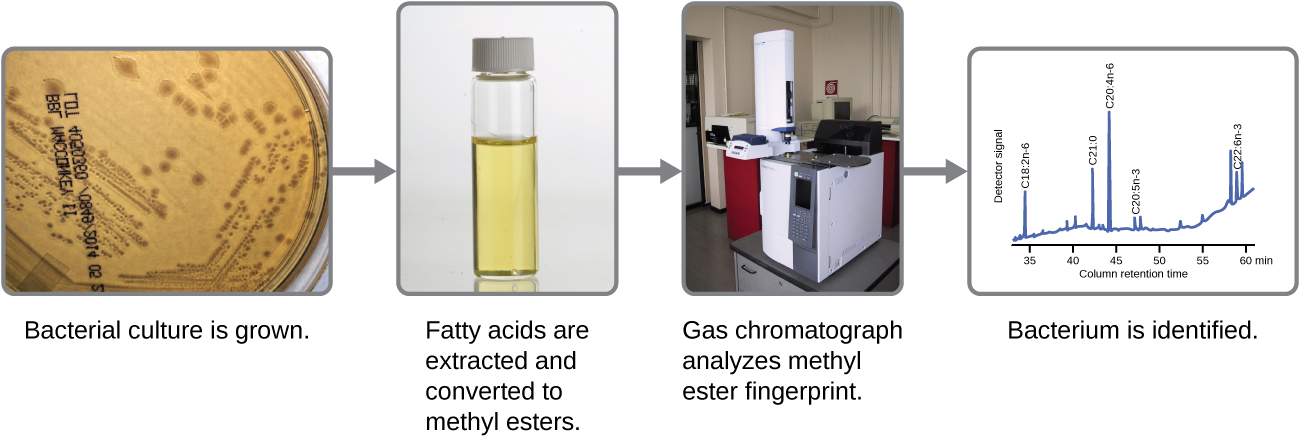
Une méthode connexe pour l'identification des microorganismes est appelée analyse des acides gras dérivés des phospholipides (PLFA). Les membranes sont principalement composées de phospholipides, qui peuvent être saponifiés (hydrolysés avec un alcali) pour libérer les acides gras. Le mélange d'acides gras obtenu est ensuite soumis à une analyse FAME, et les profils lipidiques mesurés peuvent être comparés à ceux de microorganismes connus afin d'identifier le microorganisme inconnu.
L'identification bactérienne peut également être basée sur les protéines produites dans des conditions de croissance spécifiques au sein du corps humain. Ces types de procédures d'identification sont appelés analyse protéomique. Pour effectuer une analyse protéomique, les protéines du pathogène sont d'abord séparées par chromatographie liquide à haute pression (HPLC), puis les fractions collectées sont digérées pour produire des fragments peptidiques plus petits. Ces peptides sont identifiés par spectrométrie de masse et comparés à ceux de microorganismes connus afin d'identifier le microorganisme inconnu dans l'échantillon d'origine.
Les microorganismes peuvent également être identifiés par les glucides attachés aux protéines (glycoprotéines) de la membrane plasmique ou de la paroi cellulaire. Les anticorps et autres protéines liant les glucides peuvent se fixer à des glucides spécifiques à la surface des cellules, provoquant ainsi l'agrégation des cellules. Des tests sérologiques (par exemple, les tests des groupes de Lancefield, utilisés pour identifier les espèces de streptocoques) sont effectués pour détecter les glucides uniques situés à la surface de la cellule.
Orientation clinique : résolution
Penny a cessé d'utiliser son nouvel écran solaire et a appliqué la crème corticostéroïde sur son éruption cutanée comme indiqué. Cependant, après plusieurs jours, son éruption cutanée ne s'était pas améliorée et semblait même s'aggraver. Elle a pris un rendez-vous de suivi avec son médecin, qui a observé une éruption cutanée rouge bosselée et des cloques remplies de pus autour des follicules pileux (Figure\(\PageIndex{3}\)). L'éruption cutanée était particulièrement concentrée dans les zones qui auraient été recouvertes par un maillot de bain. Après quelques questions, Penny a dit au médecin qu'elle avait récemment assisté à une fête au bord de la piscine et qu'elle avait passé du temps dans un bain à remous. À la lumière de ces nouvelles informations, le médecin a suspecté un cas d'éruption cutanée dans les spas, une infection fréquemment causée par la bactérie Pseudomonas aeruginosa, un pathogène opportuniste qui peut se développer dans les bains à remous et les piscines, en particulier lorsque l'eau n'est pas suffisamment chlorée. P. aeruginosa est la même bactérie qui est associée aux infections pulmonaires des patients atteints de mucoviscidose.
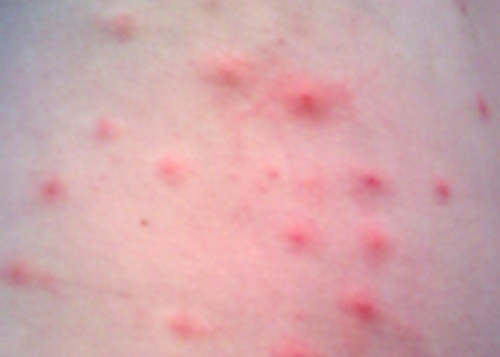
Le médecin a prélevé un échantillon de l'éruption cutanée de Penny pour l'envoyer au laboratoire de microbiologie clinique. Des tests de confirmation ont été effectués pour distinguer P. aeruginosa des agents pathogènes entériques qui peuvent également être présents dans l'eau des piscines et des cuves thermales. Le test comprenait la production de pyocyanine, un pigment bleu-vert, sur de la gélose cétrimide et la croissance à 42 °C. La cétrimide est un agent sélectif qui inhibe la croissance d'autres espèces de flore microbienne et améliore également la production de pigments de P. aeruginosa, la pyocyanine et la fluorescéine, qui sont bleu-vert et jaune-vert caractéristiques, respectivement.
Des tests ont confirmé la présence de P. aeruginosa dans l'échantillon de peau de Penny, mais le médecin a décidé de ne pas prescrire d'antibiotique. Bien que P. aeruginosa soit une bactérie, les espèces de Pseudomonas sont généralement résistantes à de nombreux antibiotiques. Heureusement, les infections cutanées comme celle de Penny disparaissent généralement d'elles-mêmes ; l'éruption cutanée dure généralement environ 2 semaines et disparaît d'elle-même, avec ou sans traitement médical. Le médecin a conseillé à Penny d'attendre la fin et de continuer à utiliser la crème corticostéroïde. La crème ne tuera pas le P. aeruginosa sur la peau de Penny, mais elle devrait calmer son éruption cutanée et minimiser les démangeaisons en supprimant la réponse inflammatoire de son corps à la bactérie.
Concepts clés et résumé
- L'identification précise des bactéries est essentielle dans un laboratoire clinique pour le diagnostic et la gestion des épidémies, des pandémies et des intoxications alimentaires causées par des épidémies bactériennes.
- L'identification phénotypique des microorganismes implique l'utilisation de traits observables, y compris des profils de composants structuraux tels que les lipides, de produits biosynthétiques tels que les sucres ou les acides aminés, ou de composés de stockage tels que le poly-β-hydroxybutyrate.
- Un microbe inconnu peut être identifié à partir du spectre de masse unique produit lors de son analyse par spectrométrie de masse à temps de vol par désorption/ionisation laser assistée par matrice (MALDI-TOF).
- Les microbes peuvent être identifiés en déterminant leur composition lipidique à l'aide d'une analyse des esters méthyliques d'acides gras (FAME) ou des acides gras dérivés des phospholipides (PLFA).
- L'analyse protéomique, c'est-à-dire l'étude de toutes les protéines accumulées dans un organisme, peut également être utilisée pour l'identification bactérienne.
- Les glycoprotéines présentes dans les structures de la membrane plasmique ou de la paroi cellulaire peuvent se lier aux lectines ou aux anticorps et peuvent être utilisées à des fins d'identification.


