9.2 : Réplication d'ADN
- Page ID
- 186306
Lorsqu'une cellule se divise, il est important que chaque cellule fille reçoive une copie identique de l'ADN. Ceci est accompli par le processus de réplication de l'ADN. La réplication de l'ADN se produit pendant la phase de synthèse, ou phase S, du cycle cellulaire, avant que la cellule n'entre en mitose ou en méiose.
L'élucidation de la structure de la double hélice a donné une idée de la manière dont l'ADN est copié. Rappelons que les nucléotides d'adénine s'associent aux nucléotides de la thymine et que la cytosine à la guanine. Cela signifie que les deux volets sont complémentaires l'un de l'autre. Par exemple, un brin d'ADN avec une séquence nucléotidique d'AGTCATGA aura un brin complémentaire avec la séquence TCAGTACT (Figure\(\PageIndex{1}\)).
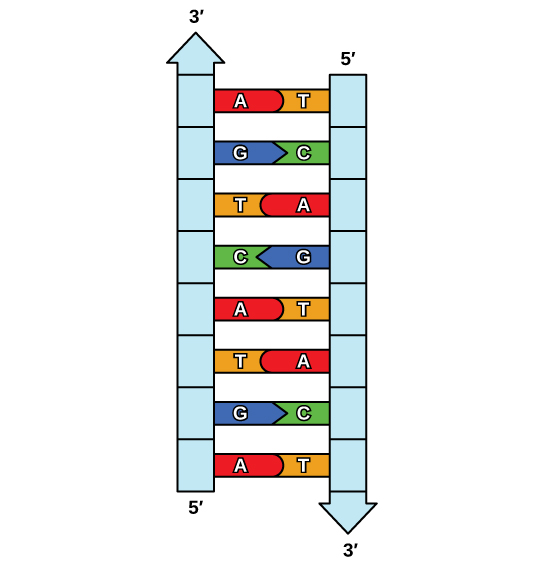
Du fait de la complémentarité des deux brins, le fait d'avoir un brin permet de recréer l'autre brin. Ce modèle de réplication suggère que les deux brins de la double hélice se séparent pendant la réplication, et que chaque brin sert de modèle à partir duquel le nouveau brin complémentaire est copié (Figure\(\PageIndex{2}\)).
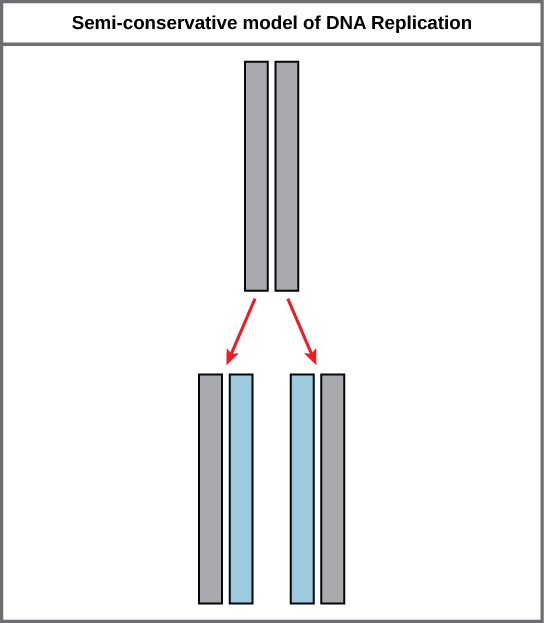
Lors de la réplication de l'ADN, chacun des deux brins qui constituent la double hélice sert de matrice à partir de laquelle les nouveaux brins sont copiés. Le nouveau volet sera complémentaire du volet parental ou « ancien » volet. Chaque nouveau volet double comprend un volet parental et un nouveau volet fille. C'est ce que l'on appelle la réplication semi-conservatrice. Lorsque deux copies d'ADN se forment, elles possèdent une séquence identique de bases nucléotidiques et sont divisées également en deux cellules filles.
Réplication d'ADN chez les eucaryotes
Les génomes des eucaryotes étant très complexes, la réplication de l'ADN est un processus très complexe qui implique plusieurs enzymes et d'autres protéines. Elle se déroule en trois étapes principales : initiation, élongation et terminaison.
Rappelons que l'ADN eucaryote est lié à des protéines appelées histones pour former des structures appelées nucléosomes. Lors de l'initiation, l'ADN est rendu accessible aux protéines et aux enzymes impliquées dans le processus de réplication. Comment la machine de réplication sait-elle où commencer la double hélice de l'ADN ? Il s'avère qu'il existe des séquences nucléotidiques spécifiques appelées origines de réplication à partir desquelles la réplication commence. Certaines protéines se lient à l'origine de la réplication tandis qu'une enzyme appelée hélicase se déroule et ouvre l'hélice de l'ADN. Lorsque l'ADN s'ouvre, des structures en forme de Y appelées fourches de réplication se forment (Figure\(\PageIndex{3}\)). Deux fourches de réplication sont formées à l'origine de la réplication et s'étendent dans les deux sens au fur et à mesure de la réplication. Il existe de multiples origines de réplication sur le chromosome eucaryote, de sorte que la réplication peut se produire simultanément à partir de plusieurs endroits du génome.
Au cours de l'élongation, une enzyme appelée ADN polymérase ajoute des nucléotides d'ADN à l'extrémité 3' de la matrice. Comme l'ADN polymérase ne peut ajouter de nouveaux nucléotides qu'à l'extrémité d'un squelette, une séquence d'amorce, qui fournit ce point de départ, est ajoutée avec des nucléotides d'ARN complémentaires. Cette amorce est retirée plus tard et les nucléotides sont remplacés par des nucléotides d'ADN. Un brin, complémentaire du brin d'ADN parental, est synthétisé en continu vers la fourche de réplication afin que la polymérase puisse ajouter des nucléotides dans cette direction. Ce brin synthétisé en continu est connu sous le nom de brin principal. Comme l'ADN polymérase ne peut synthétiser l'ADN que dans une direction de 5 à 3 pieds, l'autre nouveau brin est assemblé en petits morceaux appelés fragments d'Okazaki. Les fragments d'Okazaki nécessitent chacun une amorce constituée d'ARN pour démarrer la synthèse. Le fil contenant les fragments d'Okazaki est connu sous le nom de fil en retard. Au fur et à mesure de la synthèse, une enzyme retire l'amorce d'ARN, qui est ensuite remplacée par des nucléotides d'ADN, et les espaces entre les fragments sont scellés par une enzyme appelée ADN ligase.
Le processus de réplication de l'ADN peut être résumé comme suit :
- L'ADN se déroule à l'origine de la réplication.
- De nouvelles bases sont ajoutées aux volets parentaux complémentaires. Un nouveau fil est fabriqué en continu, tandis que l'autre brin est fabriqué en morceaux.
- Les amorces sont retirées, de nouveaux nucléotides d'ADN sont placés à la place des amorces et le squelette est scellé par de l'ADN ligase.
ART CONNECTION
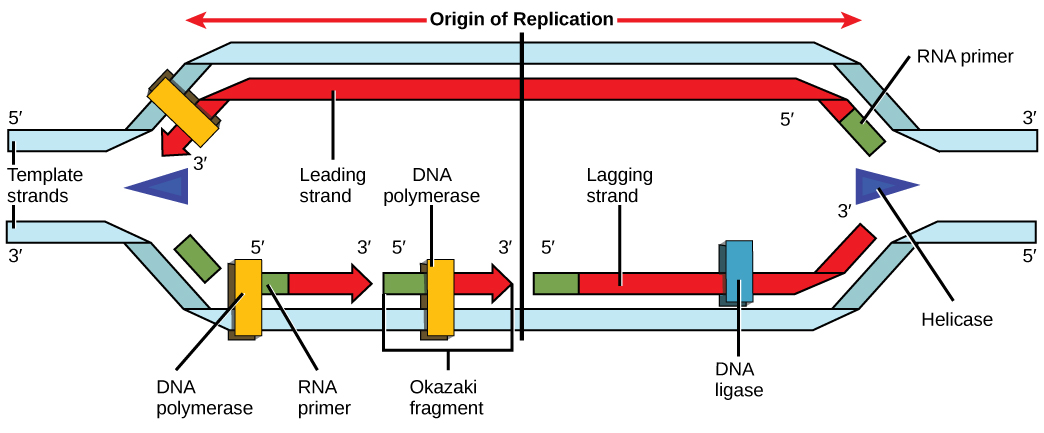
Vous isolez une souche cellulaire dans laquelle la liaison entre des fragments d'Okazaki est altérée et vous soupçonnez qu'une mutation s'est produite dans une enzyme présente au niveau de la fourche de réplication. Quelle enzyme est la plus susceptible d'être mutée ?
Réplication des télomères
Comme les chromosomes eucaryotes sont linéaires, la réplication de l'ADN se produit à l'extrémité d'une lignée dans les chromosomes eucaryotes. Comme vous l'avez appris, l'enzyme ADN polymérase ne peut ajouter des nucléotides que dans une seule direction. Dans le brin principal, la synthèse se poursuit jusqu'à ce que l'extrémité du chromosome soit atteinte ; toutefois, sur le brin en retard, il n'y a pas de place pour fabriquer une amorce pour le fragment d'ADN à copier à l'extrémité du chromosome. Cela pose un problème pour la cellule car les extrémités ne sont pas appariées et, avec le temps, ces extrémités se raccourcissent progressivement à mesure que les cellules continuent de se diviser. Les extrémités des chromosomes linéaires sont appelées télomères, qui possèdent des séquences répétitives qui ne codent pas pour un gène en particulier. Par conséquent, ce sont les télomères qui sont raccourcis à chaque cycle de réplication de l'ADN au lieu des gènes. Par exemple, chez l'homme, une séquence de six paires de bases, TTAGGG, est répétée 100 à 1 000 fois. La découverte de l'enzyme télomérase (Figure\(\PageIndex{4}\)) a permis de comprendre comment les extrémités des chromosomes sont maintenues. La télomérase se fixe à l'extrémité du chromosome et des bases complémentaires à la matrice d'ARN sont ajoutées à l'extrémité du brin d'ADN. Une fois que la matrice des brins retardataires est suffisamment allongée, l'ADN polymérase peut désormais ajouter des nucléotides complémentaires aux extrémités des chromosomes. Ainsi, les extrémités des chromosomes sont répliquées.
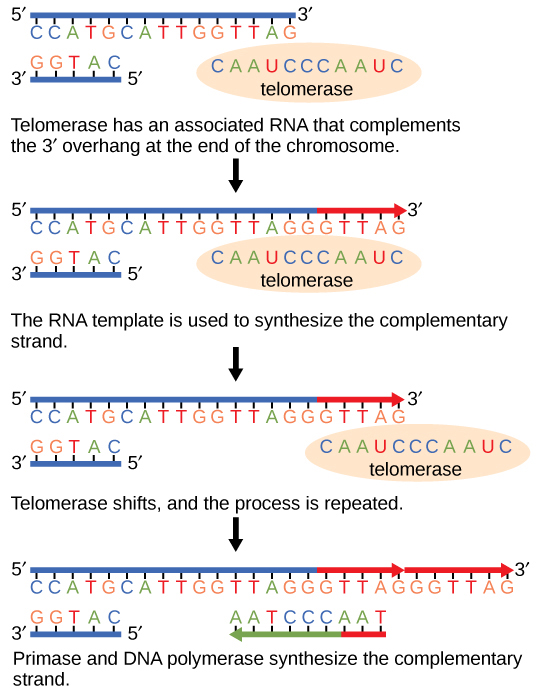
La télomérase est généralement active dans les cellules germinales, les cellules souches adultes et certaines cellules cancéreuses. Pour sa découverte de la télomérase et de son action, Elizabeth Blackburn (Figure\(\PageIndex{5}\)) a reçu le prix Nobel de médecine et de physiologie en 2009.

La télomérase n'est pas active dans les cellules somatiques adultes. Les cellules somatiques adultes qui subissent une division cellulaire continuent de voir leurs télomères raccourcis. Cela signifie essentiellement que le raccourcissement des télomères est associé au vieillissement. En 2010, des scientifiques ont découvert que la télomérase pouvait inverser certaines affections liées à l'âge chez la souris, ce qui pourrait avoir un potentiel en médecine régénérative. 1 Des souris déficientes en télomérase ont été utilisées dans ces études ; ces souris présentent une atrophie tissulaire, une déplétion des cellules souches, une défaillance du système organique et une altération de la réponse aux lésions tissulaires. La réactivation de la télomérase chez ces souris a entraîné l'extension des télomères, réduit les dommages à l'ADN, inversé la neurodégénérescence et amélioré le fonctionnement des testicules, de la rate et des intestins. Ainsi, la réactivation des télomères peut potentiellement traiter les maladies liées à l'âge chez les humains.
Réplication d'ADN chez les procaryotes
Rappelons que le chromosome procaryote est une molécule circulaire dont la structure enroulée est moins étendue que les chromosomes eucaryotes. Le chromosome eucaryote est linéaire et fortement enroulé autour des protéines. Bien qu'il existe de nombreuses similitudes dans le processus de réplication de l'ADN, ces différences structurales nécessitent certaines différences dans le processus de réplication de l'ADN chez ces deux formes de vie.
La réplication de l'ADN a été extrêmement bien étudiée chez les procaryotes, principalement en raison de la petite taille du génome et du grand nombre de variants disponibles. Escherichia coli possède 4,6 millions de paires de bases dans un seul chromosome circulaire, et toutes se répliquent en 42 minutes environ, en commençant par une seule origine de réplication et en faisant le tour du chromosome dans les deux sens. Cela signifie qu'environ 1000 nucléotides sont ajoutés par seconde. Le processus est beaucoup plus rapide que chez les eucaryotes. \(\PageIndex{1}\)Le tableau résume les différences entre les réplications procaryotes et eucaryotes.
| Propriété | Procaryotes | Eucaryotes |
|---|---|---|
| Origine de la réplication | Unique | Plusieurs |
| Taux de réplication | 1000 nucléotides/s | 50 à 100 nucléotides/s |
| Structure des chromosomes | circulaire | linéaire |
| Télomérase | Non présent | Présent |
CONCEPT EN ACTION
Cliquez sur un didacticiel sur la réplication de l'ADN.
Réparation d'ADN
L'ADN polymérase peut commettre des erreurs lors de l'ajout de nucléotides. Il édite l'ADN en relisant chaque base nouvellement ajoutée. Les bases incorrectes sont éliminées et remplacées par la base correcte, puis la polymérisation se poursuit (Figure\(\PageIndex{6}\) a). La plupart des erreurs sont corrigées lors de la réplication, mais lorsque cela ne se produit pas, le mécanisme de réparation des mésappariements est utilisé. Les enzymes de réparation des mésappariements reconnaissent la base mal incorporée et l'extraient de l'ADN, la remplaçant par la base correcte (Figure\(\PageIndex{6}\) b). Dans un autre type de réparation, la réparation par excision nucléotidique, l'ADN double brin est déroulé et séparé, les bases incorrectes sont éliminées ainsi que quelques bases aux extrémités 5' et 3', et celles-ci sont remplacées par une copie de la matrice à l'aide de l'ADN polymérase (Figure\(\PageIndex{6}\) c). La réparation par excision des nucléotides est particulièrement importante pour corriger les dimères de thymine, qui sont principalement causés par la lumière ultraviolette. Dans un dimère de thymine, deux nucléotides de thymine adjacents l'un à l'autre sur un brin sont liés l'un à l'autre par covalence plutôt que leurs bases complémentaires. Si le dimère n'est pas retiré et réparé, cela entraînera une mutation. Les personnes présentant des défauts dans leurs gènes de réparation par excision nucléotidique sont extrêmement sensibles au soleil et développent des cancers de la peau très tôt dans leur vie.
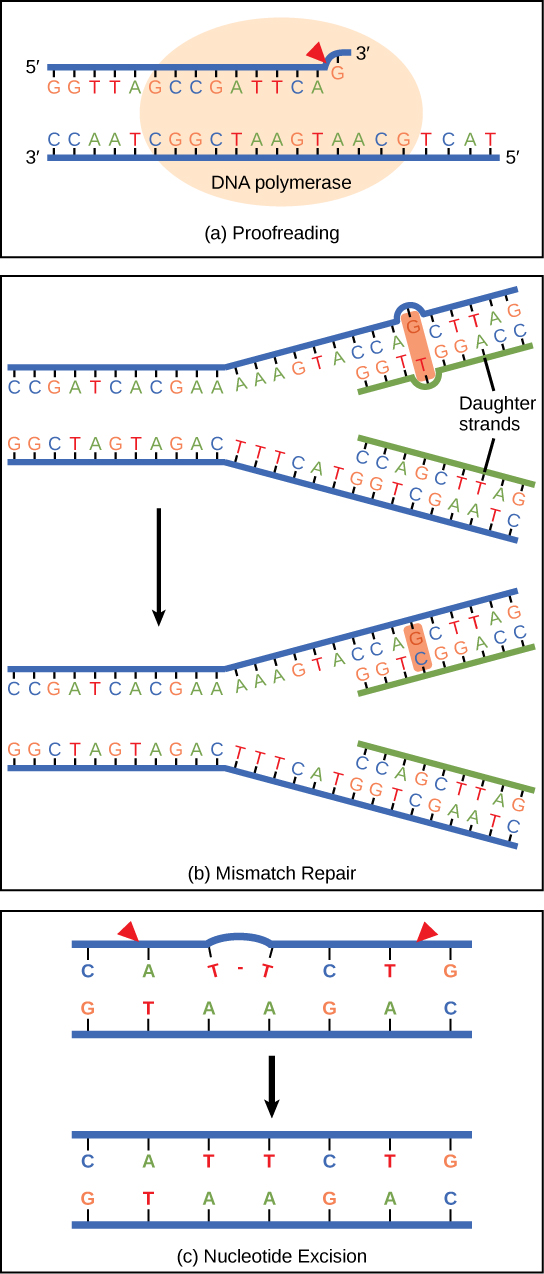
La plupart des erreurs sont corrigées ; si ce n'est pas le cas, elles peuvent entraîner une mutation, définie comme une modification permanente de la séquence d'ADN. Les mutations des gènes réparateurs peuvent avoir de graves conséquences, comme le cancer.
Résumé
L'ADN se réplique par une méthode semi-conservatrice dans laquelle chacun des deux brins d'ADN parental agit comme matrice pour le nouvel ADN à synthétiser. Après réplication, chaque ADN possède un brin parental ou « ancien » et un brin fille ou « nouveau » brin.
La réplication chez les eucaryotes commence à partir de plusieurs origines de réplication, tandis que la réplication chez les procaryotes commence à partir d'une seule origine de réplication. L'ADN est ouvert avec des enzymes, ce qui entraîne la formation de la fourche de réplication. La primase synthétise une amorce d'ARN pour initier la synthèse par l'ADN polymérase, qui ne peut ajouter des nucléotides que dans une seule direction. Un brin est synthétisé en continu en direction de la fourche de réplication ; c'est ce que l'on appelle le brin principal. L'autre brin est synthétisé dans une direction opposée à la fourche de réplication, dans de courtes portions d'ADN appelées fragments d'Okazaki. Ce brin est connu sous le nom de brin en retard. Une fois la réplication terminée, les amorces d'ARN sont remplacées par des nucléotides d'ADN et l'ADN est scellé avec de l'ADN ligase.
Les extrémités des chromosomes eucaryotes posent problème, car la polymérase est incapable de les étendre sans amorce. La télomérase, une enzyme dotée d'une matrice d'ARN intégrée, étend les extrémités en copiant la matrice d'ARN et en étendant une extrémité du chromosome. L'ADN polymérase peut ensuite étendre l'ADN à l'aide de l'amorce. De cette façon, les extrémités des chromosomes sont protégées. Les cellules disposent de mécanismes pour réparer l'ADN lorsqu'il est endommagé ou que des erreurs sont commises lors de la réplication. Ces mécanismes incluent la réparation des mésappariements pour remplacer les nucléotides associés à une base non complémentaire et la réparation par excision des nucléotides, qui élimine les bases endommagées telles que les dimères de thymine.
Connexions artistiques
Figure\(\PageIndex{3}\) : Vous isolez une souche cellulaire dans laquelle la liaison entre des fragments d'okazaki est altérée et vous soupçonnez qu'une mutation s'est produite dans une enzyme présente au niveau de la fourche de réplication. Quelle enzyme est la plus susceptible d'être mutée ?
- Réponse
-
Ligase, car cette enzyme réunit les fragments d'Okazaki.
Notes
- 1 Mariella Jaskelioff, et al., « La réactivation de la télomérase inverse la dégénérescence tissulaire chez les souris âgées déficientes en télomérase », Nature, 469 (2011) :102—7.
Lexique
- ADN ligase
- l'enzyme qui catalyse l'assemblage de fragments d'ADN
- ADN polymérase
- une enzyme qui synthétise un nouveau brin d'ADN complémentaire à un brin matrice
- hélicase
- une enzyme qui aide à ouvrir l'hélice de l'ADN lors de la réplication de l'ADN en rompant les liaisons hydrogène
- brin en retard
- lors de la réplication du brin de 3' à 5', le brin qui est répliqué en fragments courts et loin de la fourchette de réplication
- brin principal
- le brin synthétisé en continu dans la direction 5' à 3' qui est synthétisé dans la direction de la fourche de réplication
- réparation des discordances
- une forme de réparation de l'ADN dans laquelle les nucléotides non complémentaires sont reconnus, excisés et remplacés par des nucléotides corrects
- mutation
- une variation permanente de la séquence nucléotidique d'un génome
- réparation par excision nucléotidique
- une forme de réparation de l'ADN dans laquelle la molécule d'ADN est déroulée et séparée dans la région endommagée par le nucléotide, les nucléotides endommagés sont retirés et remplacés par de nouveaux nucléotides à l'aide du brin complémentaire, et le brin d'ADN est rescellé et laissé rejoindre son complément
- Fragments d'Okazaki
- les fragments d'ADN qui sont synthétisés sur de courtes distances sur le brin en retard
- apprêt
- un court segment de nucléotides d'ARN qui est nécessaire pour initier la réplication et permettre à l'ADN polymérase de se lier et de commencer la réplication
- fourche de réplication
- la structure en Y formée lors du début de la réplication
- réplication semi-conservatrice
- la méthode utilisée pour répliquer l'ADN dans laquelle la molécule double brin est séparée et où chaque brin sert de matrice pour un nouveau brin à synthétiser, de sorte que les molécules d'ADN résultantes sont composées d'un nouveau brin de nucléotides et d'un ancien brin de nucléotides
- télomérase
- une enzyme qui contient une partie catalytique et une matrice d'ARN intégrée ; elle a pour fonction de maintenir les télomères aux extrémités des chromosomes
- télomère
- l'ADN à l'extrémité des chromosomes linéaires


