12.2: 可视化和表征 DNA
- Page ID
- 200475
学习目标
- 解释使用核酸探针可视化特定 DNA 序列的方法
- 解释使用凝胶电泳分离 DNA 片段的方法
- 解释限制片段长度多态性分析的原理及其用途
- 比较和对比南方和北方印迹
- 解释微阵列分析的原理和用途
- 描述用于分离和可视化蛋白质变异的方法
- 解释聚合酶链反应和DNA测序的方法和用途
与数据库中的已知序列相比,DNA分子的序列可以帮助我们识别生物体。 该序列还可以告诉我们一些有关DNA特定部分功能的信息,例如它是否编码特定的蛋白质。 比较样本之间的蛋白质特征(特定蛋白质阵列的表达水平)是评估细胞对多种环境因素和压力的反应的重要方法。 蛋白质特征分析可以揭示生物体的身份或细胞在疾病期间的反应。
感兴趣的DNA和蛋白质是微观的,通常与许多其他分子混合在一起,包括与我们的兴趣无关的DNA或蛋白质。 已经开发出许多技术来分离和表征感兴趣的分子。 这些方法最初是为研究目的而开发的,但在许多情况下,它们已被简化到可以进行常规临床使用的程度。 例如,许多病原体,例如导致胃溃疡的幽门螺杆菌,可以使用基于蛋白质的测试来检测。 此外,越来越多的基于DNA扩增的高特异性和准确的鉴定分析现在可以检测病原体,例如抗生素耐药性肠道细菌、单纯疱疹病毒、水痘带状疱疹病毒等。
DNA 的分子分析
在本小节中,我们将概述一些用于分离和可视化科学家感兴趣的特定DNA片段的基本方法。 其中一些方法不需要了解 DNA 分子的完整序列。 在快速DNA测序问世之前,这些方法是唯一可用于处理DNA的方法,但它们仍然是分子遗传学家用来研究人体对微生物和其他疾病反应的基本工具库。
核酸探测
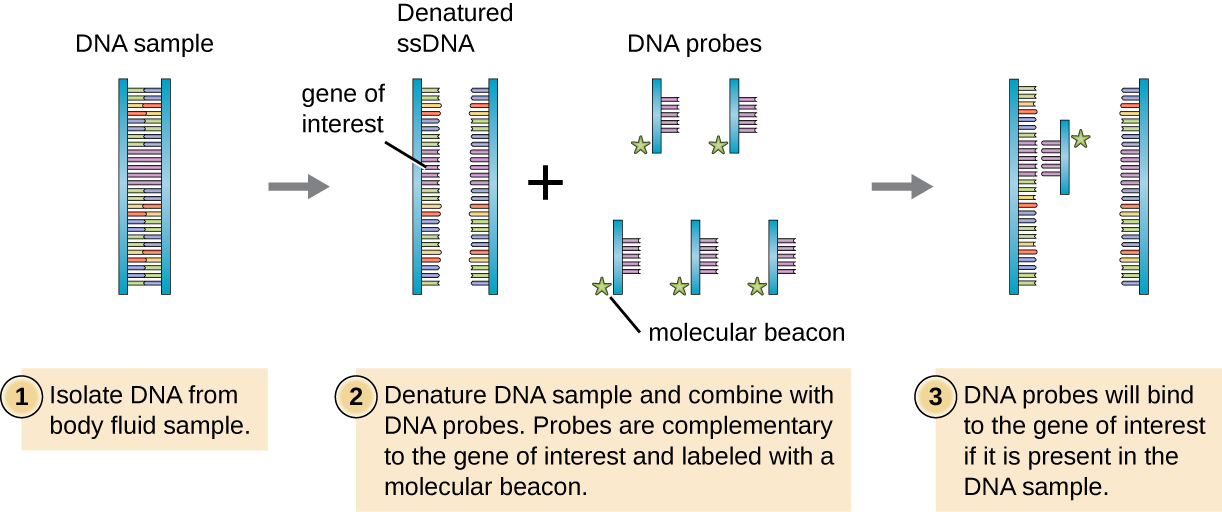
DNA 分子很小,其序列中包含的信息是不可见的。 研究人员如何分离出特定的DNA片段或将其分离出来,确定它来自哪个生物,其序列是什么,或者其功能是什么? 鉴定某个 DNA 序列存在的一种方法是使用人工构造的 DNA 片段,称为探针。 探针可用于识别环境中的不同细菌种类,现在有许多 DNA 探针可用于临床检测病原体。 例如,DNA探针用于检测阴道病原体白色念珠菌、阴道加德纳菌和阴道滴虫。
要在基因组库中筛选特定的感兴趣基因或序列,研究人员必须对该基因有所了解。 如果研究人员拥有相关基因的 DNA 序列的一部分,他们就可以设计一个 DNA 探针,一种单链 DNA 片段,它与目标基因的一部分互补,不同于样本中的其他 DNA 序列。 DNA探针可以由商业实验室以化学方式合成,也可以通过克隆、分离和变性来自活生物体的DNA片段来创建。 无论哪种情况,DNA 探针都必须用分子标签或信标进行标记,例如放射性磷原子(用于放射自显影)或荧光染料(用于荧光原位杂交或 FISH),以便可以看到探针及其结合的 DNA(图\(\PageIndex{1}\))。 正在探测的DNA样本还必须变性以使其成为单链,这样单链DNA探针就可以在其序列互补的位置与单链DNA样本退火。 尽管这些技术对诊断很有价值,但由于这些样本的复杂性,直接用于痰液和其他身体样本可能会出现问题。 通常必须首先通过化学提取方法从身体样本中分离出DNA,然后才能使用DNA探针识别病原体。
临床重点:第 2 部分
凯拉出现的类似流感的轻微症状可能是由任意数量的传染病原体引起的。 此外,几种非传染性自身免疫性疾病,例如多发性硬化症、系统性红斑狼疮(SLE)和肌萎缩性侧索硬化症(ALS),其症状也与凯拉的早期症状一致。 但是,在几周的时间里,凯拉的症状恶化了。 她的膝盖开始出现关节疼痛、心悸,面部肌肉出现奇怪的跛行。 此外,她脖子僵硬,头痛痛。 她很不情愿,决定是时候寻求医疗救助了。
练习\(\PageIndex{1}\)
- 凯拉的新症状能否为她可能患有哪种类型的感染或其他疾病提供任何线索?
- 医疗保健提供者可以使用哪些测试或工具来查明导致凯拉症状的病原体?
琼脂糖凝胶电泳
在许多情况下,研究人员可能希望对不同大小的DNA片段进行物理分离。 研究人员还可以使用限制酶消化 DNA 样本以形成片段。 由此产生的大小和片段分布模式通常可以产生有关DNA碱基序列的有用信息,这些信息可用来识别DNA所属的个体或物种,就像条形码扫描一样。
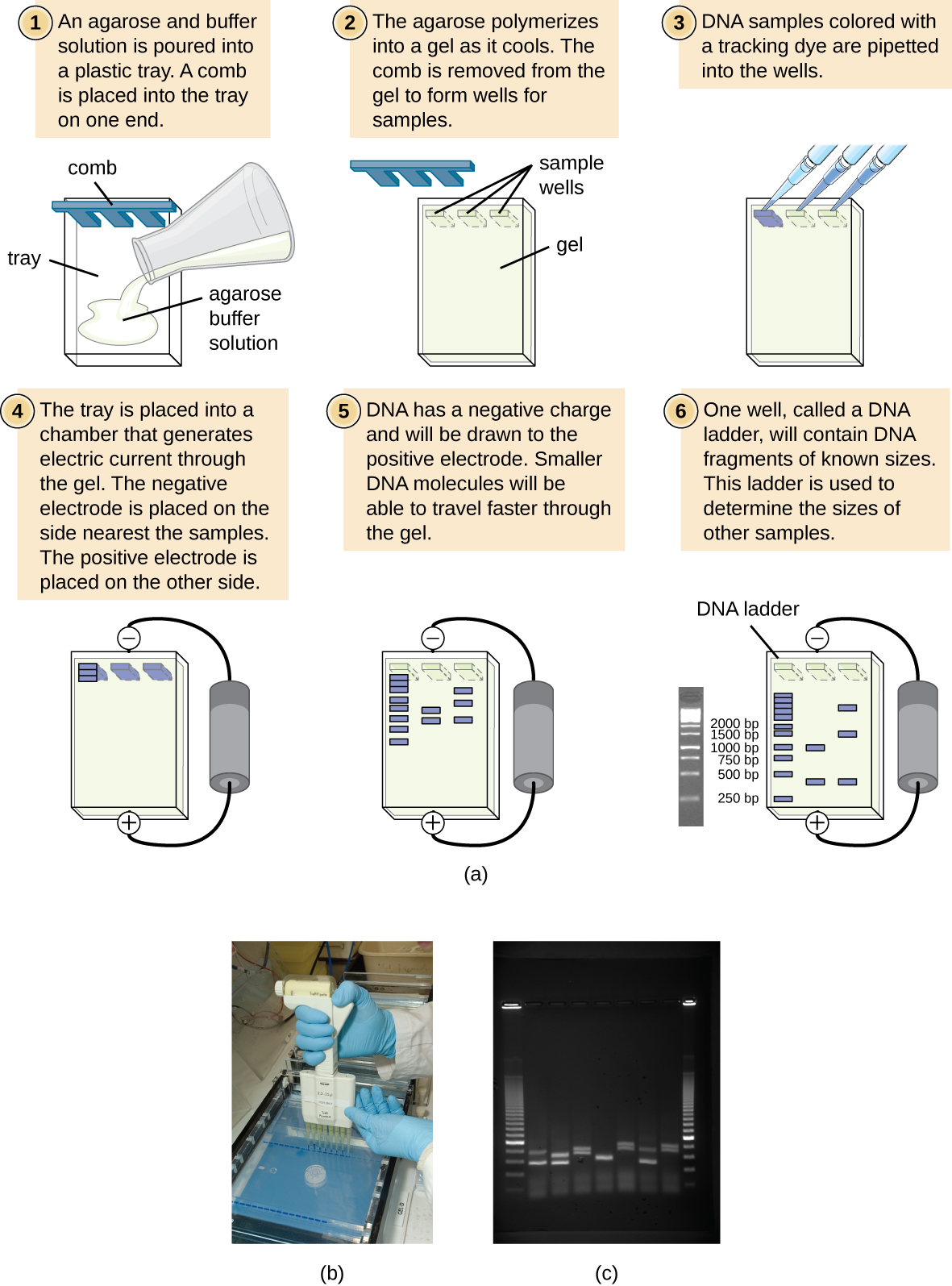
凝胶电泳是一种常用于根据大小和生化特性(例如电荷和极性)分离生物分子的技术。 琼脂糖凝胶电泳广泛用于分离不同大小的DNA(或RNA),这些DNA(或RNA)可能是通过限制性酶消化或其他方式(例如聚合酶链反应)产生的(图\(\PageIndex{2}\))。
由于其带负电荷的骨干,DNA被正电极强烈吸引。 在琼脂糖凝胶电泳中,凝胶在缓冲溶液中水平定向。 样品被装入最靠近负电极的凝胶一侧的样品孔中,然后通过琼脂糖基质的分子筛向正电极抽取。 琼脂糖基质阻碍较大分子在凝胶中的移动,而较小的分子更容易通过。 因此,迁移距离与DNA片段的大小成反比,较小的片段在凝胶中传播的距离更长。 样本中DNA片段的大小可以通过与同样在同一凝胶上运行的DNA梯中已知大小的片段进行比较来估算。 为了分离非常大的DNA片段,例如染色体或病毒基因组,可以在脉冲场凝胶电泳(PFGE)期间通过定期交替电场的方向来修改琼脂糖凝胶电泳。 在 PFGE 中,较小的碎片可以调整自身方向,迁移速度比较大的碎片稍快一些,因此这种技术可以分离非常大的碎片,否则这些碎片在标准琼脂糖凝胶电泳过程中会一起传播。 在任何一种电泳技术中,都可以通过各种方法检测凝胶中DNA或RNA片段的位置。 一种常用的方法是添加溴化乙钠,这种染色剂可在非特异性部位插入核酸,暴露在紫外线下时可以看到。 其他比潜在致癌物质溴化乙钠更安全的污渍现已上市。
限制性片段长度多态性 (RFLP) 分析
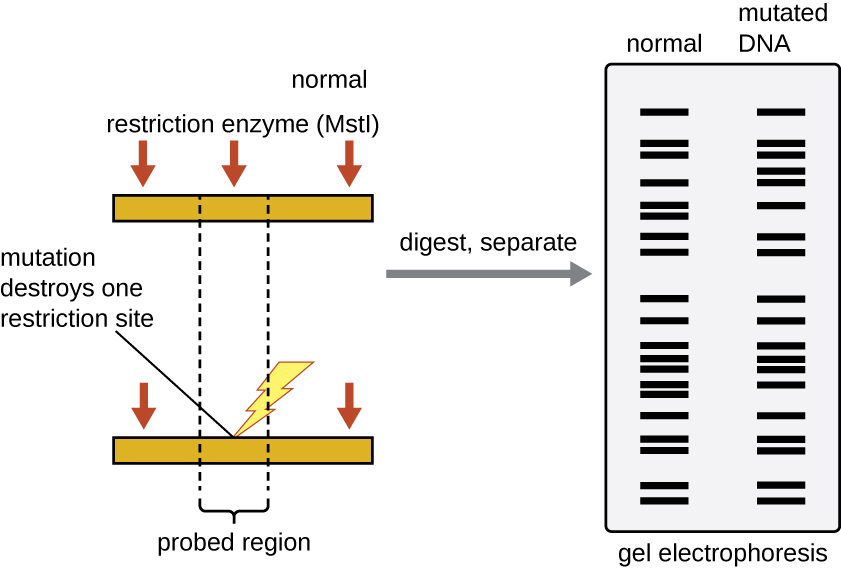
限制性酶识别位点很短(只有几个核苷酸长),是序列特异性回文,可以在整个基因组中找到。 因此,个体基因组中DNA序列的差异将导致限制酶识别位点分布的差异,这些位点可以在琼脂糖凝胶电泳后将其可视化为凝胶上的不同带状图案。 限制性片段长度多态性(RFLP)分析比较了限制性消化后不同 DNA 样本的 DNA 带状模式(图\(\PageIndex{3}\))。
RFLP 分析在医学和法医学中有许多实际应用。 例如,流行病学家使用 RFLP 分析来跟踪和识别与食物中毒或某些传染病疫情有关的特定微生物的来源。 RFLP 分析还可用于人类 DNA,以确定具有变异基因(包括与遗传性疾病相关的染色体)的遗传模式或建立亲子关系。
法医科学家使用 RFLP 分析作为 DNA 指纹识别的一种形式,可用于分析从犯罪现场、嫌疑人和受害者获得的 DNA。 采集 DNA 样本,使用聚合酶链反应增加样本 DNA 分子的拷贝数,然后进行限制性酶消化和琼脂糖凝胶电泳以生成特定的带状图案。 通过将从犯罪现场采集的样本的条带模式与从嫌疑人或受害者那里采集的样本进行比较,调查人员可以明确确定在现场收集的DNA证据是嫌疑人还是受害者留下的。
南方印迹和修改
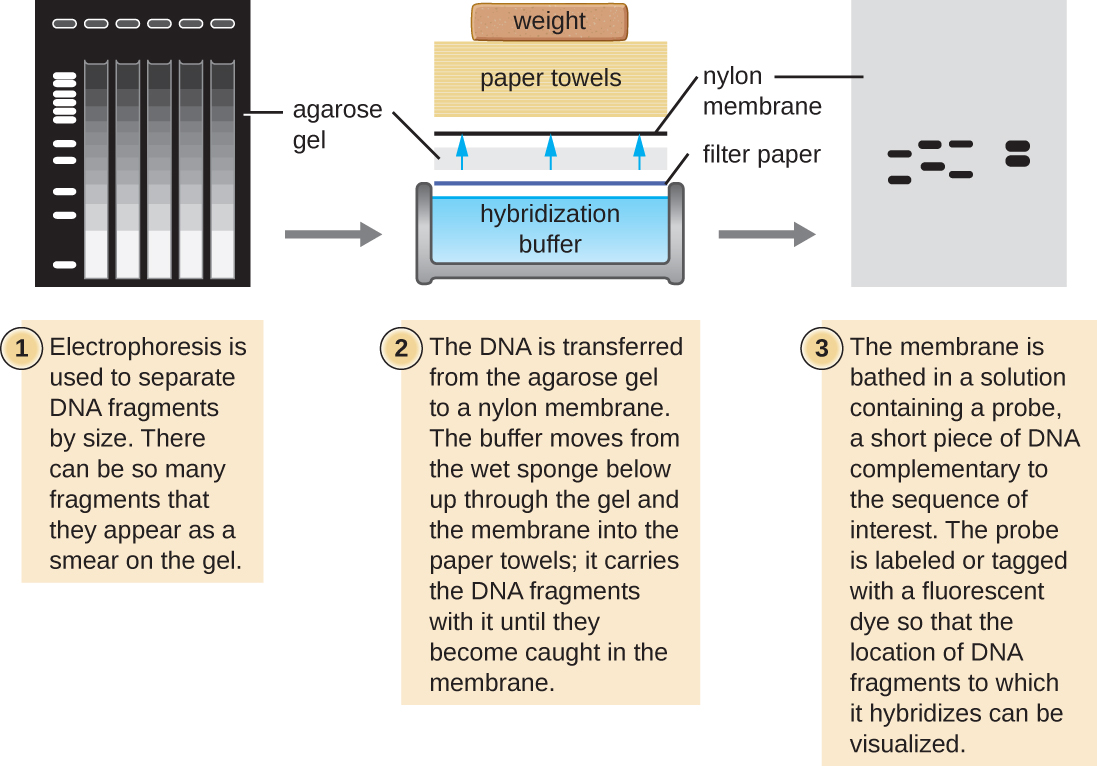
几种分子技术利用了样品核酸与 DNA 探针之间的序列互补性和杂交。 通常,探测凝胶中的核酸样本不成功,因为当 DNA 探针浸入凝胶中时,凝胶中的样本核酸会扩散出来。 因此,印迹技术通常用于将核酸转移到由硝化纤维素或尼龙制成的带正电荷的薄膜上。 在埃德温·南方爵士于1975年开发的南方印迹技术中,样本中的DNA片段首先通过琼脂糖凝胶电泳分离,然后通过毛细管作用转移到膜中(图\(\PageIndex{4}\))。 然后,将与膜表面结合的 DNA 片段暴露于标有放射性或荧光分子信标的特定单链 DNA 探针中,以帮助检测。 南方印迹可用于检测给定的 DNA 样本中是否存在某些 DNA 序列。 一旦膜内的靶DNA被可视化,研究人员就可以切出膜中含有该片段的部分,以恢复感兴趣的DNA片段。
Southern blot 的变异——点状印迹、缝隙印迹和斑点印迹——不涉及电泳,而是将样本中的 DNA 浓缩到膜上的一个小位置。 与DNA探针杂交后,测量检测到的信号强度,从而使研究人员能够估计样本中存在的靶DNA的数量。
菌落印迹是南方印迹的另一种变体,其中代表基因组库中不同克隆的菌落通过将膜压在培养板上转移到膜上。 膜上的细胞被裂解,然后可以对膜进行探测,以确定基因组库中的哪些菌落含有靶基因。 由于板块上的菌落仍在生长,因此可以将感兴趣的细胞从板块中分离出来。
在南方印迹的另一种变异北方印迹中,RNA(不是DNA)被固定在膜上并进行探测。 Northern blots 通常用于检测组织或生物样本中通过基因表达产生的 mRNA 量。
微阵列分析
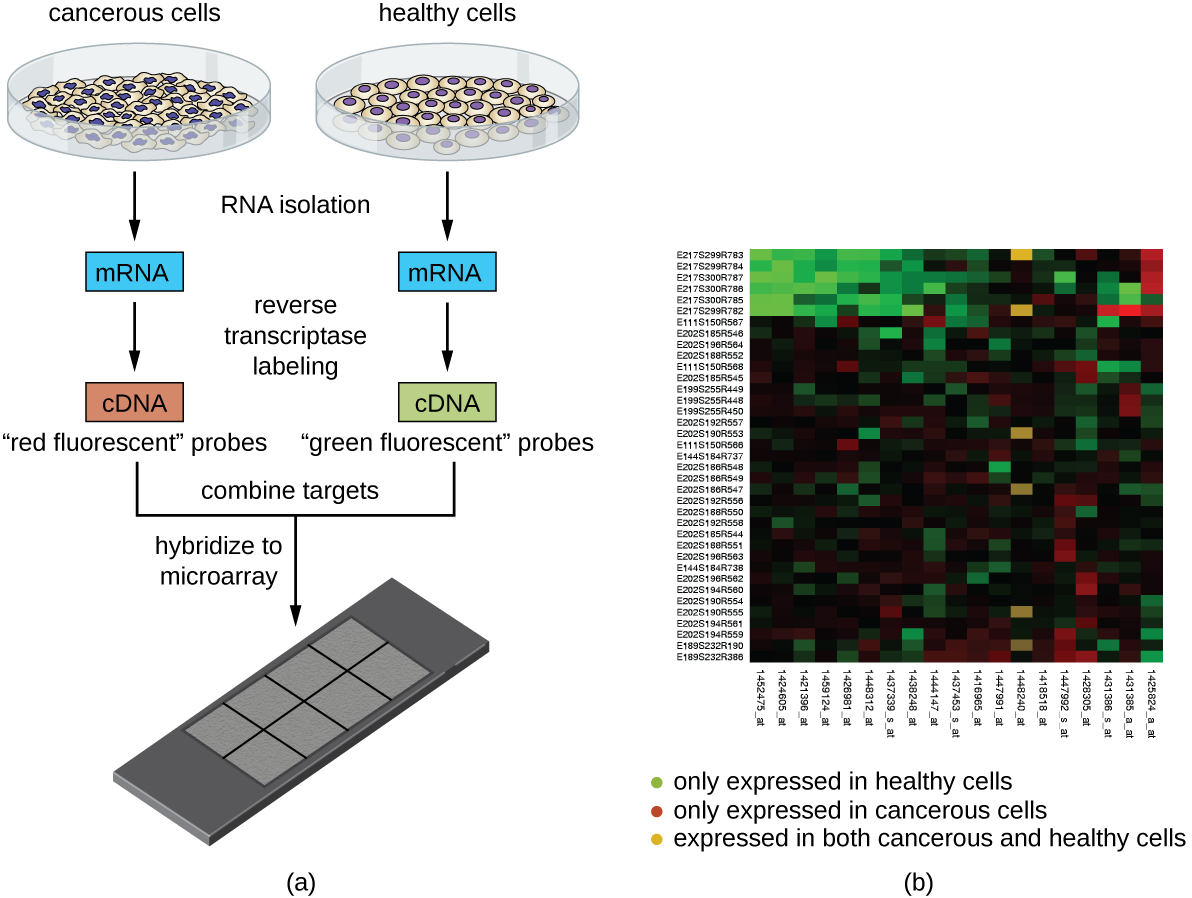
另一种利用互补核酸序列杂交的技术称为微阵列分析。 微阵列分析可用于比较不同细胞类型之间的基因表达模式,例如,感染病毒的细胞与未感染的细胞,或者癌细胞与健康细胞(图\(\PageIndex{5}\))。
通常,来自实验样本的 DNA 或 cDNA 与已知的 DNA 序列一起沉积在玻璃载玻片上。 每张幻灯片可容纳 30,000 多种不同的 DNA 片段类型。 可以在玻璃载玻片上单独发现不同的DNA片段(包括生物体的整个基因组库)或cDNA片段(对应于生物体表达基因的完整补体)。
一旦沉积在载玻片上,就可以从两个样本中分离出基因组DNA或mRNA进行比较。 如果分离出mRNA,则使用逆转录酶将其逆转录为cDNA。 然后用不同的荧光染料(通常是红色和绿色)标记两个基因组DNA或cDNA样本。 然后将经过标记的基因组DNA样本按等量合并,添加到微阵列芯片中,并允许杂交到微阵列上的互补点。
样本基因组DNA分子的杂交可以通过测量微阵列上特定点的荧光强度来监测。 可以很容易地观察到样品之间杂交量的差异。 如果只有一个样品的核酸与微阵列上的特定点杂交,则该点将显示为绿色或红色。 但是,如果两个样品的核酸杂交,则由于红色和绿色染料的组合,该斑点将显示为黄色。
尽管微阵列技术允许在短时间内对两个样品进行整体比较,但它需要复杂(且昂贵的)检测设备和分析软件。 由于费用昂贵,这项技术通常仅限于研究环境。 研究人员使用微阵列分析来研究被细菌或病毒感染或接受某些化学处理的生物体中的基因表达是如何受到影响的。
在这个互动网站上探索微芯片技术。
练习\(\PageIndex{2}\)
- DNA 探针由什么组成?
- 为什么在对 DNA 消化物进行凝胶电泳后使用南方印迹?
蛋白质的分子分析
在许多情况下,直接研究 DNA 或 RNA 可能不可取或不可能。 蛋白质可以提供用于鉴定的特定物种信息,也可以提供有关细胞或组织如何以及是否对致病微生物的存在做出反应的重要信息。 不同的蛋白质需要不同的分离和表征方法。
聚丙烯酰胺凝胶电泳
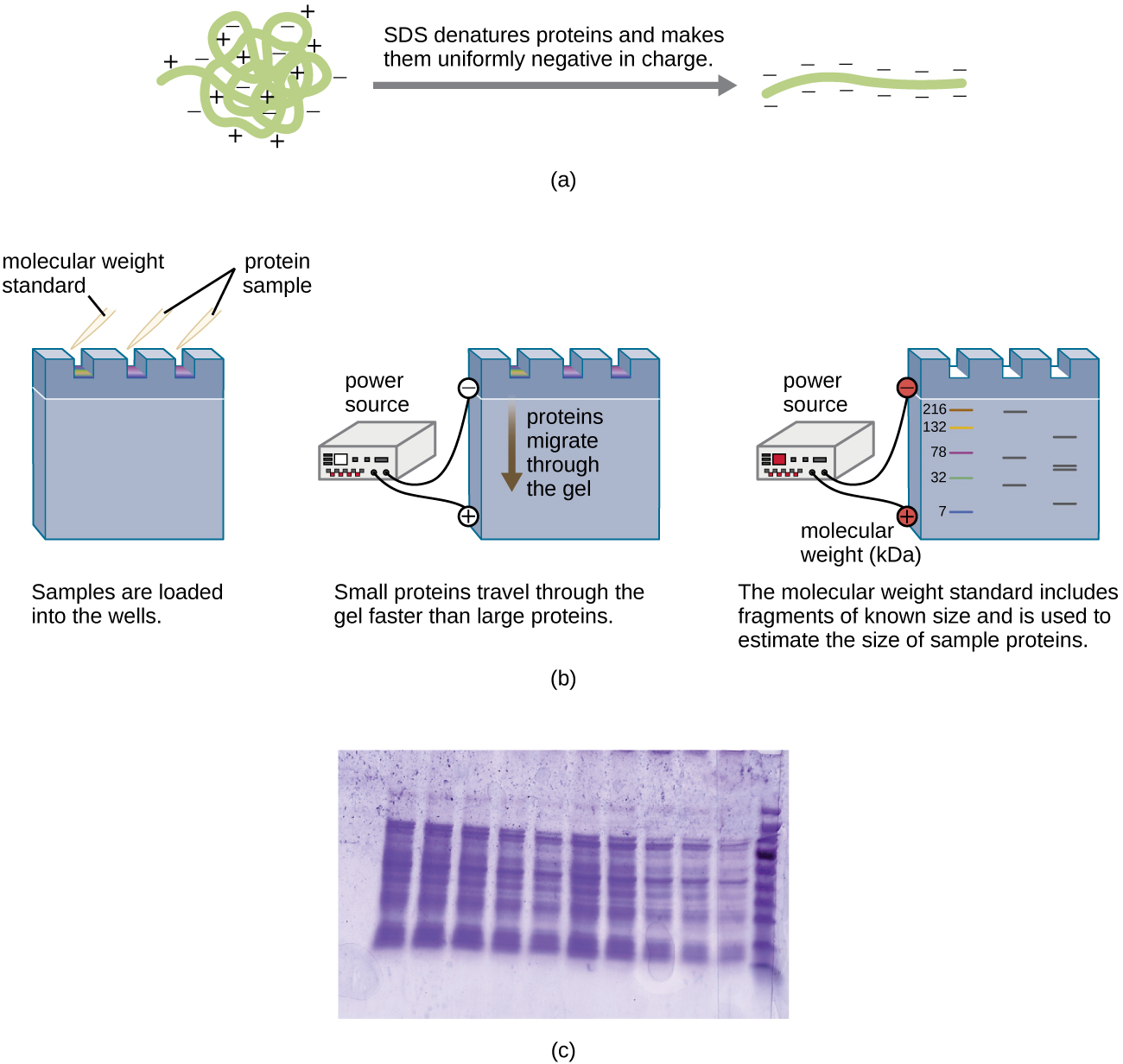
凝胶电泳的一种变体,称为聚丙烯酰胺凝胶电泳(PAGE),通常用于分离蛋白质。 在 PAGE 中,凝胶基质更精细,由聚丙烯酰胺而不是琼脂糖组成。 此外,PAGE 通常使用垂直凝胶设备进行(图\(\PageIndex{6}\))。 由于与氨基酸侧链相关的电荷各不相同,PAGE 可用于根据其净电荷分离完整蛋白质。 或者,蛋白质可以变性并涂上一种称为十二烷基硫酸钠(SDS)的带负电荷的洗涤剂,从而掩盖天然电荷并允许仅根据大小进行分离。 通过使用二维 PAGE,可以进一步修改 PAGE,根据两个特征(例如它们在不同 pH 值下的电荷以及它们的大小)分离蛋白质。 在任何一种情况下,在电泳之后,蛋白质都会通过染色进行可视化,通常使用库马西蓝色或银色染色。
练习\(\PageIndex{3}\)
在SDS-PAGE中分离蛋白质的依据是什么?
临床重点:第 3 部分
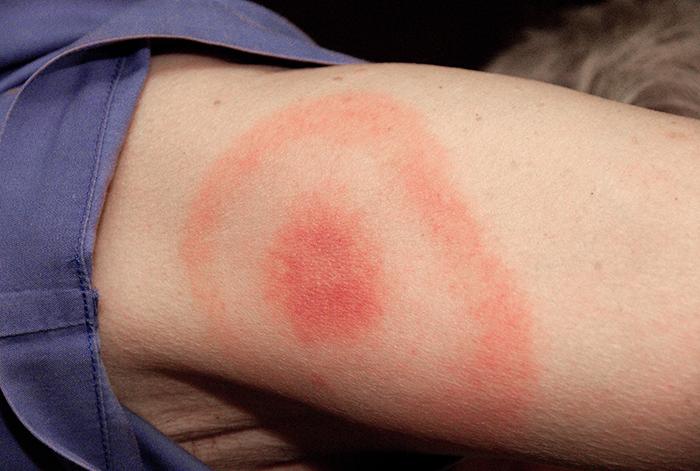
当凯拉描述她的症状时,她的医生起初怀疑是细菌性脑膜炎,这与她的头痛和脖子僵硬相符。 但是,她很快排除了这种可能性,因为脑膜炎的发展速度通常比凯拉所经历的要快。 她的许多症状仍然与肌萎缩性侧索硬化症(ALS)和系统性红斑狼疮(SLE)相似,考虑到凯拉在树林里待了多少时间,医生还认为莱姆病是一种可能性。 凯拉不记得最近有过蜱虫叮咬(莱姆病的典型传播方式),也没有出现与莱姆病相关的典型靶心疹(图\(\PageIndex{7}\))。 但是,20-30%的莱姆病患者从未出现过这种皮疹,因此医生不想排除这种情况。
凯拉的医生下令对她的大脑进行核磁共振检查,进行全血细胞计数以检测贫血,进行血液检查以评估肝脏和肾脏功能,并进行其他检查以确认或排除系统性红斑狼疮或莱姆病。 她的测试结果与系统性红斑狼疮和肌萎缩性侧索硬化症均不一致,寻找莱姆病抗体的测试结果 “模棱两可”,这意味着没有定论。 在排除了肌萎缩性侧索硬化症和系统性红斑狼疮之后,凯拉的医生决定对莱姆病进行额外的检查。
练习\(\PageIndex{4}\)
- 即使检测结果没有检测到血液中的莱姆抗体,为什么凯拉的医生还会怀疑莱姆病?
- 哪种类型的分子测试可用于检测莱姆病的血液抗体?
基于扩增的 DNA 分析方法
可以使用各种方法来获取 DNA 序列,这对于研究致病生物很有用。 随着快速测序技术的出现,我们对致病生物整个基因组的知识库显著增长。 我们首先描述聚合酶链反应,这不是一种测序方法,而是使研究人员和临床医生能够获得测序和其他研究所需的大量DNA。 聚合酶链反应消除了我们过去对细胞制作多个 DNA 拷贝的依赖,通过细胞外相对简单的反应得出了相同的结果。
聚合酶链反应 (PCR)
大多数DNA分析方法,例如限制酶消化和琼脂糖凝胶电泳或DNA测序,都需要大量的特定的DNA片段。 过去,大量的DNA是通过培育基因组文库的宿主细胞产生的。 但是,文库需要花费时间和精力来制备,感兴趣的 DNA 样本通常数量很少。 聚合酶链反应 (PCR) 允许快速扩增特定 DNA 序列的拷贝数量,以供进一步分析(图\(\PageIndex{8}\))。 聚合酶链反应是分子生物学中最强大的技术之一,由 Kary Mullis 于 1983 年在 Cetus Corporation 任职期间开发。 聚合酶链反应在研究、法医和临床实验室中有特定的应用,包括:
- 测定 DNA 特定区域中核苷酸的序列
- 扩增 DNA 的靶区以便克隆到质粒载体中
- 确定留在犯罪现场的 DNA 样本的来源
- 分析样本以确定亲子关系
- 将古代 DNA 样本与现代生物进行比较
- 确定人体或环境样本中是否存在难以培养或无法培养的微生物
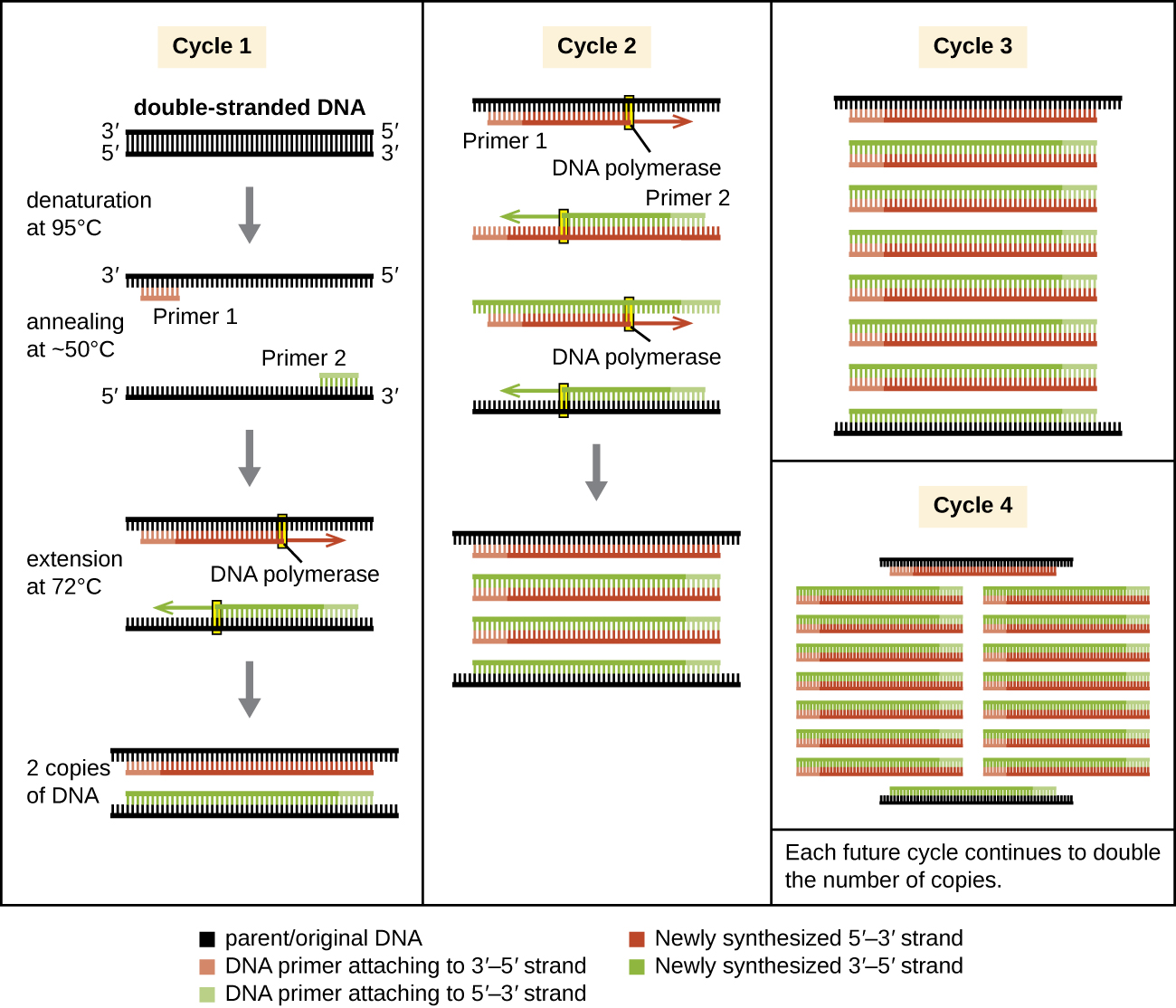
聚合酶链反应是一种体外实验室技术,它利用了 DNA 复制的自然过程。 聚合酶链反应中使用的热稳定性 DNA 聚合酶来自超嗜热原核生物。 Taq DNA 聚合酶通常用于聚合酶链反应,源自从黄石国家公园温泉中分离出来的 Thermus aquaticus 细菌。 DNA 复制需要使用引物开始复制,以获得游离的 3-羟基基团,以便通过 DNA 聚合酶添加核苷酸。 但是,虽然由RNA组成的引物通常用于细胞,但DNA引物用于聚合酶链反应。 由于其稳定性,DNA引物更可取,具有已知序列的靶向特定 DNA 区域的 DNA 引物可以通过商业方式进行化学合成。 这些DNA引物在功能上与用于前面描述的各种杂交技术的DNA探针相似,由于靶DNA序列和引物之间的互补性,它们与特定靶点结合。
聚合酶链反应分为多个周期,每个周期包含三个步骤:变性、退火和延伸。 称为热循环仪的机器用于聚合酶链反应;可以对这些机器进行编程,使其在每个步骤所需的温度下自动循环(图 12.1)。 首先,含有靶序列的双链模板 DNA 在大约 95 °C 下变性。物理(而不是酶法)分离 DNA 链所需的高温是需要热稳定的 DNA 聚合酶的原因。 接下来,温度降低到大约 50 °C。这允许与靶序列末端互补的 DNA 引物退火(粘附)到模板链,每条链上有一条引物退火。 最后,将温度提高到72°C,这是保持热稳定DNA聚合酶活性的最佳温度,允许使用单链靶标作为模板在引物中添加核苷酸。 每个周期都会使双链靶DNA拷贝的数量翻一番。 通常,聚合酶链反应方案包括25—40个周期,允许将单个靶序列扩增数千万至超过一万亿。
自然 DNA 复制旨在复制整个基因组,并在一个或多个起源位点启动。 引物是在复制期间构造的,而不是在复制之前,并且不由几个特定的序列组成。 聚合酶链反应使用序列特异性引物靶向 DNA 样本的特定区域。 近年来,利用有助于 DNA 复制过程的辅助蛋白,开发了各种规避热循环需求的等温聚合酶链反应扩增方法。 随着这些方法的不断发展及其在研究、法医和临床实验室中的使用变得越来越普遍,热循环仪可能会过时。
PCR 变异
后来对聚合酶链反应的几次修改进一步提高了这种技术的效用。 逆转录酶聚合酶链反应(RT-PCR)用于获取特定 mRNA 分子的 DNA 拷贝。 逆转录聚合酶链反应从使用逆转录酶将 mRNA 分子转化为 cDNA 开始。 然后,该cDNA被用作传统聚合酶链反应扩增的模板。 逆转录聚合酶链反应可以检测样本中是否表达了特定基因。 聚合酶链反应最近的另一个应用是实时聚合酶链反应,也称为定量聚合酶链反应(qPCR)。 标准聚合酶链反应和逆转录聚合酶链反应方案不是定量的,因为在方案中的所有周期完成之前,任何一种试剂都可能受到限制,并且样本只能在最后进行分析。 由于无法确定在聚合酶链反应或逆转录聚合酶链反应方案中给定试剂何时受到限制,因此无法知道在此之前完成了多少次循环,因此无法确定样品中存在多少原始模板分子PCR。 但是,在 qPCR 中,荧光的使用允许人们在聚合酶链反应过程中监测双链模板的增加。 然后,这些动力学数据可用于量化原始目标序列的数量。 近年来,qPCR的使用进一步扩大了聚合酶链反应的能力,使研究人员能够确定样本中存在的DNA拷贝数,有时还包括生物的数量。 在临床环境中,qrt-PCR 用于测定 HIV 阳性患者的病毒载量,以评估其治疗的有效性。
DNA 测序
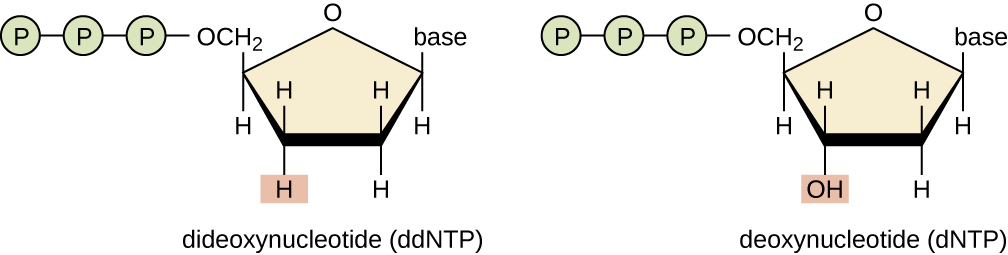
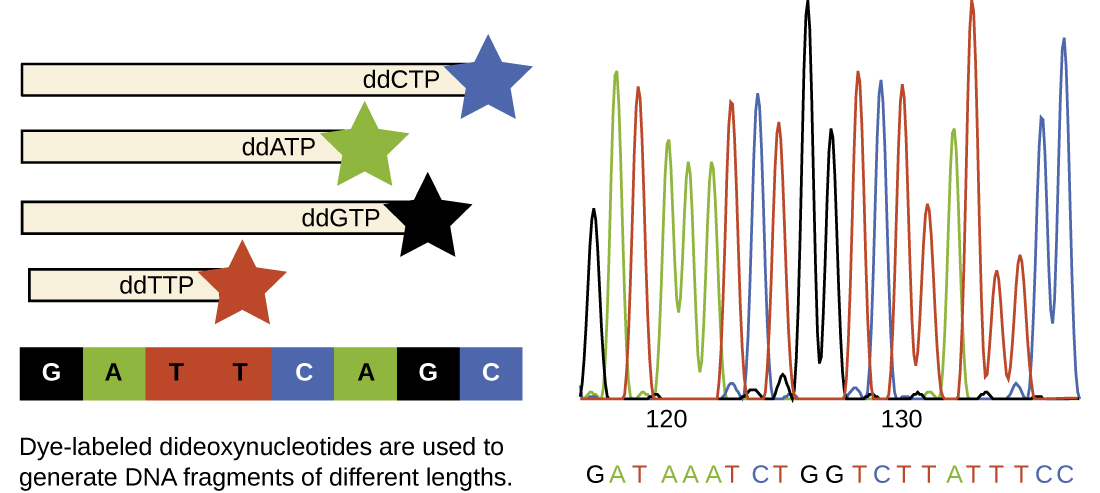
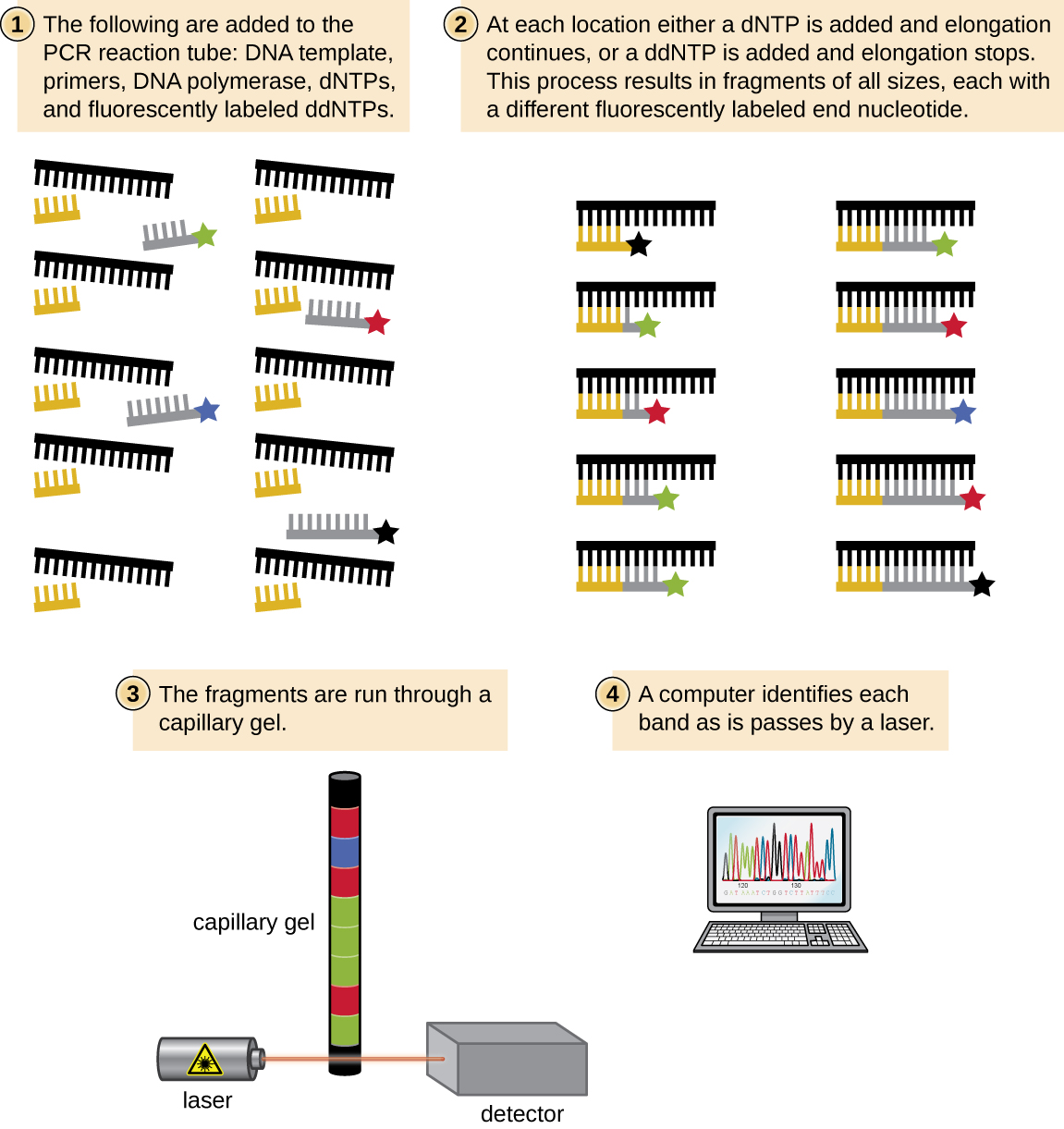
一种基本的测序技术是链终止方法,也称为二脱氧法或桑格DNA测序方法,由弗雷德里克·桑格在1972年开发。 链终止方法包括使用DNA引物对单链模板进行DNA复制,以启动互补链、DNA聚合酶、四种常规脱氧核苷酸(dnTP)的混合物和少量双脱氧核苷酸(ddNTP)的合成,每种都标有分子信标。 DDNTP 是缺少羟基 (—OH) 的单体,在另一个核苷酸通常附着形成链的部位(图\(\PageIndex{9}\))。 每当 ddNTP 被随机掺入不断增长的互补链中时,它就会终止该特定链的 DNA 复制过程。 这会导致多条短链的复制 DNA,每条链在复制过程中的不同点终止。 当对反应混合物进行凝胶电泳时,多条新复制的DNA链形成不同大小的梯子。 由于 ddNTP 是经过标记的,因此 ddnTP 终止反应时,凝胶上的每条带都会反映 DNTP 链的大小。
在桑格时代,为每个正在测序的DNA分子设置了四个反应,每个反应只包含四种可能的DDNTP中的一个。 每个 ddnTP 都标有放射性磷分子。 然后,四种反应的产物在长而窄的 PAGE 凝胶上并排在不同的通道中,通过放射自显影检测到不同长度的波段。 如今,在一次测序反应中使用 ddNTP(每种都标有不同颜色的荧光染料或荧光色素\(\PageIndex{10}\)),该过程已得到简化,每个测序反应中包含每个正在测序的 DNTP 的所有四种可能的 ddNTP(图\(\PageIndex{11}\))。 这些荧光色素是通过荧光光谱法检测出来的。 确定每个波段经过探测器时的荧光颜色会生成模板链的核苷酸序列。
自2005年以来,实验室使用的自动测序技术属于下一代测序的范畴,下一代测序是一组用于快速DNA测序的自动化技术。 这些方法彻底改变了分子遗传学领域,因为低成本测序器可以在一天之内生成成千上万或数百万个短片段(25 到 600 个碱基对)的序列。 尽管不同公司开发了几种下一代测序技术的变体(例如454 Life Sciences的焦磷酸测序和Illumina的Solexa技术),但它们都允许对数百万个碱基进行快速测序,从而使整个基因组的测序相对容易、便宜,而且司空见惯。 例如,在454测序(焦磷酸测序)中,DNA样本被分成400—600-bp的单链片段,并在每个片段的两端添加DNA适配器进行修改。 然后,每个 DNA 片段都被固定在珠子上,通过聚合酶链反应进行扩增,使用设计用于与适配器退火的引物,形成一个含有该 DNA 片段的许多拷贝的珠子。 然后将每个珠子放入一个单独的含有测序酶的井中。 在井中,四个核苷酸中的每一个都一个接一个地添加;当每一个核苷酸被掺入时,焦磷酸作为聚合的副产物被释放,发出一小闪光,由探测器记录。 这提供了生成新的 DNA 链时加入的核苷酸的顺序,也是合成测序的一个例子。 下一代测序器使用复杂的软件来完成整理所有片段的繁琐过程。 总体而言,这些技术继续快速发展,降低了测序成本,并迅速提高了来自各种生物的序列数据的可用性。
国家生物技术信息中心拥有一个名为GenBank的广泛使用的遗传序列数据库,研究人员在其中存放遗传信息供公众使用。 序列数据发布后,研究人员将其上传到GenBank,从而使其他研究人员可以访问该信息。 此次合作使研究人员能够将新发现或未知的样本序列信息与已经存在的大量序列数据进行比较。
观看有关 454 测序的动画,加深您对这种方法的理解。
使用 NAAT 诊断艰难梭菌感染
哈维尔是一名有心脏病史的80岁患者,在接受血管成形术将支架插入心动脉后,最近从医院返回家中。 为了最大限度地减少感染的可能性,哈维尔在手术期间和手术后不久接受了静脉注射广谱抗生素。 他在手术四天后获释,但一周后,他开始每天出现几次轻度腹绞痛和水样腹泻。 他食欲不振,严重脱水,发烧。 他还注意到他的粪便中有血。 哈维尔的妻子打电话给医生,医生指示她立即带他去急诊室。
医院工作人员进行了几次检查,发现与血液中的水平相比,哈维尔的肾肌酐水平升高,这表明他的肾脏功能不佳。 哈维尔的症状表明可能感染了艰难梭菌,这种细菌对许多抗生素具有耐药性。 医院收集并培养了一个粪便样本,以寻找艰难梭菌产生的毒素A和B,但结果为阴性。 但是,阴性结果不足以排除艰难梭菌感染,因为艰难梭菌的培养和检测其特征毒素可能很困难,尤其是在某些类型的样本中。 为了安全起见,他们进行了诊断性核酸扩增测试(NAAT)。 目前,NAAT是临床诊断师检测病原体遗传物质的黄金标准。 就哈维尔而言,qPCR 被用来寻找编码艰难梭菌毒素 B(tcd B)的基因。 当 qPCR 分析结果呈阳性时,主治医生得出结论,哈维尔确实患有艰难梭菌感染,因此立即开了抗生素万古霉素处方,静脉注射。 抗生素清除了感染,哈维尔完全康复。
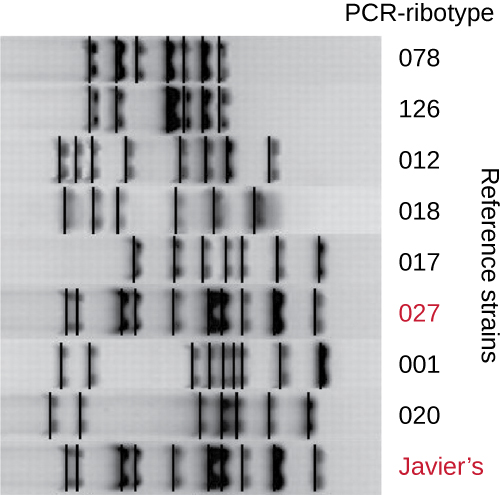
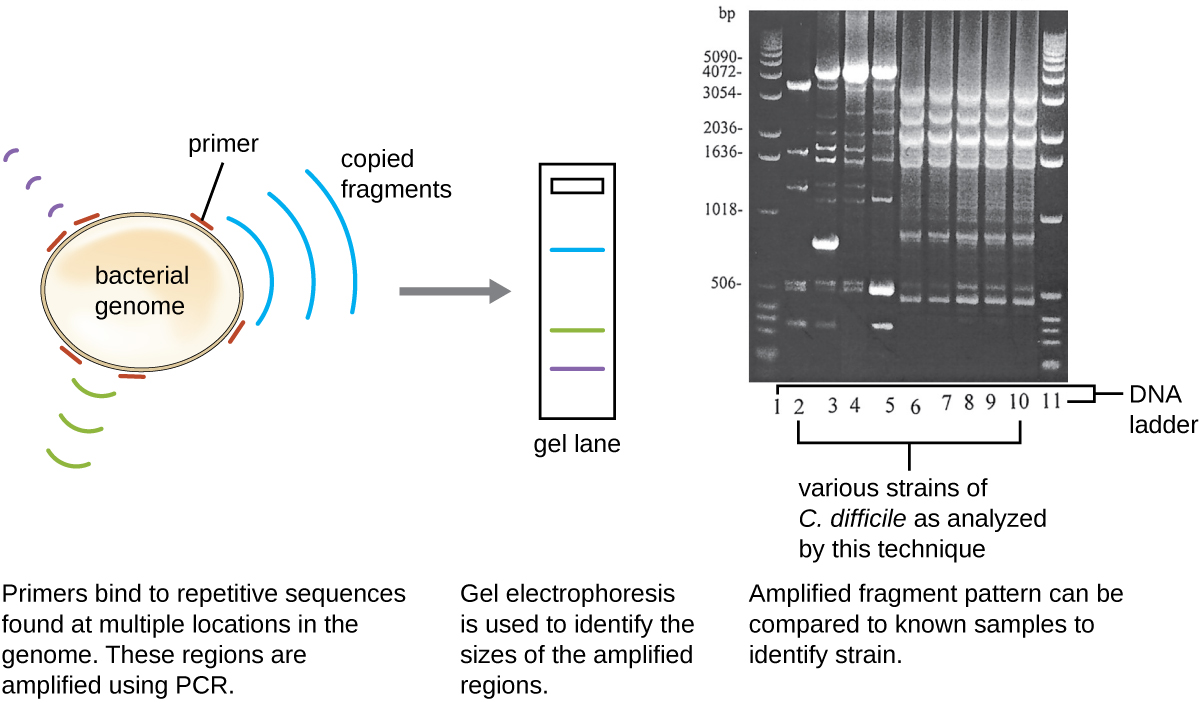
由于艰难梭菌感染在哈维尔社区越来越普遍,因此对他的样本进行了进一步分析,以确定是否可以识别出艰难梭菌的特定菌株。 对哈维尔的粪便样本进行了核糖分型和基于序列的重复聚合酶链反应(rep-PCR)分析。 在核糖分型中,16S rRNA和23S rRNA基因之间的短DNA序列被扩增并受到限制消化(图\(\PageIndex{12}\))。 这个序列因艰难梭菌菌株而异,因此限制酶会在不同的地方切割。 在rep-PCR中,设计用于结合艰难梭菌基因组中常见的重复短序列的DNA引物用于聚合酶链反应。 限制消化后,在这两种类型的分析中都进行了琼脂糖凝胶电泳,以检查每种手术产生的带状模式(图\(\PageIndex{13}\))。 rep-PCR 可用于进一步对各种核型进行亚型,从而提高检测菌株之间差异的分辨率。 感染哈维尔的菌株的核型被发现为核型27,该菌株以其毒力增加、抗生素耐药性增加以及美国、加拿大、日本和欧洲流行率上升而闻名。 1
练习\(\PageIndex{5}\)
- 艰难梭菌菌株之间的带状模式有何不同?
- 你为什么认为实验室测试无法直接检测毒素的产生?
练习\(\PageIndex{6}\)
- 聚合酶链反应与细胞中的自然 DNA 复制过程有何相似之处? 有什么不同?
- 根据逆转录聚合酶链反应和定量聚合酶链反应各自的用途进行比较。
- 在链终止测序中,序列中每个核苷酸的身份是如何确定的?
关键概念和摘要
- 在样本中寻找感兴趣的基因需要使用标有分子信标(通常是放射性或荧光)的单链 DNA 探针,该探针可以与样本中的互补单链核酸杂交。
- 琼脂糖凝胶电泳允许根据大小分离 DNA 分子。
- 限制性片段长度多态性 (RFLP) 分析允许通过琼脂糖凝胶电泳可视化由限制位点差异引起的 DNA 序列的不同变体。
- 南方印迹分析允许研究人员在样本中找到特定的DNA序列,而北方印迹分析允许研究人员检测样本中表达的特定mRNA序列。
- 微阵列技术是一种核酸杂交技术,它允许一次检查数千个基因,以发现两个基因组DNA或cDNA样本之间的基因或基因表达模式的差异,
- 聚丙烯酰胺凝胶电泳(PAGE)允许按大小分离蛋白质,尤其是在使用 SDS 预处理掩盖天然蛋白质电荷的情况下。
- 聚合酶链反应允许快速扩增特定的DNA序列。 聚合酶链反应变体可用于检测 mRNA 的表达(逆转录酶 PCR)或量化原始样本中的特定序列(实时聚合酶链反应)。
- 尽管桑格DNA测序的开发是革命性的,但下一代测序的进步允许对许多生物的基因组进行快速而廉价的测序,从而加快了新序列数据的数量。
脚注
- 1 Patrizia Spigaglia、Fabrizio Barbanti、Anna Maria Dionisi 和 Paola Mastrantonio。 “意大利对氟喹诺酮类抗药的艰难梭菌分离物:聚合酶链反应核型018的出现。” 《临床微生物学杂志》第 4 8 期第 8 期(2010):2892—2896。


