15.5: ריבוזומים וסינתזת חלבונים
- Page ID
- 205947
מיומנויות לפיתוח
- תאר את השלבים השונים בסינתזת החלבון
- דון בתפקיד הריבוזומים בסינתזת חלבון
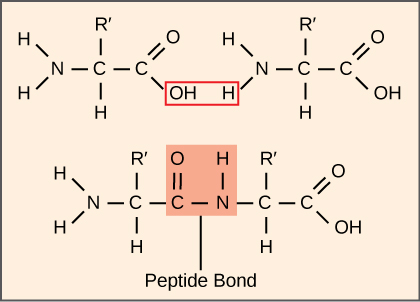
סינתזה של חלבונים צורכת יותר אנרגיה של התא מכל תהליך מטבולי אחר. בתורו, חלבונים מהווים יותר מסה מכל מרכיב אחר של אורגניזמים חיים (למעט מים), וחלבונים מבצעים כמעט כל פונקציה של תא. תהליך התרגום, או סינתזת החלבון, כולל פענוח של הודעת mRNA למוצר פוליפפטיד. חומצות אמינו נמתחות יחד באופן קוולנטי על ידי קישור קשרי פפטיד באורכים הנעים בין כ-50 שאריות חומצות אמינו ליותר מ-1,000. לכל חומצת אמינו בודדת יש קבוצת אמינו (NH 2) וקבוצת קרבוקסיל (COOH). פוליפפטידים נוצרים כאשר קבוצת האמינו של חומצת אמינו אחת יוצרת קשר אמיד (כלומר פפטיד) עם קבוצת הקרבוקסיל של חומצת אמינו אחרת (איור). \(\PageIndex{1}\) תגובה זו מזורזת על ידי ריבוזומים ומייצרת מולקולת מים אחת.
מכונות סינתזת החלבון
בנוסף לתבנית ה- mRNA, מולקולות ומקרומולקולות רבות תורמות לתהליך התרגום. ההרכב של כל רכיב עשוי להשתנות בין המינים; לדוגמה, ריבוזומים עשויים להיות מורכבים ממספר שונה של rRNA ופוליפפטידים בהתאם לאורגניזם. עם זאת, המבנים והתפקודים הכלליים של מכונות סינתזת החלבון דומים מחיידקים לתאים אנושיים. תרגום דורש קלט של תבנית mRNA, ריבוזומים, tRNAs וגורמים אנזימטיים שונים.
ריבוזומים
עוד לפני תרגום mRNA, תא חייב להשקיע אנרגיה כדי לבנות כל אחד מהריבוזומים שלו. ב אי - קולי, ישנם בין 10,000 ל -70,000 ריבוזומים בכל תא בכל זמן נתון. ריבוזום הוא מקרומולקולה מורכבת המורכבת מ- rRNA מבניים וקטליטיים, ופוליפפטידים רבים ומובחנים. באאוקריוטים, הגרעין מתמחה לחלוטין לסינתזה והרכבה של rRNAs.
ריבוזומים קיימים בציטופלזמה בפרוקריוטים ובציטופלזמה ורטיקולום אנדופלזמי מחוספס באאוקריוטים. למיטוכונדריה ולכלורופלסטים יש גם ריבוזומים משלהם במטריצה ובסטרומה, שנראים דומים יותר לריבוזומים פרוקריוטיים (ובעלי רגישויות דומות לתרופות) מאשר לריבוזומים ממש מחוץ לממברנות החיצוניות שלהם בציטופלזמה. ריבוזומים מתנתקים ליחידות משנה גדולות וקטנות כאשר הם אינם מסנתזים חלבונים ומתחברים מחדש במהלך תחילת התרגום. ב אי - קולי, יחידת המשנה הקטנה מתוארת כ- 30S, ויחידת המשנה הגדולה היא 50S, בסך הכל 70S (נזכיר כי יחידות סוודברג אינן תוספות). לריבוזומים של יונקים יש תת-יחידה קטנה של 40S ויחידת משנה גדולה של 60S, בסך הכל 80S. יחידת המשנה הקטנה אחראית לקשירת תבנית ה-mRNA, בעוד שתת-היחידה הגדולה קושרת ברצף tRNAs. כל מולקולת mRNA מתורגמת בו זמנית על ידי ריבוזומים רבים, כולם מסנתזים חלבון באותו כיוון: קריאת ה-mRNA מ-5' עד 3' וסינתזה של הפוליפפטיד מקצה ה-N לקצה C. מבנה ה- mRNA/פולי-ריבוזום המלא נקרא פוליזום.
tRNAs
ה- tRNA הם מולקולות RNA מבניות שהועתקו מגנים על ידי RNA פולימראז III. בהתאם למין, 40 עד 60 סוגים של tRNAs קיימים בציטופלזמה. משמשים כמתאמים, tRNAs ספציפיים נקשרים לרצפים בתבנית ה- mRNA ומוסיפים את חומצת האמינו המתאימה לשרשרת הפוליפפטיד. לכן, tRNAs הן המולקולות שלמעשה "מתרגמות" את שפת ה- RNA לשפת החלבונים.
מתוך 64 קודוני ה- mRNA האפשריים - או שילובי שלישיות של A, U, G ו- C - שלושה מציינים את סיום סינתזת החלבון ו -61 מציינים תוספת של חומצות אמינו לשרשרת הפוליפפטיד. מתוך 61 אלה, קודון אחד (AUG) מקודד גם את תחילת התרגום. כל אנטיקודון tRNA יכול להתאים בסיס לאחד מקודוני ה-mRNA ולהוסיף חומצת אמינו או לסיים תרגום, על פי הקוד הגנטי. לדוגמה, אם הרצף CUA התרחש על תבנית mRNA במסגרת הקריאה הנכונה, הוא היה קושר tRNA המבטא את הרצף המשלים, GAU, שיקושר לחומצת האמינו לאוצין.
כמולקולות המתאם של התרגום, מפתיע ש-tRNAs יכולים להתאים כל כך הרבה ספציפיות לחבילה כל כך קטנה. קחו בחשבון ש- tRNAs צריכים לקיים אינטראקציה עם שלושה גורמים: 1) עליהם להיות מוכרים על ידי סינתטאז האמינואציל הנכון (ראה להלן); 2) עליהם להיות מוכרים על ידי ריבוזומים; ו- 3) עליהם להיקשר לרצף הנכון ב- mRNA.
סינתזות tRNA אמינואציל
תהליך סינתזת טרום tRNA על ידי RNA פולימראז III יוצר רק את חלק ה- RNA של מולקולת המתאם. יש להוסיף את חומצת האמינו המתאימה מאוחר יותר, לאחר עיבוד ה- tRNA ומיוצא לציטופלזמה. באמצעות תהליך ה"טעינה" של tRNA, כל מולקולת tRNA מקושרת לחומצת האמינו הנכונה שלה על ידי קבוצת אנזימים הנקראים aminoacyl tRNA synthetases. קיים לפחות סוג אחד של סינתטאז tRNA aminoacyl עבור כל אחת מ-20 חומצות האמינו; המספר המדויק של סינתזות tRNA aminoacyl משתנה לפי מינים. אנזימים אלה קושרים תחילה והידרוליזה של ATP כדי לזרז קשר בעל אנרגיה גבוהה בין חומצת אמינו לאדנוסין מונופוספט (AMP); מולקולת פירופוספט גורשת בתגובה זו. חומצת האמינו המופעלת מועברת לאחר מכן ל- tRNA, ו- AMP משתחרר.
מנגנון סינתזת החלבון
כמו בסינתזת mRNA, ניתן לחלק את סינתזת החלבון לשלושה שלבים: התחלה, התארכות וסיום. תהליך התרגום דומה בפרוקריוטים ובאוקריוטים. כאן נחקור כיצד מתרחש תרגום ב אי - קולי, פרוקריוט מייצג, ונציין את ההבדלים בין תרגום פרוקריוטי לאיקריוטי.
ייזום תרגום
סינתזת חלבון מתחילה ביצירת קומפלקס חניכה. ב אי - קולי, קומפלקס זה כולל את הריבוזום הקטן 30S, תבנית ה- mRNA, שלושה גורמי התחלה (IFs; IF-1, IF-2 ו- IF-3), ו- tRNA יוזם מיוחד, הנקרא \(\text{tRNA}_\text{f}^\text{Met}\). The initiator tRNA interacts with the start codon AUG (or rarely, GUG), links to a formylated methionine called fMet, and can also bind IF-2. Formylated methionine is inserted by \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) at the beginning of every polypeptide chain synthesized by E. coli, but it is usually clipped off after translation is complete. When an in-frame AUG is encountered during translation elongation, a non-formylated methionine is inserted by a regular Met-tRNAMet.
In E. coli mRNA, a sequence upstream of the first AUG codon, called the Shine-Dalgarno sequence (AGGAGG), interacts with the rRNA molecules that compose the ribosome. This interaction anchors the 30S ribosomal subunit at the correct location on the mRNA template. Guanosine triphosphate (GTP), which is a purine nucleotide triphosphate, acts as an energy source during translation—both at the start of elongation and during the ribosome’s translocation.
In eukaryotes, a similar initiation complex forms, comprising mRNA, the 40S small ribosomal subunit, IFs, and nucleoside triphosphates (GTP and ATP). The charged initiator tRNA, called Met-tRNAi, does not bind fMet in eukaryotes, but is distinct from other Met-tRNAs in that it can bind IFs.
Instead of depositing at the Shine-Dalgarno sequence, the eukaryotic initiation complex recognizes the 7-methylguanosine cap at the 5' end of the mRNA. A cap-binding protein (CBP) and several other IFs assist the movement of the ribosome to the 5' cap. Once at the cap, the initiation complex tracks along the mRNA in the 5' to 3' direction, searching for the AUG start codon. Many eukaryotic mRNAs are translated from the first AUG, but this is not always the case. According to Kozak’s rules, the nucleotides around the AUG indicate whether it is the correct start codon. Kozak’s rules state that the following consensus sequence must appear around the AUG of vertebrate genes: 5'-gccRccAUGG-3'. The R (for purine) indicates a site that can be either A or G, but cannot be C or U. Essentially, the closer the sequence is to this consensus, the higher the efficiency of translation.
Once the appropriate AUG is identified, the other proteins and CBP dissociate, and the 60S subunit binds to the complex of Met-tRNAi, mRNA, and the 40S subunit. This step completes the initiation of translation in eukaryotes.
Translation, Elongation, and Termination
In prokaryotes and eukaryotes, the basics of elongation are the same, so we will review elongation from the perspective of E. coli. The 50S ribosomal subunit of E. coli consists of three compartments: the A (aminoacyl) site binds incoming charged aminoacyl tRNAs. The P (peptidyl) site binds charged tRNAs carrying amino acids that have formed peptide bonds with the growing polypeptide chain but have not yet dissociated from their corresponding tRNA. The E (exit) site releases dissociated tRNAs so that they can be recharged with free amino acids. There is one exception to this assembly line of tRNAs: in E. coli, \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) is capable of entering the P site directly without first entering the A site. Similarly, the eukaryotic Met-tRNAi, with help from other proteins of the initiation complex, binds directly to the P site. In both cases, this creates an initiation complex with a free A site ready to accept the tRNA corresponding to the first codon after the AUG.
During translation elongation, the mRNA template provides specificity. As the ribosome moves along the mRNA, each mRNA codon comes into register, and specific binding with the corresponding charged tRNA anticodon is ensured. If mRNA were not present in the elongation complex, the ribosome would bind tRNAs nonspecifically.
Elongation proceeds with charged tRNAs entering the A site and then shifting to the P site followed by the E site with each single-codon “step” of the ribosome. Ribosomal steps are induced by conformational changes that advance the ribosome by three bases in the 3' direction. The energy for each step of the ribosome is donated by an elongation factor that hydrolyzes GTP. Peptide bonds form between the amino group of the amino acid attached to the A-site tRNA and the carboxyl group of the amino acid attached to the P-site tRNA. The formation of each peptide bond is catalyzed by peptidyl transferase, an RNA-based enzyme that is integrated into the 50S ribosomal subunit. The energy for each peptide bond formation is derived from GTP hydrolysis, which is catalyzed by a separate elongation factor. The amino acid bound to the P-site tRNA is also linked to the growing polypeptide chain. As the ribosome steps across the mRNA, the former P-site tRNA enters the E site, detaches from the amino acid, and is expelled (Figure \(\PageIndex{2}\)). Amazingly, the E. coli translation apparatus takes only 0.05 seconds to add each amino acid, meaning that a 200-amino acid protein can be translated in just 10 seconds.
Art Connection
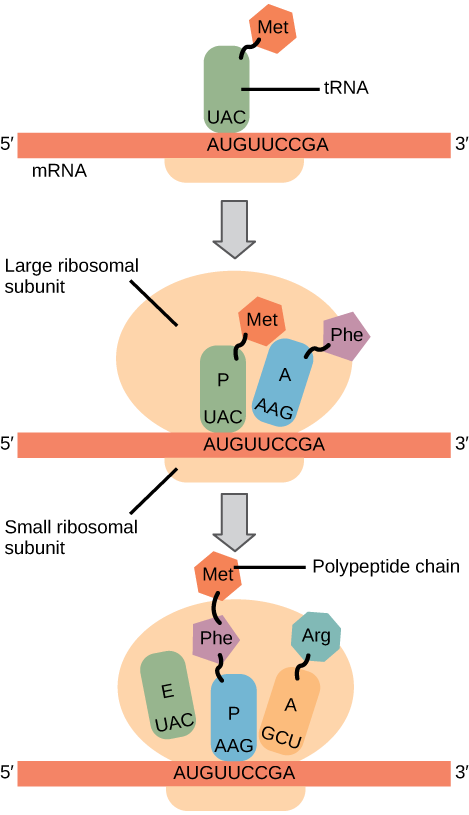
Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Termination of translation occurs when a nonsense codon (UAA, UAG, or UGA) is encountered. Upon aligning with the A site, these nonsense codons are recognized by release factors in prokaryotes and eukaryotes that instruct peptidyl transferase to add a water molecule to the carboxyl end of the P-site amino acid. This reaction forces the P-site amino acid to detach from its tRNA, and the newly made protein is released. The small and large ribosomal subunits dissociate from the mRNA and from each other; they are recruited almost immediately into another translation initiation complex. After many ribosomes have completed translation, the mRNA is degraded so the nucleotides can be reused in another transcription reaction.
Protein Folding, Modification, and Targeting
During and after translation, individual amino acids may be chemically modified, signal sequences may be appended, and the new protein “folds” into a distinct three-dimensional structure as a result of intramolecular interactions. A signal sequence is a short tail of amino acids that directs a protein to a specific cellular compartment. These sequences at the amino end or the carboxyl end of the protein can be thought of as the protein’s “train ticket” to its ultimate destination. Other cellular factors recognize each signal sequence and help transport the protein from the cytoplasm to its correct compartment. For instance, a specific sequence at the amino terminus will direct a protein to the mitochondria or chloroplasts (in plants). Once the protein reaches its cellular destination, the signal sequence is usually clipped off.
Many proteins fold spontaneously, but some proteins require helper molecules, called chaperones, to prevent them from aggregating during the complicated process of folding. Even if a protein is properly specified by its corresponding mRNA, it could take on a completely dysfunctional shape if abnormal temperature or pH conditions prevent it from folding correctly.
Summary
The players in translation include the mRNA template, ribosomes, tRNAs, and various enzymatic factors. The small ribosomal subunit forms on the mRNA template either at the Shine-Dalgarno sequence (prokaryotes) or the 5' cap (eukaryotes). Translation begins at the initiating AUG on the mRNA, specifying methionine. The formation of peptide bonds occurs between sequential amino acids specified by the mRNA template according to the genetic code. Charged tRNAs enter the ribosomal A site, and their amino acid bonds with the amino acid at the P site. The entire mRNA is translated in three-nucleotide “steps” of the ribosome. When a nonsense codon is encountered, a release factor binds and dissociates the components and frees the new protein. Folding of the protein occurs during and after translation.
Art Connections
Figure \(\PageIndex{2}\): Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
- Answer
-
Tetracycline: a; Chloramphenicol: c.
Glossary
- aminoacyl tRNA synthetase
- enzyme that “charges” tRNA molecules by catalyzing a bond between the tRNA and a corresponding amino acid
- initiator tRNA
- in prokaryotes, called \(\text{tRNA}_\text{f}^\text{Met}\); in eukaryotes, called tRNAi; a tRNA that interacts with a start codon, binds directly to the ribosome P site, and links to a special methionine to begin a polypeptide chain
- Kozak’s rules
- determines the correct initiation AUG in a eukaryotic mRNA; the following consensus sequence must appear around the AUG: 5’-GCC(purine)CCAUGG-3’; the bolded bases are most important
- peptidyl transferase
- RNA-based enzyme that is integrated into the 50S ribosomal subunit and catalyzes the formation of peptide bonds
- polysome
- mRNA molecule simultaneously being translated by many ribosomes all going in the same direction
- Shine-Dalgarno sequence
- (AGGAGG); initiates prokaryotic translation by interacting with rRNA molecules comprising the 30S ribosome
- signal sequence
- short tail of amino acids that directs a protein to a specific cellular compartment
- start codon
- AUG (or rarely, GUG) on an mRNA from which translation begins; always specifies methionine



