15.5: Ribossomos e síntese proteica
- Page ID
- 182288
Habilidades para desenvolver
- Descreva as diferentes etapas da síntese de proteínas
- Discuta o papel dos ribossomos na síntese de proteínas
A síntese de proteínas consome mais energia de uma célula do que qualquer outro processo metabólico. Por sua vez, as proteínas representam mais massa do que qualquer outro componente dos organismos vivos (com exceção da água), e as proteínas realizam praticamente todas as funções de uma célula. O processo de tradução, ou síntese de proteínas, envolve a decodificação de uma mensagem de mRNA em um produto polipeptídico. Os aminoácidos são unidos covalentemente pela interligação de ligações peptídicas em comprimentos que variam de aproximadamente 50 resíduos de aminoácidos a mais de 1.000. Cada aminoácido individual tem um grupo amino (NH 2) e um grupo carboxil (COOH). Os polipeptídeos são formados quando o grupo amino de um aminoácido forma uma ligação amida (ou seja, peptídeo) com o grupo carboxila de outro aminoácido (Figura\(\PageIndex{1}\)). Essa reação é catalisada por ribossomos e gera uma molécula de água.
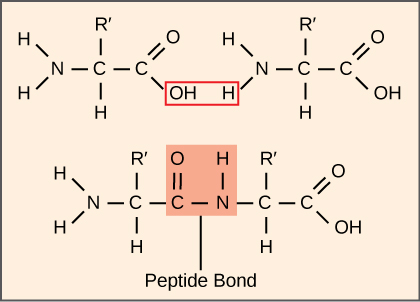
A maquinaria de síntese prote
Além do modelo de mRNA, muitas moléculas e macromoléculas contribuem para o processo de tradução. A composição de cada componente pode variar entre as espécies; por exemplo, os ribossomos podem consistir em diferentes números de rRNAs e polipeptídeos, dependendo do organismo. No entanto, as estruturas e funções gerais do maquinário de síntese de proteínas são comparáveis, desde bactérias até células humanas. A tradução requer a entrada de um modelo de mRNA, ribossomos, tRNAs e vários fatores enzimáticos.
Link para o aprendizado

Clique nas etapas deste PBS interativo para ver a síntese de proteínas em ação.
Ribossomos
Mesmo antes de um mRNA ser traduzido, uma célula deve investir energia para construir cada um de seus ribossomos. Em E. coli, existem entre 10.000 e 70.000 ribossomos presentes em cada célula a qualquer momento. Um ribossomo é uma macromolécula complexa composta por rRNAs estruturais e catalíticos e muitos polipeptídeos distintos. Em eucariotos, o nucléolo é totalmente especializado na síntese e montagem de rRNAs.
Os ribossomos existem no citoplasma dos procariontes e no citoplasma e no retículo endoplasmático rugoso dos eucariotos. As mitocôndrias e os cloroplastos também têm seus próprios ribossomos na matriz e no estroma, que se parecem mais com os ribossomos procarióticos (e têm sensibilidades similares a medicamentos) do que os ribossomos fora de suas membranas externas no citoplasma. Os ribossomos se dissociam em subunidades grandes e pequenas quando não estão sintetizando proteínas e se reassociam durante o início da tradução. Em E. coli, a subunidade pequena é descrita como 30S e a subunidade grande é 50S, para um total de 70S (lembre-se de que as unidades Svedberg não são aditivas). Os ribossomos de mamíferos têm uma pequena subunidade 40S e uma grande subunidade 60S, totalizando 80S. A subunidade pequena é responsável pela ligação ao modelo de mRNA, enquanto a subunidade grande se liga sequencialmente aos tRNAs. Cada molécula de mRNA é traduzida simultaneamente por muitos ribossomos, todos sintetizando proteínas na mesma direção: lendo o mRNA de 5' a 3' e sintetizando o polipeptídeo do terminal N para o terminal C. A estrutura completa do mRNA/poliribossomo é chamada de polissomo.
tRNAs
Os tRNAs são moléculas de RNA estrutural que foram transcritas de genes pela RNA polimerase III. Dependendo da espécie, existem 40 a 60 tipos de tRNAs no citoplasma. Servindo como adaptadores, tRNAs específicos se ligam a sequências no modelo de mRNA e adicionam o aminoácido correspondente à cadeia polipeptídica. Portanto, os tRNAs são as moléculas que realmente “traduzem” a linguagem do RNA para a linguagem das proteínas.
Dos 64 códons de mRNA possíveis - ou combinações trigêmeas de A, U, G e C - três especificam o término da síntese protéica e 61 especificam a adição de aminoácidos à cadeia polipeptídica. Desses 61, um códon (AUG) também codifica o início da tradução. Cada anticódon de tRNA pode emparelhar bases com um dos códons de mRNA e adicionar um aminoácido ou terminar a tradução, de acordo com o código genético. Por exemplo, se a sequência CUA ocorresse em um modelo de mRNA no quadro de leitura adequado, ela se ligaria a um tRNA expressando a sequência complementar, GAU, que estaria ligada ao aminoácido leucina.
Como moléculas adaptadoras da tradução, é surpreendente que os tRNAs possam encaixar tanta especificidade em um pacote tão pequeno. Considere que os tRNAs precisam interagir com três fatores: 1) eles devem ser reconhecidos pela aminoacil sintetase correta (veja abaixo); 2) eles devem ser reconhecidos pelos ribossomos; e 3) eles devem se ligar à sequência correta no mRNA.
Aminoacil tRNA sintetases
O processo de síntese de pré-tRNA pela RNA polimerase III cria apenas a porção de RNA da molécula adaptadora. O aminoácido correspondente deve ser adicionado posteriormente, uma vez que o tRNA é processado e exportado para o citoplasma. Por meio do processo de “carga” do tRNA, cada molécula de tRNA é ligada ao seu aminoácido correto por um grupo de enzimas chamadas aminoacil tRNA sintetases. Existe pelo menos um tipo de aminoacil tRNA sintetase para cada um dos 20 aminoácidos; o número exato de aminoacil tRNA sintetases varia de acordo com a espécie. Essas enzimas primeiro se ligam e hidrolisam o ATP para catalisar uma ligação de alta energia entre um aminoácido e o monofosfato de adenosina (AMP); uma molécula de pirofosfato é expelida nessa reação. O aminoácido ativado é então transferido para o tRNA e o AMP é liberado.
O mecanismo da síntese protéica
Assim como na síntese de mRNA, a síntese de proteínas pode ser dividida em três fases: iniciação, alongamento e término. O processo de tradução é semelhante em procariontes e eucariotas. Aqui, exploraremos como a tradução ocorre em E. coli, um procariota representativo, e especificaremos quaisquer diferenças entre a tradução procariótica e eucariótica.
Início da Tradução
A síntese de proteínas começa com a formação de um complexo de iniciação. Em E. coli, esse complexo envolve o pequeno ribossomo 30S, o modelo de mRNA, três fatores de iniciação (IFs; IF-1, IF-2 e IF-3) e um tRNA iniciador especial, chamado\(\text{tRNA}_\text{f}^\text{Met}\). The initiator tRNA interacts with the start codon AUG (or rarely, GUG), links to a formylated methionine called fMet, and can also bind IF-2. Formylated methionine is inserted by \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) at the beginning of every polypeptide chain synthesized by E. coli, but it is usually clipped off after translation is complete. When an in-frame AUG is encountered during translation elongation, a non-formylated methionine is inserted by a regular Met-tRNAMet.
In E. coli mRNA, a sequence upstream of the first AUG codon, called the Shine-Dalgarno sequence (AGGAGG), interacts with the rRNA molecules that compose the ribosome. This interaction anchors the 30S ribosomal subunit at the correct location on the mRNA template. Guanosine triphosphate (GTP), which is a purine nucleotide triphosphate, acts as an energy source during translation—both at the start of elongation and during the ribosome’s translocation.
In eukaryotes, a similar initiation complex forms, comprising mRNA, the 40S small ribosomal subunit, IFs, and nucleoside triphosphates (GTP and ATP). The charged initiator tRNA, called Met-tRNAi, does not bind fMet in eukaryotes, but is distinct from other Met-tRNAs in that it can bind IFs.
Instead of depositing at the Shine-Dalgarno sequence, the eukaryotic initiation complex recognizes the 7-methylguanosine cap at the 5' end of the mRNA. A cap-binding protein (CBP) and several other IFs assist the movement of the ribosome to the 5' cap. Once at the cap, the initiation complex tracks along the mRNA in the 5' to 3' direction, searching for the AUG start codon. Many eukaryotic mRNAs are translated from the first AUG, but this is not always the case. According to Kozak’s rules, the nucleotides around the AUG indicate whether it is the correct start codon. Kozak’s rules state that the following consensus sequence must appear around the AUG of vertebrate genes: 5'-gccRccAUGG-3'. The R (for purine) indicates a site that can be either A or G, but cannot be C or U. Essentially, the closer the sequence is to this consensus, the higher the efficiency of translation.
Once the appropriate AUG is identified, the other proteins and CBP dissociate, and the 60S subunit binds to the complex of Met-tRNAi, mRNA, and the 40S subunit. This step completes the initiation of translation in eukaryotes.
Translation, Elongation, and Termination
In prokaryotes and eukaryotes, the basics of elongation are the same, so we will review elongation from the perspective of E. coli. The 50S ribosomal subunit of E. coli consists of three compartments: the A (aminoacyl) site binds incoming charged aminoacyl tRNAs. The P (peptidyl) site binds charged tRNAs carrying amino acids that have formed peptide bonds with the growing polypeptide chain but have not yet dissociated from their corresponding tRNA. The E (exit) site releases dissociated tRNAs so that they can be recharged with free amino acids. There is one exception to this assembly line of tRNAs: in E. coli, \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) is capable of entering the P site directly without first entering the A site. Similarly, the eukaryotic Met-tRNAi, with help from other proteins of the initiation complex, binds directly to the P site. In both cases, this creates an initiation complex with a free A site ready to accept the tRNA corresponding to the first codon after the AUG.
During translation elongation, the mRNA template provides specificity. As the ribosome moves along the mRNA, each mRNA codon comes into register, and specific binding with the corresponding charged tRNA anticodon is ensured. If mRNA were not present in the elongation complex, the ribosome would bind tRNAs nonspecifically.
Elongation proceeds with charged tRNAs entering the A site and then shifting to the P site followed by the E site with each single-codon “step” of the ribosome. Ribosomal steps are induced by conformational changes that advance the ribosome by three bases in the 3' direction. The energy for each step of the ribosome is donated by an elongation factor that hydrolyzes GTP. Peptide bonds form between the amino group of the amino acid attached to the A-site tRNA and the carboxyl group of the amino acid attached to the P-site tRNA. The formation of each peptide bond is catalyzed by peptidyl transferase, an RNA-based enzyme that is integrated into the 50S ribosomal subunit. The energy for each peptide bond formation is derived from GTP hydrolysis, which is catalyzed by a separate elongation factor. The amino acid bound to the P-site tRNA is also linked to the growing polypeptide chain. As the ribosome steps across the mRNA, the former P-site tRNA enters the E site, detaches from the amino acid, and is expelled (Figure \(\PageIndex{2}\)). Amazingly, the E. coli translation apparatus takes only 0.05 seconds to add each amino acid, meaning that a 200-amino acid protein can be translated in just 10 seconds.
Art Connection
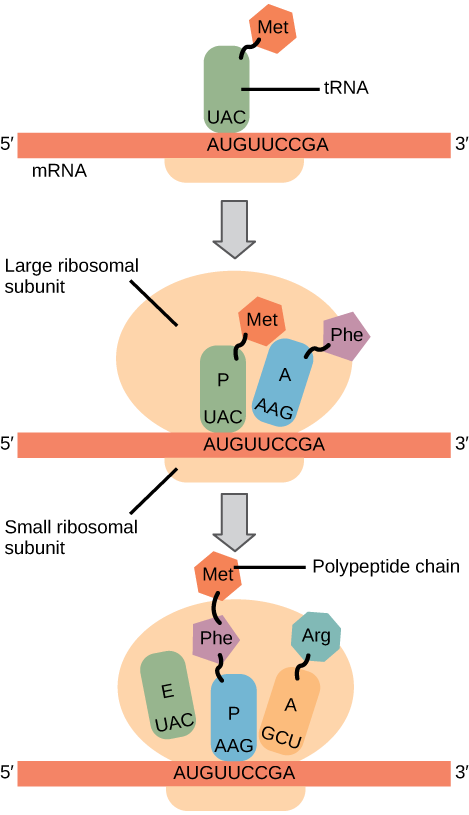
Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Termination of translation occurs when a nonsense codon (UAA, UAG, or UGA) is encountered. Upon aligning with the A site, these nonsense codons are recognized by release factors in prokaryotes and eukaryotes that instruct peptidyl transferase to add a water molecule to the carboxyl end of the P-site amino acid. This reaction forces the P-site amino acid to detach from its tRNA, and the newly made protein is released. The small and large ribosomal subunits dissociate from the mRNA and from each other; they are recruited almost immediately into another translation initiation complex. After many ribosomes have completed translation, the mRNA is degraded so the nucleotides can be reused in another transcription reaction.
Protein Folding, Modification, and Targeting
During and after translation, individual amino acids may be chemically modified, signal sequences may be appended, and the new protein “folds” into a distinct three-dimensional structure as a result of intramolecular interactions. A signal sequence is a short tail of amino acids that directs a protein to a specific cellular compartment. These sequences at the amino end or the carboxyl end of the protein can be thought of as the protein’s “train ticket” to its ultimate destination. Other cellular factors recognize each signal sequence and help transport the protein from the cytoplasm to its correct compartment. For instance, a specific sequence at the amino terminus will direct a protein to the mitochondria or chloroplasts (in plants). Once the protein reaches its cellular destination, the signal sequence is usually clipped off.
Many proteins fold spontaneously, but some proteins require helper molecules, called chaperones, to prevent them from aggregating during the complicated process of folding. Even if a protein is properly specified by its corresponding mRNA, it could take on a completely dysfunctional shape if abnormal temperature or pH conditions prevent it from folding correctly.
Summary
The players in translation include the mRNA template, ribosomes, tRNAs, and various enzymatic factors. The small ribosomal subunit forms on the mRNA template either at the Shine-Dalgarno sequence (prokaryotes) or the 5' cap (eukaryotes). Translation begins at the initiating AUG on the mRNA, specifying methionine. The formation of peptide bonds occurs between sequential amino acids specified by the mRNA template according to the genetic code. Charged tRNAs enter the ribosomal A site, and their amino acid bonds with the amino acid at the P site. The entire mRNA is translated in three-nucleotide “steps” of the ribosome. When a nonsense codon is encountered, a release factor binds and dissociates the components and frees the new protein. Folding of the protein occurs during and after translation.
Art Connections
Figure \(\PageIndex{2}\): Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
- Answer
-
Tetracycline: a; Chloramphenicol: c.
Glossary
- aminoacyl tRNA synthetase
- enzyme that “charges” tRNA molecules by catalyzing a bond between the tRNA and a corresponding amino acid
- initiator tRNA
- in prokaryotes, called \(\text{tRNA}_\text{f}^\text{Met}\); in eukaryotes, called tRNAi; a tRNA that interacts with a start codon, binds directly to the ribosome P site, and links to a special methionine to begin a polypeptide chain
- Kozak’s rules
- determines the correct initiation AUG in a eukaryotic mRNA; the following consensus sequence must appear around the AUG: 5’-GCC(purine)CCAUGG-3’; the bolded bases are most important
- peptidyl transferase
- RNA-based enzyme that is integrated into the 50S ribosomal subunit and catalyzes the formation of peptide bonds
- polysome
- mRNA molecule simultaneously being translated by many ribosomes all going in the same direction
- Shine-Dalgarno sequence
- (AGGAGG); initiates prokaryotic translation by interacting with rRNA molecules comprising the 30S ribosome
- signal sequence
- short tail of amino acids that directs a protein to a specific cellular compartment
- start codon
- AUG (or rarely, GUG) on an mRNA from which translation begins; always specifies methionine


