9.5: Como os genes são regulados
- Page ID
- 179191
Para que uma célula funcione adequadamente, as proteínas necessárias devem ser sintetizadas no momento adequado. Todos os organismos e células controlam ou regulam a transcrição e a tradução de seu DNA em proteína. O processo de ativar um gene para produzir RNA e proteína é chamado de expressão gênica. Seja em um organismo unicelular simples ou em um organismo multicelular complexo, cada célula controla quando e como seus genes são expressos. Para que isso ocorra, deve haver um mecanismo para controlar quando um gene é expresso para produzir RNA e proteína, quanto da proteína é produzida e quando é hora de parar de produzir essa proteína porque ela não é mais necessária.
As células em organismos multicelulares são especializadas; as células em diferentes tecidos têm uma aparência muito diferente e desempenham funções diferentes. Por exemplo, uma célula muscular é muito diferente de uma célula do fígado, que é muito diferente de uma célula da pele. Essas diferenças são consequência da expressão de diferentes conjuntos de genes em cada uma dessas células. Todas as células têm certas funções básicas que devem realizar por si mesmas, como converter a energia das moléculas de açúcar em energia no ATP. Cada célula também tem muitos genes que não são expressos e expressa muitos que não são expressos por outras células, de forma que ela possa realizar suas funções especializadas. Além disso, as células ativarão ou desativarão certos genes em momentos diferentes em resposta a mudanças no ambiente ou em momentos diferentes durante o desenvolvimento do organismo. Organismos unicelulares, tanto eucarióticos quanto procarióticos, também ativam e desativam genes em resposta às demandas de seu ambiente para que possam responder a condições especiais.
O controle da expressão gênica é extremamente complexo. O mau funcionamento desse processo é prejudicial à célula e pode levar ao desenvolvimento de muitas doenças, incluindo câncer.
Expressão gênica procariótica versus eucariótica
Para entender como a expressão gênica é regulada, precisamos primeiro entender como um gene se torna uma proteína funcional em uma célula. O processo ocorre em células procarióticas e eucarióticas, apenas de formas ligeiramente diferentes.
Como os organismos procarióticos não têm um núcleo celular, os processos de transcrição e tradução ocorrem quase simultaneamente. Quando a proteína não é mais necessária, a transcrição é interrompida. Como resultado, o principal método para controlar o tipo e a quantidade de proteína expressa em uma célula procariótica é por meio da regulação da transcrição do DNA em RNA. Todas as etapas subsequentes acontecem automaticamente. Quando mais proteína é necessária, ocorre mais transcrição. Portanto, nas células procarióticas, o controle da expressão gênica é quase inteiramente no nível transcricional.
O primeiro exemplo desse controle foi descoberto usando E. coli nas décadas de 1950 e 1960 por pesquisadores franceses e é chamada de operon lac. O operon lac é um trecho de DNA com três genes adjacentes que codificam proteínas que participam da absorção e metabolismo da lactose, uma fonte de alimento para E. coli. Quando a lactose não está presente no ambiente da bactéria, os genes lac são transcritos em pequenas quantidades. Quando a lactose está presente, os genes são transcritos e a bactéria é capaz de usar a lactose como fonte de alimento. O operon também contém uma sequência promotora à qual a RNA polimerase se liga para iniciar a transcrição; entre o promotor e os três genes está uma região chamada operador. Quando não há lactose presente, uma proteína conhecida como repressora se liga ao operador e impede que a RNA polimerase se ligue ao promotor, exceto em casos raros. Assim, muito pouco dos produtos proteicos dos três genes é produzido. Quando a lactose está presente, um produto final do metabolismo da lactose se liga à proteína repressora e impede que ela se ligue ao operador. Isso permite que a RNA polimerase se ligue ao promotor e transcreva livremente os três genes, permitindo que o organismo metabolize a lactose.
As células eucarióticas, ao contrário, têm organelas intracelulares e são muito mais complexas. Lembre-se de que nas células eucarióticas, o DNA está contido dentro do núcleo da célula e lá é transcrito em mRNA. O mRNA recém-sintetizado é então transportado do núcleo para o citoplasma, onde os ribossomos traduzem o mRNA em proteína. Os processos de transcrição e tradução são fisicamente separados pela membrana nuclear; a transcrição ocorre somente dentro do núcleo e a tradução só ocorre fora do núcleo no citoplasma. A regulação da expressão gênica pode ocorrer em todas as etapas do processo (Figura\(\PageIndex{1}\)). A regulação pode ocorrer quando o DNA é desenrolado e solto dos nucleossomos para se ligar aos fatores de transcrição (nível epigenético), quando o RNA é transcrito (nível transcricional), quando o RNA é processado e exportado para o citoplasma após sua transcrição (nível pós-transcricional), quando o RNA é traduzido em proteína (nível translacional) ou após a proteína ter sido produzida (nível pós-traducional).
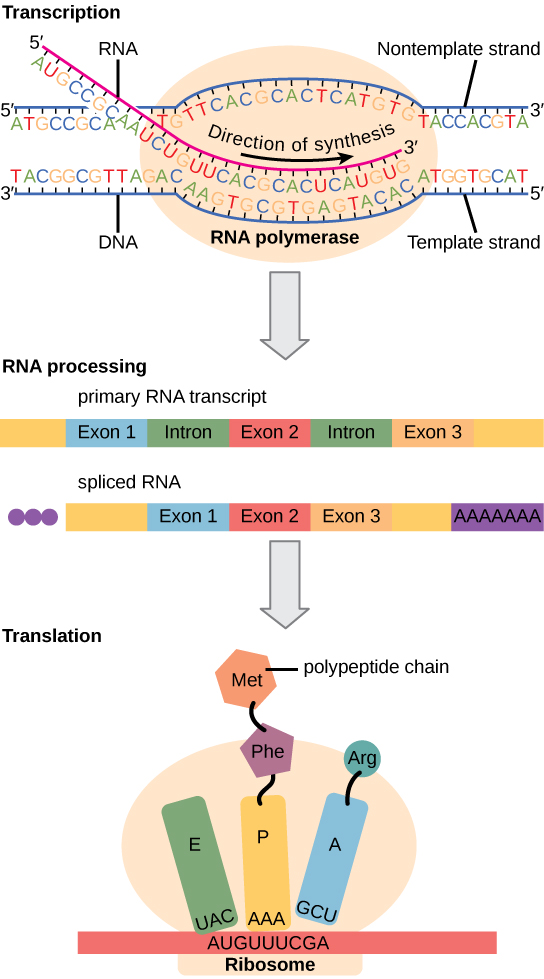
As diferenças na regulação da expressão gênica entre procariontes e eucariotos estão resumidas na Tabela\(\PageIndex{1}\).
| Organismos procarióticos | Organismos eucarióticos |
|---|---|
| Falta de núcleo | Contém núcleo |
| A transcrição de RNA e a tradução de proteínas ocorrem quase simultaneamente |
|
| A expressão gênica é regulada principalmente no nível transcricional | A expressão gênica é regulada em vários níveis (epigenético, transcricional, pós-transcricional, translacional e pós-traducional) |
EVOLUÇÃO EM AÇÃO: Splicing alternativo de RNA
Na década de 1970, foram observados pela primeira vez genes que exibiam splicing alternativo de RNA. O splicing alternativo de RNA é um mecanismo que permite que diferentes produtos proteicos sejam produzidos a partir de um gene quando diferentes combinações de íntrons (e às vezes éxons) são removidas da transcrição (Figura\(\PageIndex{2}\)). Esse splicing alternativo pode ser aleatório, mas mais frequentemente é controlado e atua como um mecanismo de regulação gênica, com a frequência de diferentes alternativas de splicing controladas pela célula como uma forma de controlar a produção de diferentes produtos proteicos em diferentes células, ou em diferentes estágios de desenvolvimento. O splicing alternativo agora é entendido como um mecanismo comum de regulação gênica em eucariotos; de acordo com uma estimativa, 70% dos genes em humanos são expressos como múltiplas proteínas por meio de splicing alternativo.
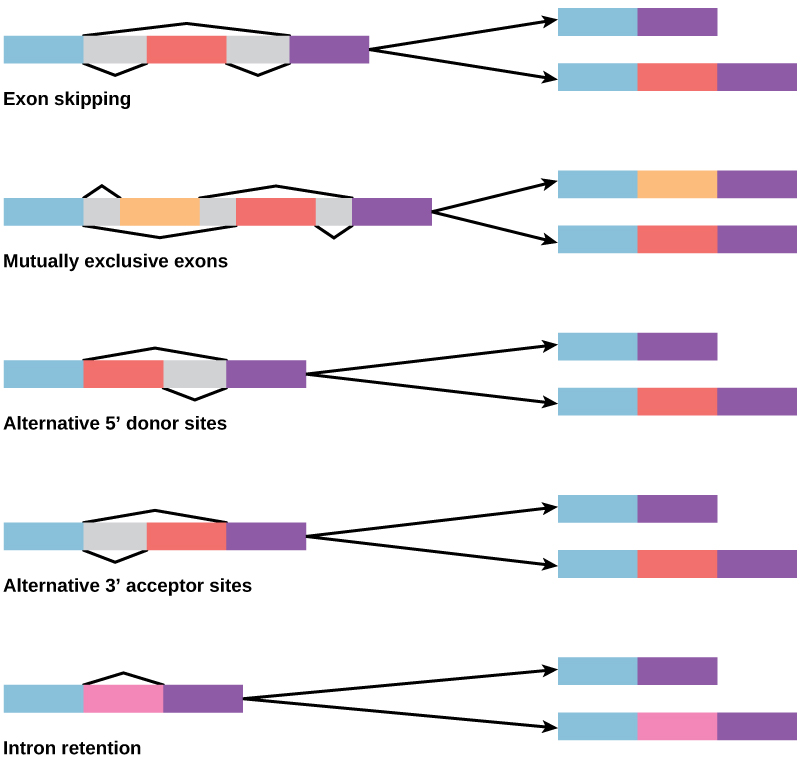
Como o splicing alternativo poderia evoluir? Os íntrons têm uma sequência de reconhecimento inicial e final, e é fácil imaginar a falha do mecanismo de empalme em identificar o fim de um íntron e encontrar o fim do próximo íntron, removendo assim dois íntrons e o éxon intermediário. Na verdade, existem mecanismos para evitar que esse éxon pule, mas é provável que mutações levem à sua falha. Esses “erros” provavelmente produziriam uma proteína não funcional. De fato, a causa de muitas doenças genéticas é o splicing alternativo, em vez de mutações em uma sequência. No entanto, o splicing alternativo criaria uma variante proteica sem a perda da proteína original, abrindo possibilidades de adaptação da nova variante a novas funções. A duplicação de genes desempenhou um papel importante na evolução de novas funções de forma semelhante, fornecendo genes que podem evoluir sem eliminar a proteína funcional original.
Resumo
Embora todas as células somáticas de um organismo contenham o mesmo DNA, nem todas as células desse organismo expressam as mesmas proteínas. Organismos procarióticos expressam todo o DNA que codificam em cada célula, mas não necessariamente todos ao mesmo tempo. As proteínas são expressas somente quando são necessárias. Organismos eucarióticos expressam um subconjunto do DNA que é codificado em qualquer célula. Em cada tipo de célula, o tipo e a quantidade de proteína são regulados pelo controle da expressão gênica. Para expressar uma proteína, o DNA é primeiro transcrito em RNA, que é então traduzido em proteínas. Nas células procarióticas, esses processos ocorrem quase simultaneamente. Nas células eucarióticas, a transcrição ocorre no núcleo e é separada da tradução que ocorre no citoplasma. A expressão gênica em procariontes é regulada apenas no nível transcricional, enquanto nas células eucarióticas, a expressão gênica é regulada nos níveis epigenético, transcricional, pós-transcricional, translacional e pós-traducional.
Glossário
- splicing alternativo de RNA
- um mecanismo de regulação gênica pós-transcricional em eucariotos no qual vários produtos proteicos são produzidos por um único gene por meio de combinações alternativas de splicing do transcrito de RNA
- epigenética
- descrevendo fatores regulatórios não genéticos, como mudanças nas modificações nas proteínas de histonas e no DNA que controlam a acessibilidade a genes nos cromossomos
- expressão gênica
- processos que controlam se um gene é expresso
- pós-transcricional
- controle da expressão gênica após a criação da molécula de RNA, mas antes de ser traduzida em proteína
- pós-traducional
- controle da expressão gênica após a criação de uma proteína


