9.3: Transcrição
- Page ID
- 179190
Tanto em procariontes quanto em eucariotos, a segunda função do DNA (a primeira era a replicação) é fornecer as informações necessárias para construir as proteínas necessárias para que a célula possa realizar todas as suas funções. Para fazer isso, o DNA é “lido” ou transcrito em uma molécula de mRNA. O mRNA então fornece o código para formar uma proteína por meio de um processo chamado tradução. Por meio dos processos de transcrição e tradução, uma proteína é construída com uma sequência específica de aminoácidos que foi originalmente codificada no DNA. Este módulo discute os detalhes da transcrição.
O dogma central: o DNA codifica o RNA; o RNA codifica a proteína
O fluxo de informação genética nas células do DNA ao mRNA e à proteína é descrito pelo dogma central (Figura\(\PageIndex{1}\)), que afirma que os genes especificam as sequências de mRNAs, que por sua vez especificam as sequências de proteínas.
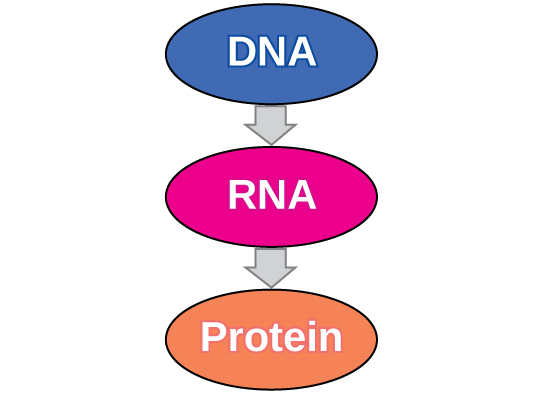
A cópia do DNA para o mRNA é relativamente simples, com um nucleotídeo sendo adicionado à fita de mRNA para cada nucleotídeo complementar lido na fita de DNA. A tradução para proteína é mais complexa porque grupos de três nucleotídeos de mRNA correspondem a um aminoácido da sequência proteica. No entanto, como veremos no próximo módulo, a tradução para proteína ainda é sistemática, de forma que os nucleotídeos 1 a 3 correspondem ao aminoácido 1, os nucleotídeos 4 a 6 correspondem ao aminoácido 2 e assim por diante.
Transcrição: do DNA ao mRNA
Tanto os procariontes quanto os eucariotos realizam fundamentalmente o mesmo processo de transcrição, com a importante diferença do núcleo ligado à membrana nos eucariotos. Com os genes ligados ao núcleo, a transcrição ocorre no núcleo da célula e o transcrito do mRNA deve ser transportado para o citoplasma. Os procariontes, que incluem bactérias e arquéias, não possuem núcleos ligados à membrana e outras organelas, e a transcrição ocorre no citoplasma da célula. Tanto nos procariontes quanto nos eucariotos, a transcrição ocorre em três estágios principais: iniciação, alongamento e término.
Iniciação
A transcrição requer que a dupla hélice do DNA se desenrole parcialmente na região da síntese de mRNA. A região de desenrolamento é chamada de bolha de transcrição. A sequência de DNA na qual as proteínas e enzimas envolvidas na transcrição se ligam para iniciar o processo é chamada de promotora. Na maioria dos casos, os promotores existem a montante dos genes que eles regulam. A sequência específica de um promotor é muito importante porque determina se o gene correspondente é transcrito o tempo todo, parte do tempo ou quase nunca (Figura\(\PageIndex{2}\)).
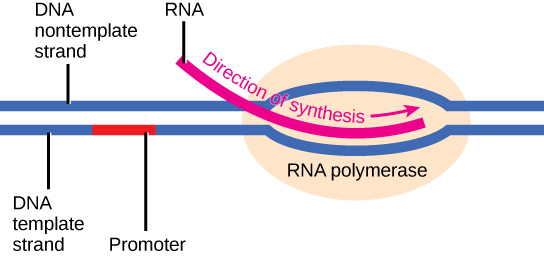
Alongamento
A transcrição sempre procede de uma das duas cadeias de DNA, que é chamada de fita modelo. O produto de mRNA é complementar à fita modelo e é quase idêntico à outra fita de DNA, chamada de fita não modelo, com a exceção de que o RNA contém um uracil (U) no lugar da timina (T) encontrada no DNA. Durante o alongamento, uma enzima chamada RNA polimerase prossegue ao longo do modelo de DNA adicionando nucleotídeos por emparelhamento de bases com o modelo de DNA de maneira semelhante à replicação do DNA, com a diferença de que uma fita de RNA está sendo sintetizada e não permanece ligada ao modelo de DNA. À medida que o alongamento prossegue, o DNA é continuamente desenrolado à frente da enzima central e rebobinado atrás dela (Figura\(\PageIndex{3}\)).
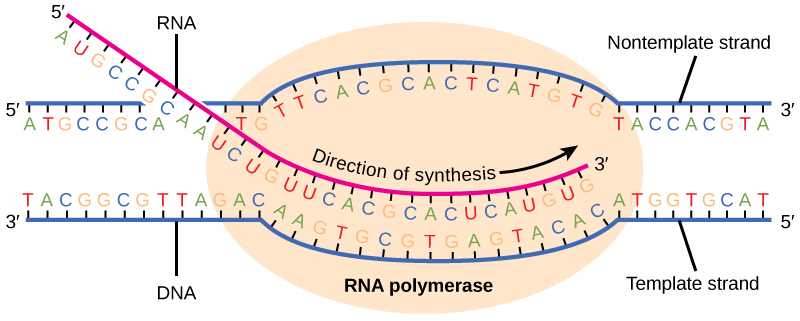
Rescisão
Depois que um gene é transcrito, a polimerase procariótica precisa ser instruída a se dissociar do modelo de DNA e liberar o mRNA recém-criado. Dependendo do gene que está sendo transcrito, existem dois tipos de sinais de terminação, mas ambos envolvem sequências repetidas de nucleotídeos no modelo de DNA que resultam na paralisação da RNA polimerase, saindo do molde de DNA e liberando a transcrição do mRNA.
No encerramento, o processo de transcrição é concluído. Em uma célula procariótica, no momento em que ocorre o término, a transcrição já teria sido usada para sintetizar parcialmente várias cópias da proteína codificada porque esses processos podem ocorrer simultaneamente usando vários ribossomos (poliribossomos) (Figura\(\PageIndex{4}\)). Em contraste, a presença de um núcleo nas células eucarióticas impede a transcrição e tradução simultâneas.
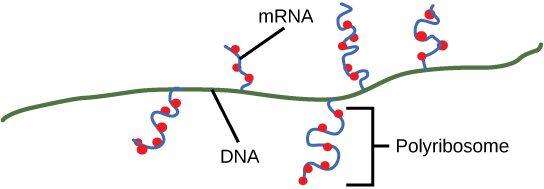
Processamento de RNA eucariótico
Os mRNAs eucarióticos recém-transcritos devem passar por várias etapas de processamento antes de serem transferidos do núcleo para o citoplasma e traduzidos em uma proteína. As etapas adicionais envolvidas na maturação do mRNA eucariótico criam uma molécula que é muito mais estável do que um mRNA procariótico. Por exemplo, os mRNAs eucarióticos duram várias horas, enquanto o mRNA procariótico típico não dura mais do que cinco segundos.
O transcrito do mRNA é primeiro revestido com proteínas estabilizadoras de RNA para evitar que se degrade enquanto é processado e exportado para fora do núcleo. Isso ocorre enquanto o pré-mRNA ainda está sendo sintetizado, adicionando uma “tampa” de nucleotídeo especial à extremidade 5' da transcrição crescente. Além de impedir a degradação, os fatores envolvidos na síntese de proteínas reconhecem a tampa para ajudar a iniciar a tradução pelos ribossomos.
Quando o alongamento é concluído, uma enzima então adiciona uma sequência de aproximadamente 200 resíduos de adenina na extremidade 3', chamada cauda de poli-A. Essa modificação protege ainda mais o pré-mRNA da degradação e sinaliza aos fatores celulares que a transcrição precisa ser exportada para o citoplasma.
Os genes eucarióticos são compostos por sequências codificadoras de proteínas chamadas éxons (ex- on significa que eles são expressos) e sequências inerventes chamadas introns (int- ron denota seu papel int ervening). Os introns são removidos do pré-mRNA durante o processamento. As sequências de íntrons no mRNA não codificam proteínas funcionais. É essencial que todos os introns de um pré-mRNA sejam removidos de forma completa e precisa antes da síntese protéica, para que os éxons se unam para codificar os aminoácidos corretos. Se o processo errar até mesmo por um único nucleotídeo, a sequência dos éxons reunidos seria alterada e a proteína resultante não funcionaria. O processo de remoção de íntrons e reconexão de éxons é chamado de splicing (Figura\(\PageIndex{5}\)). Os íntrons são removidos e degradados enquanto o pré-mRNA ainda está no núcleo.
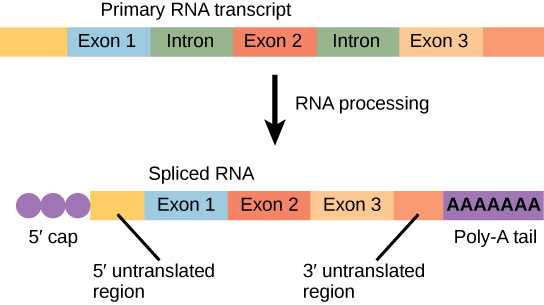
Resumo
Em procariontes, a síntese de mRNA é iniciada em uma sequência promotora no modelo de DNA. O alongamento sintetiza um novo mRNA. A terminação libera o mRNA e ocorre por mecanismos que impedem a RNA polimerase e fazem com que ela caia do molde de DNA. Os mRNAs eucarióticos recém-transcritos são modificados com uma tampa e uma cauda de poli-A. Essas estruturas protegem o mRNA maduro da degradação e ajudam a exportá-lo do núcleo. Os mRNAs eucarióticos também passam por splicing, no qual os íntrons são removidos e os éxons são reconectados com precisão de nucleotídeo único. Somente mRNAs acabados são exportados do núcleo para o citoplasma.
Glossário
- éxon
- uma sequência presente no mRNA codificador de proteínas após a conclusão do splicing pré-mRNA
- íntron
- sequências intervenientes não codificadoras de proteínas que são emendadas a partir do mRNA durante o processamento
- mRNA
- RNA mensageiro; uma forma de RNA que carrega o código de sequência de nucleotídeos para uma sequência de proteína que é traduzida em uma sequência polipeptídica
- fio não modelo
- a fita de DNA que não é usada para transcrever mRNA; essa fita é idêntica ao mRNA, exceto que os nucleotídeos T no DNA são substituídos por nucleotídeos U no mRNA
- promoter
- uma sequência no DNA à qual a RNA polimerase e fatores associados se ligam e iniciam a transcrição
- RNA polimerase
- uma enzima que sintetiza uma fita de RNA a partir de uma fita modelo de DNA
- emenda
- o processo de remoção de introns e reconexão de éxons em um pré-mRNA
- vertente modelo
- a fita de DNA que especifica a molécula complementar de mRNA
- bolha de transcrição
- a região do DNA desenrolado localmente que permite a transcrição do mRNA


