11.2 : Réplication d'ADN
- Page ID
- 187682
Objectifs d'apprentissage
- Expliquer la signification de la réplication semi-conservatrice de l'ADN
- Expliquer pourquoi la réplication de l'ADN est bidirectionnelle et comprend à la fois un brin principal et un brin retardé
- Expliquez pourquoi des fragments d'Okazaki se forment
- Décrire le processus de réplication de l'ADN et les fonctions des enzymes impliquées
- Identifier les différences entre la réplication de l'ADN chez les bactéries et chez les eucaryotes
- Expliquer le processus de réplication circulaire
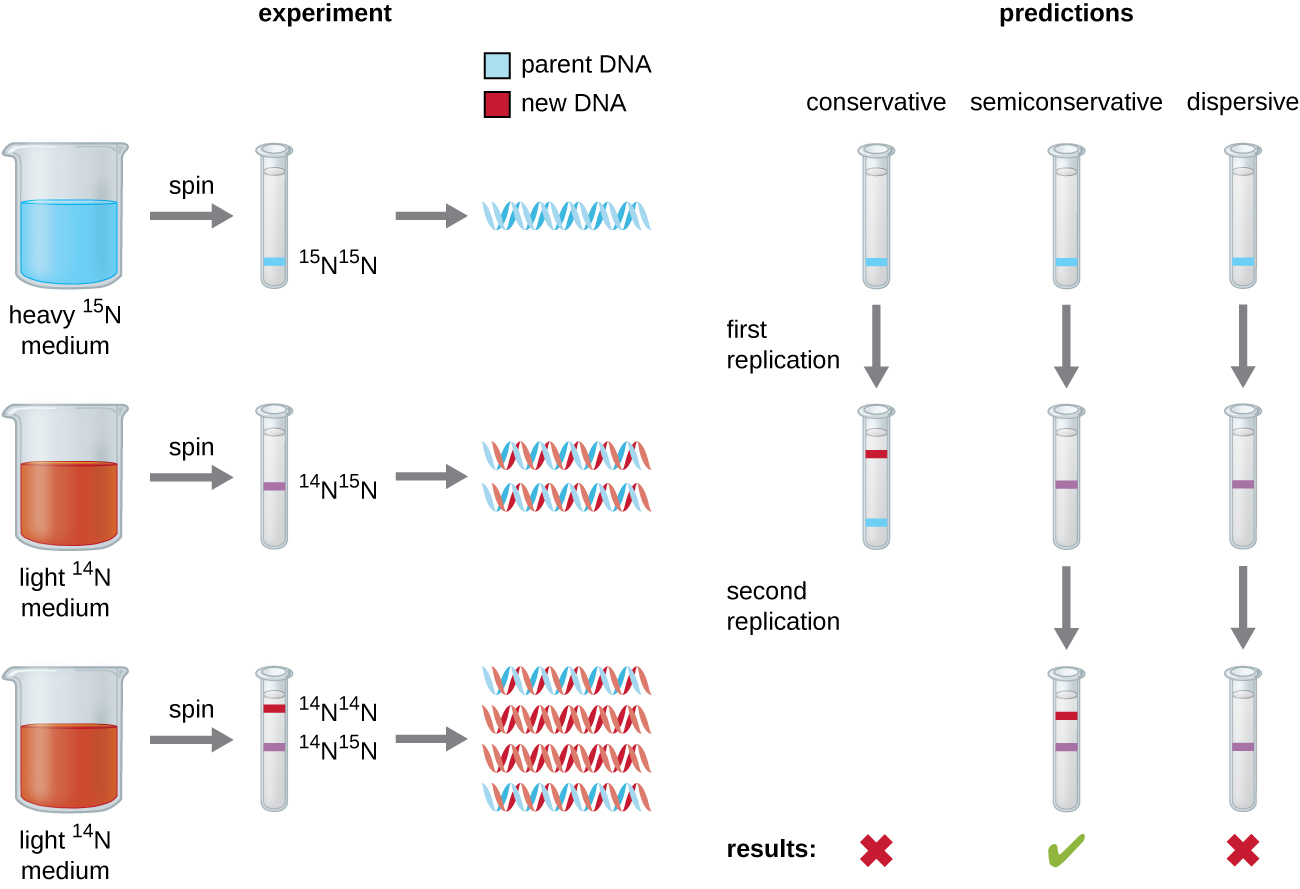
L'élucidation de la structure de la double hélice par James Watson et Francis Crick en 1953 a fourni une idée de la manière dont l'ADN est copié au cours du processus de réplication. La séparation des brins de la double hélice fournirait deux modèles pour la synthèse de nouveaux brins complémentaires, mais la manière exacte dont les nouvelles molécules d'ADN étaient construites n'était toujours pas claire. Dans un modèle, la réplication semi-conservatrice, les deux brins de la double hélice se séparent lors de la réplication de l'ADN, et chaque brin sert de matrice à partir de laquelle le nouveau brin complémentaire est copié ; après réplication, chaque ADN double brin comprend un brin parent ou « ancien » brin et un « nouveau » brin. Deux modèles concurrents ont également été suggérés : conservateur et dispersif, qui sont illustrés dans la figure\(\PageIndex{1}\).

Matthew Meselson (1930—) et Franklin Stahl (1929—) ont conçu une expérience en 1958 pour tester lequel de ces modèles représente correctement la réplication de l'ADN (Figure\(\PageIndex{2}\)). Ils ont cultivé E. coli pendant plusieurs générations dans un milieu contenant un isotope « lourd » de l'azote (15 N) qui a été incorporé dans des bases azotées et, finalement, dans l'ADN. Cela a marqué l'ADN parental. La culture d'E. coli a ensuite été transférée dans un milieu contenant 14 N et laissée croître pendant une génération. Les cellules ont été récoltées et l'ADN a été isolé. L'ADN a été séparé par ultracentrifugation, au cours de laquelle l'ADN a formé des bandes en fonction de sa densité. L'ADN cultivé dans 15 N devrait former une bande à une densité plus élevée que celle cultivée dans 14 N. Meselson et Stahl ont noté qu'après une génération de croissance dans le 14 N, la bande unique observée était intermédiaire entre l'ADN de cellules cultivées exclusivement dans 15 N ou 14 N. Cela suggère un mode de réplication semi-conservateur ou dispersif. Certaines cellules ont été laissées croître pendant une génération supplémentaire dans 14 N et ont été filées à nouveau. L'ADN prélevé à partir de cellules cultivées pendant deux générations dans du 14 N a formé deux bandes : une bande d'ADN se trouvait à une position intermédiaire entre 15 N et 14 N, et l'autre correspondait à la bande d'ADN 14 N. Ces résultats ne peuvent être expliqués que si l'ADN se réplique de manière semi-conservatrice. Les deux autres modèles ont donc été exclus. À la suite de cette expérience, nous savons maintenant que lors de la réplication de l'ADN, chacun des deux brins qui composent la double hélice sert de matrice à partir de laquelle les nouveaux brins sont copiés. Le nouveau volet sera complémentaire du volet parental ou « ancien » volet. Les molécules d'ADN qui en résultent ont la même séquence et sont divisées à parts égales entre les deux cellules filles.
Exercice\(\PageIndex{1}\)
Quelle aurait été la conclusion de l'expérience de Meselson et Stahl si, après la première génération, ils avaient découvert deux bandes d'ADN ?
Réplication de l'ADN chez
La réplication de l'ADN a été bien étudiée chez les bactéries, principalement en raison de la petite taille du génome et des mutants disponibles. E. coli possède 4,6 millions de paires de bases (Mbp) dans un seul chromosome circulaire et l'ensemble est répliqué en 42 minutes environ, en partant d'une seule origine de réplication et en faisant le tour du cercle de manière bidirectionnelle (c'est-à-dire dans les deux sens). Cela signifie qu'environ 1000 nucléotides sont ajoutés par seconde. Le processus est assez rapide et se produit avec peu d'erreurs.
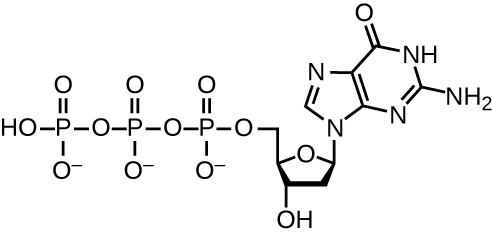
La réplication de l'ADN utilise un grand nombre de protéines et d'enzymes (Tableau\(\PageIndex{1}\)). L'un des principaux acteurs est l'enzyme ADN polymérase, également connue sous le nom d'ADN pol. Chez les bactéries, trois principaux types d'ADN polymérases sont connus : l'ADN pol I, l'ADN pol II et l'ADN pol III. On sait maintenant que l'ADN pol III est l'enzyme nécessaire à la synthèse de l'ADN ; l'ADN pol I et l'ADN pol II sont principalement nécessaires à la réparation. L'ADN pol III ajoute des désoxyribonucléotides, chacun complémentaire d'un nucléotide sur le brin matrice, un par un au groupe 3'-OH de la chaîne d'ADN en croissance. L'ajout de ces nucléotides nécessite de l'énergie. Cette énergie est présente dans les liaisons de trois groupes phosphates attachés à chaque nucléotide (un nucléotide triphosphate), de la même manière que l'énergie est stockée dans les liaisons phosphate de l'adénosine triphosphate (ATP) (Figure\(\PageIndex{3}\)). Lorsque la liaison entre les phosphates est rompue et que le diphosphate est libéré, l'énergie libérée permet la formation d'une liaison phosphodiester covalente par synthèse par déshydratation entre le nucléotide entrant et le groupe 3'-OH libre sur le brin d'ADN en croissance.
Initiation
L'initiation de la réplication se produit au niveau d'une séquence nucléotidique spécifique appelée origine de la réplication, où diverses protéines se lient pour amorcer le processus de réplication. E. coli possède une seule origine de réplication (comme la plupart des procaryotes), appelée ORIC, sur son seul chromosome. L'origine de la réplication est longue d'environ 245 paires de bases et est riche en séquences d'adénine-thymine (AT).
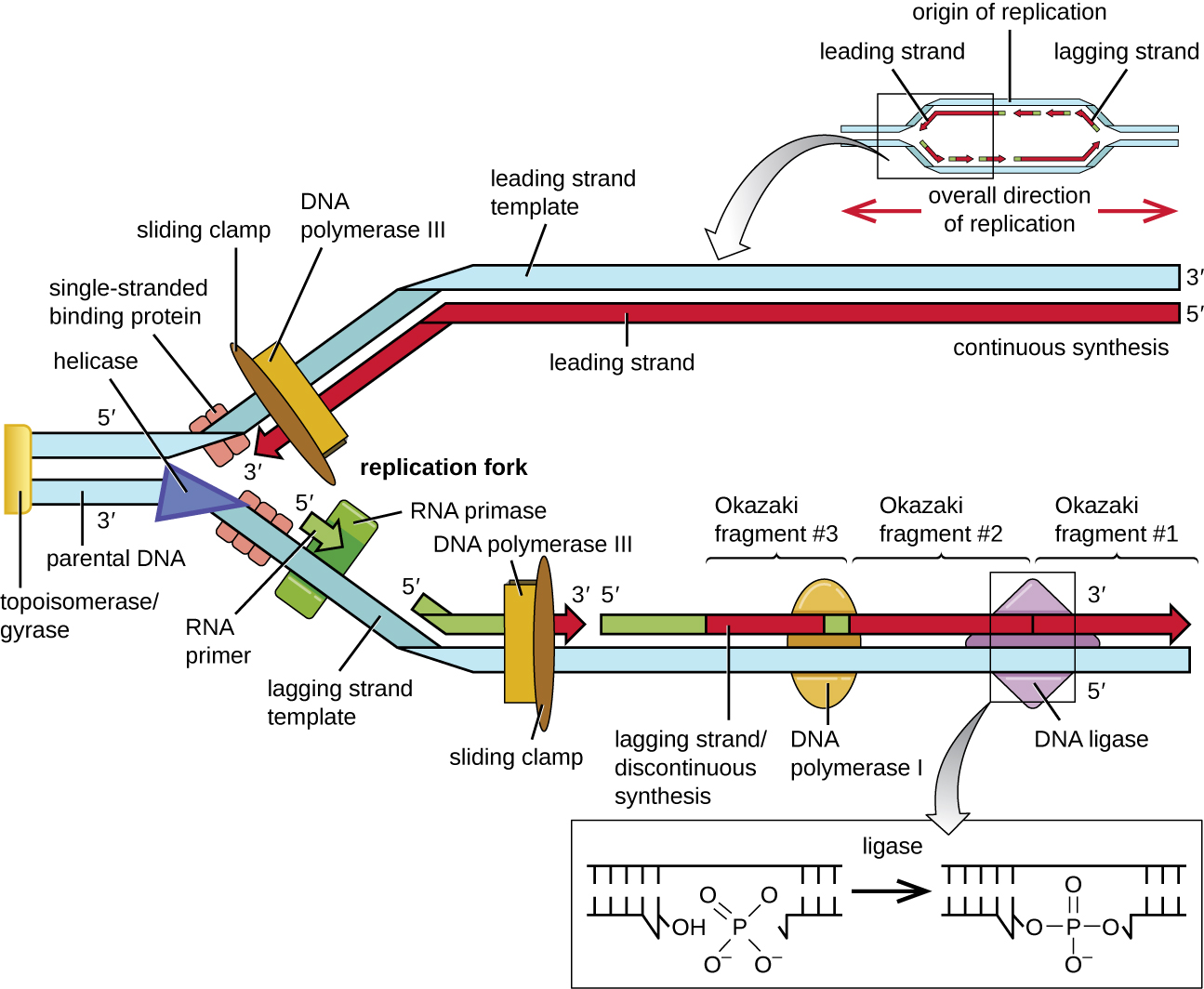
Certaines des protéines qui se lient à l'origine de la réplication jouent un rôle important dans la mise à disposition des régions monocaténaires de l'ADN à des fins de réplication. L'ADN chromosomique est généralement enroulé autour d'histones (chez les eucaryotes et les archées) ou de protéines analogues aux histones (chez les bactéries), et est superenroulé ou largement enroulé et tordu sur lui-même. Cet emballage rend les informations contenues dans la molécule d'ADN inaccessibles. Cependant, des enzymes appelées topoisomérases modifient la forme et le superenroulement du chromosome. Pour que la réplication de l'ADN bactérien commence, le chromosome superenroulé est relâché par la topoisomérase II, également appelée ADN gyrase. Une enzyme appelée hélicase sépare ensuite les brins d'ADN en rompant les liaisons hydrogène entre les paires de bases azotées. Rappelons que les séquences AT ont moins de liaisons hydrogène et, par conséquent, ont des interactions plus faibles que les séquences guanine-cytosine (GC). Ces enzymes nécessitent une hydrolyse de l'ATP. Lorsque l'ADN s'ouvre, des structures en forme de Y appelées fourches de réplication se forment. Deux fourches de réplication sont formées à l'origine de la réplication, ce qui permet une réplication bidirectionnelle et la formation d'une structure qui ressemble à une bulle lorsqu'on l'observe au microscope électronique à transmission ; par conséquent, cette structure est appelée bulle de réplication. L'ADN situé à proximité de chaque fourche de réplication est recouvert de protéines de liaison monocaténaires pour empêcher l'ADN monocaténaire de se rembobiner en une double hélice.
Une fois que l'ADN monocaténaire est accessible à l'origine de la réplication, la réplication de l'ADN peut commencer. Cependant, l'ADN pol III est capable d'ajouter des nucléotides uniquement dans la direction 5' à 3' (un nouveau brin d'ADN ne peut être étendu que dans cette direction). En effet, l'ADN polymérase a besoin d'un groupe 3'-OH libre auquel elle peut ajouter des nucléotides en formant une liaison phosphodiester covalente entre l'extrémité 3'-OH et le phosphate 5' du nucléotide suivant. Cela signifie également qu'il ne peut pas ajouter de nucléotides si aucun groupe 3'-OH libre n'est disponible, ce qui est le cas pour un seul brin d'ADN. Le problème est résolu à l'aide d'une séquence d'ARN qui fournit l'extrémité 3'-OH libre. Comme cette séquence permet de démarrer la synthèse de l'ADN, elle est appelée à juste titre amorce. L'amorce a une longueur de cinq à dix nucléotides et est complémentaire de l'ADN parental ou de la matrice. Elle est synthétisée par l'ARN primase, qui est une ARN polymérase. Contrairement aux ADN polymérases, les ARN polymérases n'ont pas besoin d'un groupe 3'-OH libre pour synthétiser une molécule d'ARN. Maintenant que l'amorce fournit le groupe 3'-OH libre, l'ADN polymérase III peut désormais étendre cette amorce d'ARN, en ajoutant un par un des nucléotides d'ADN complémentaires au brin matrice (Figure\(\PageIndex{1}\)).
Allongement
Lors de l'élongation de la réplication de l'ADN, l'ajout de nucléotides se produit à sa vitesse maximale d'environ 1 000 nucléotides par seconde. L'ADN polymérase III ne peut s'étendre que dans la direction 5' à 3', ce qui pose un problème au niveau de la fourche de réplication. La double hélice de l'ADN est antiparallèle, c'est-à-dire qu'un brin est orienté dans la direction 5' à 3' et l'autre est orienté dans la direction 3' à 5' (voir Structure et fonction de l'ADN). Pendant la réplication, un brin, complémentaire du brin d'ADN parental de 3' à 5', est synthétisé en continu vers la fourche de réplication car la polymérase peut ajouter des nucléotides dans cette direction. Ce brin synthétisé en continu est connu sous le nom de brin principal. L'autre brin, complémentaire de l'ADN parental de 5' à 3', s'éloigne de la fourche de réplication, de sorte que la polymérase doit revenir vers la fourche de réplication pour commencer à ajouter des bases à une nouvelle amorce, toujours dans la direction opposée à la fourche de réplication. Il le fait jusqu'à ce qu'il heurte le brin précédemment synthétisé, puis qu'il recule (Figure\(\PageIndex{4}\)). Ces étapes produisent de petits fragments de séquences d'ADN appelés fragments d'Okazaki, chacun étant séparé par une amorce d'ARN. Les fragments d'Okazaki portent le nom de l'équipe de recherche japonaise et du couple marié Reiji et Tsuneko Okazaki, qui les ont découverts pour la première fois en 1966. Le brin contenant les fragments d'Okazaki est connu sous le nom de brin en retard, et sa synthèse est dite discontinue.
Le brin principal peut être étendu à partir d'une seule amorce, tandis que le brin en retard a besoin d'un nouvel apprêt pour chacun des courts fragments d'Okazaki. La direction générale du brin en retard sera de 3' à 5', et celle du brin avant de 5' à 3'. Une protéine appelée pince coulissante maintient l'ADN polymérase en place alors qu'elle continue à ajouter des nucléotides. La pince coulissante est une protéine en forme d'anneau qui se lie à l'ADN et maintient la polymérase en place. Au-delà de son rôle dans l'initiation, la topoisomérase empêche également le débordement de la double hélice de l'ADN avant la fourche de réplication lorsque l'ADN s'ouvre ; elle le fait en provoquant des entailles temporaires dans l'hélice de l'ADN puis en la refermant. Au fur et à mesure de la synthèse, les amorces d'ARN sont remplacées par l'ADN. Les amorces sont éliminées par l'activité exonucléasique de l'ADN polymérase I, et les lacunes sont comblées. Les entailles qui subsistent entre l'ADN nouvellement synthétisé (qui a remplacé l'amorce d'ARN) et l'ADN précédemment synthétisé sont scellées par l'enzyme ADN ligase qui catalyse la formation d'une liaison phosphodiester covalente entre l'extrémité 3'-OH d'un fragment d'ADN et l'extrémité phosphate 5' de l'autre fragment, stabiliser le squelette sucre-phosphate de la molécule d'ADN.
Résiliation
Une fois que le chromosome complet a été répliqué, la réplication de l'ADN doit se terminer. Bien que l'on sache beaucoup de choses sur le lancement de la réplication, on en sait moins sur le processus de terminaison. Après la réplication, les génomes circulaires complets des procaryotes qui en résultent sont concaténés, ce qui signifie que les chromosomes circulaires de l'ADN sont imbriqués et doivent être séparés les uns des autres. Cela se fait grâce à l'activité de la topoisomérase IV bactérienne, qui introduit des cassures bicaténaires dans les molécules d'ADN, leur permettant ainsi de se séparer les unes des autres ; l'enzyme referme ensuite les chromosomes circulaires. La résolution des concatémères est un problème propre à la réplication de l'ADN procaryote en raison de leurs chromosomes circulaires. Comme l'ADN gyrase et la topoisomérase IV des bactéries sont toutes deux distinctes de leurs homologues eucaryotes, ces enzymes servent de cibles à une classe de médicaments antimicrobiens appelés quinolones.
| Enzyme ou facteur | Fonction |
|---|---|
| Pôle ADN II | L'activité de l'exonucléase élimine l'amorce d'ARN et la remplace par de l'ADN nouvellement synthé |
| ADN pol III | Enzyme principale qui ajoute des nucléotides dans la direction 5' à 3' |
| Hélicase | Ouvre l'hélice de l'ADN en rompant les liaisons hydrogène entre les bases azotées |
| Ligase | Comble les espaces entre les fragments d'Okazaki sur le brin en retard pour créer un brin d'ADN continu |
| Primase | Synthétise les amorces d'ARN nécessaires pour démarrer la réplication |
| Protéines de liaison à brin | Liez-vous à l'ADN monocaténaire pour empêcher la liaison hydrogène entre les brins d'ADN, reformant ainsi l'ADN double brin |
| Pince coulissante | Aide à maintenir l'ADN pol III en place lorsque des nucléotides sont ajoutés |
| Topoisomérase II (ADN gyrase) | Relaxe le chromosome superenroulé pour rendre l'ADN plus accessible pour le début de la réplication ; aide à soulager le stress sur l'ADN lors du déroulement, en provoquant des cassures puis en refermant l'ADN |
| Topoisomérase IV | Introduit une rupture monocaténaire dans les chromosomes concaténés pour les libérer les uns des autres, puis rescelle l'ADN |
Exercice\(\PageIndex{2}\)
- Quelle enzyme rompt les liaisons hydrogène qui maintiennent les deux brins d'ADN ensemble afin que la réplication puisse se produire ?
- Est-ce le brin en retard ou le brin principal qui est synthétisé dans le sens de l'ouverture de la fourche de réplication ?
- Quelle enzyme est responsable de l'élimination des amorces d'ARN dans l'ADN bactérien nouvellement répliqué ?
Réplication d'ADN chez les eucaryotes
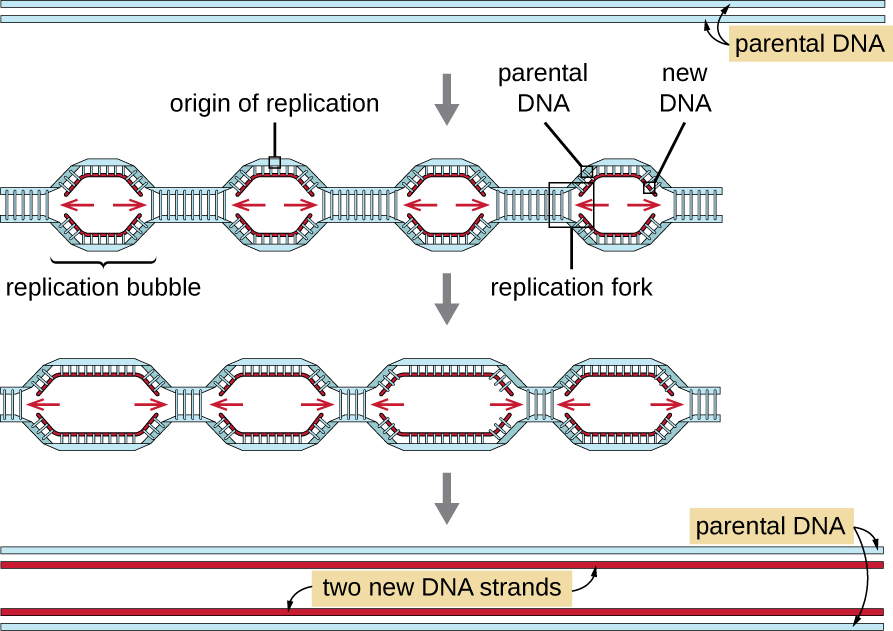
Les génomes eucaryotes sont beaucoup plus complexes et plus volumineux que les génomes procaryotes et sont généralement composés de plusieurs chromosomes linéaires (Tableau\(\PageIndex{2}\)). Le génome humain, par exemple, possède 3 milliards de paires de bases par ensemble haploïde de chromosomes, et 6 milliards de paires de bases sont insérées lors de la réplication. Il existe de multiples origines de réplication sur chaque chromosome eucaryote (Figure\(\PageIndex{5}\)) ; le génome humain possède 30 000 à 50 000 origines de réplication. Le taux de réplication est d'environ 100 nucléotides par seconde, soit 10 fois plus lent que la réplication procaryote.
Les étapes essentielles de la réplication chez les eucaryotes sont les mêmes que chez les procaryotes. Avant que la réplication puisse commencer, l'ADN doit être disponible sous forme de modèle. L'ADN eucaryote est hautement superenroulé et emballé, ce qui est facilité par de nombreuses protéines, y compris les histones (voir Structure et fonction des génomes cellulaires). À l'origine de la réplication, un complexe de préréplication composé de plusieurs protéines, dont l'hélicase, forme et recrute d'autres enzymes impliquées dans l'initiation de la réplication, notamment la topoisomérase pour détendre le superenroulement, la protéine de liaison monocaténaire, l'ARN primase et l'ADN polymérase. Après le début de la réplication, selon un processus similaire à celui observé chez les procaryotes, l'élongation est facilitée par les ADN polymérases eucaryotes. Le brin principal est synthétisé en continu par l'enzyme polymérase eucaryote pol δ, tandis que le brin en retard est synthétisé par pol ε. Une protéine à pince coulissante maintient l'ADN polymérase en place afin qu'elle ne tombe pas de l'ADN. L'enzyme ribonucléase H (RNase H), au lieu d'une ADN polymérase comme chez les bactéries, élimine l'amorce d'ARN, qui est ensuite remplacée par des nucléotides d'ADN. Les lacunes qui subsistent sont scellées par l'ADN ligase.
Comme les chromosomes eucaryotes sont linéaires, on peut s'attendre à ce que leur réplication soit plus simple. Comme chez les procaryotes, l'ADN polymérase eucaryote ne peut ajouter des nucléotides que dans la direction 5' à 3'. Dans le brin principal, la synthèse se poursuit jusqu'à ce qu'elle atteigne l'extrémité du chromosome ou une autre fourche de réplication progressant dans la direction opposée. Sur le brin en retard, l'ADN est synthétisé en courtes périodes, chacune étant initiée par une amorce distincte. Lorsque la fourche de réplication atteint l'extrémité du chromosome linéaire, il n'y a aucun endroit où créer une amorce pour le fragment d'ADN à copier à l'extrémité du chromosome. Ces extrémités ne sont donc pas appariées et, au fil du temps, elles peuvent se raccourcir progressivement à mesure que les cellules continuent de se diviser.
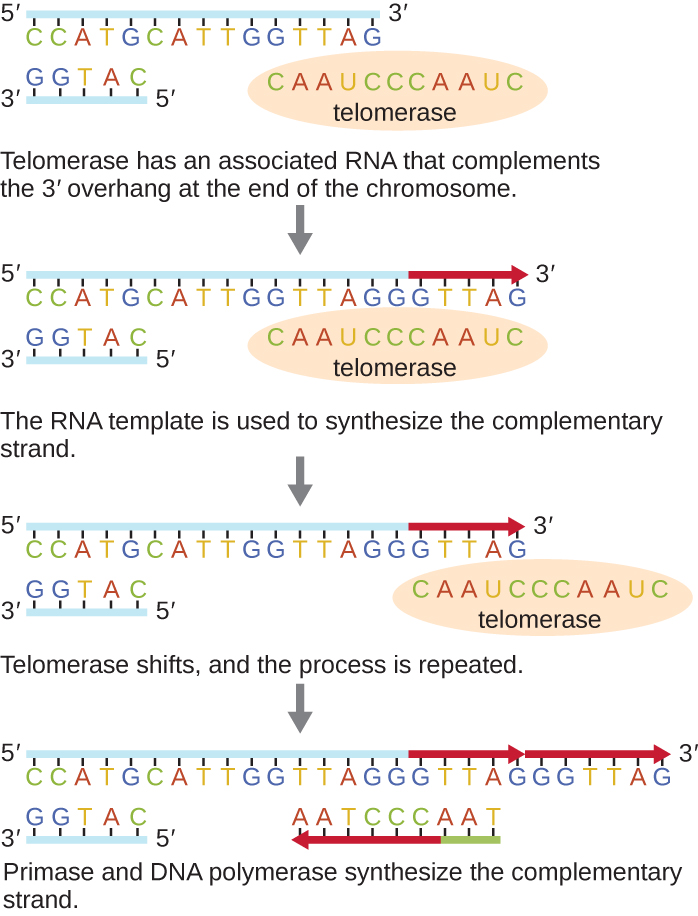
Les extrémités des chromosomes linéaires sont appelées télomères et sont constituées de séquences répétitives non codantes. Les télomères protègent les séquences codantes contre la perte lorsque les cellules continuent de se diviser. Chez l'homme, une séquence de six paires de bases, TTAGGG, est répétée 100 à 1 000 fois pour former le télomère. La découverte de l'enzyme télomérase (Figure\(\PageIndex{6}\)) a permis de mieux comprendre comment les extrémités des chromosomes sont maintenues. La télomérase contient une partie catalytique et une matrice d'ARN intégrée. Il se fixe à l'extrémité du chromosome et des bases complémentaires à la matrice d'ARN sont ajoutées à l'extrémité 3' du brin d'ADN. Une fois que l'extrémité 3' de la matrice des brins retardataires est suffisamment allongée, l'ADN polymérase peut ajouter les nucléotides complémentaires aux extrémités des chromosomes. De cette façon, les extrémités des chromosomes sont répliquées. Chez l'homme, la télomérase est généralement active dans les cellules germinales et les cellules souches adultes ; elle n'est pas active dans les cellules somatiques adultes et peut être associée au vieillissement de ces cellules. Les microbes eucaryotes, y compris les champignons et les protozoaires, produisent également de la télomérase pour maintenir l'intégrité chromosomique. Pour sa découverte de la télomérase et de son action, Elizabeth Blackburn (1948—) a reçu le prix Nobel de médecine ou de physiologie en 2009.
| Propriété | bactéries | Eucaryotes |
|---|---|---|
| Structure du génome | Chromosome circulaire unique | Chromosomes linéaires multiples |
| Nombre d'origines par chromosome | Unique | Multiple |
| Taux de réplication | 1000 nucléotides par seconde | 100 nucléotides par seconde |
| Télomérase | Non présent | Présent |
| Élimination de l'amor | Pôle ADN II | RNase H |
| Allongation du fil | ADN pol III | pol δ, pol ε |
Exercice\(\PageIndex{3}\)
- En quoi l'origine de la réplication diffère-t-elle entre les eucaryotes et les procaryotes ?
- Quelles enzymes polymérases sont responsables de la synthèse de l'ADN lors de la réplication eucaryote ?
- Que trouve-t-on aux extrémités des chromosomes chez les eucaryotes et pourquoi ?
Réplication de l'ADN d'éléments extrachromosomaux : plasmides et virus
Pour copier leurs acides nucléiques, les plasmides et les virus utilisent fréquemment des variations du schéma de réplication de l'ADN décrit pour les génomes des procaryotes. Pour plus d'informations sur le large éventail de stratégies de réplication virale, voir Le cycle de vie viral.
Réplication en cercle
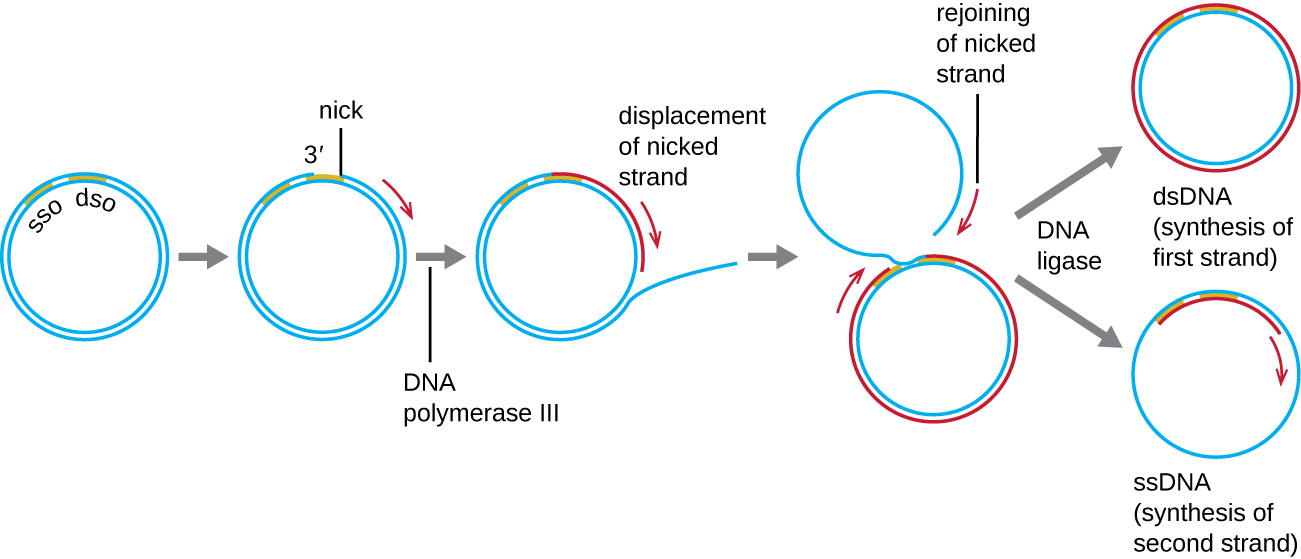
Alors que de nombreux plasmides bactériens (voir Caractéristiques uniques des cellules procaryotes) se répliquent par un processus similaire à celui utilisé pour copier le chromosome bactérien, d'autres plasmides, plusieurs bactériophages et certains virus d'eucaryotes utilisent la réplication en cercle roulant (Figure\(\PageIndex{7}\)). La nature circulaire des plasmides et la circularisation de certains génomes viraux lors de l'infection rendent cela possible. La réplication en cercle roulant commence par l'entaille enzymatique d'un brin de la molécule circulaire bicaténaire sur le site d'origine bicaténaire (dso). Chez les bactéries, l'ADN polymérase III se lie au groupe 3'-OH du brin entaillé et commence à répliquer l'ADN de manière unidirectionnelle en utilisant le brin non entaillé comme matrice, déplaçant ainsi le brin entaillé. L'achèvement de la réplication de l'ADN sur le site de l'entaille initiale entraîne le déplacement complet du brin entaillé, qui peut ensuite se recirculariser en une molécule d'ADN monocaténaire. L'ARN primase synthétise ensuite une amorce pour initier la réplication de l'ADN sur le site d'origine monocaténaire (sso) de la molécule d'ADN monocaténaire (SSDNA), ce qui donne une molécule d'ADN double brin (dSDNA) identique à l'autre molécule d'ADN circulaire.
Exercice\(\PageIndex{4}\)
Y a-t-il un fil à la traîne dans la réplication en cercle roulant ? Pourquoi ou pourquoi pas ?
Concepts clés et résumé
- Le processus de réplication de l'ADN est semi-conservateur, ce qui donne lieu à deux molécules d'ADN, chacune ayant un brin d'ADN parental et un brin nouvellement synthétisé.
- Chez les bactéries, l'initiation de la réplication se produit à l'origine de la réplication, où l'ADN superenroulé est déroulé par de l'ADN gyrase, fabriqué en simple brin par de l'hélicase et lié par une protéine de liaison monocaténaire pour maintenir son monocaténaire État échoué. La primase synthétise une courte amorce d'ARN, fournissant un groupe 3'-OH libre auquel l'ADN polymérase III peut ajouter des nucléotides d'ADN.
- Pendant l'élongation, le brin principal de l'ADN est synthétisé en continu à partir d'une seule amorce. Le brin en retard est synthétisé de manière discontinue en courts fragments d'Okazaki, chacun nécessitant sa propre amorce. Les amorces d'ARN sont retirées et remplacées par des nucléotides d'ADN par l'ADN polymérase I bactérienne, et l'ADN ligase comble les espaces entre ces fragments.
- L'arrêt de la réplication chez les bactéries implique la résolution des concatémères circulaires de l'ADN par la topoisomérase IV pour libérer les deux copies du chromosome circulaire.
- Les eucaryotes possèdent généralement plusieurs chromosomes linéaires, chacun ayant de multiples origines de réplication. Dans l'ensemble, la réplication chez les eucaryotes est similaire à celle des procaryotes.
- La nature linéaire des chromosomes eucaryotes nécessite la présence de télomères pour protéger les gènes situés à l'extrémité des chromosomes. La télomérase étend les télomères, empêchant ainsi leur dégradation, dans certains types de cellules.
- La réplication en cercle roulant est un type de synthèse unidirectionnelle rapide de l'ADN d'une molécule d'ADN circulaire utilisée pour la réplication de certains plasmides.


