12.1 : Les microbes et les outils du génie génétique
- Page ID
- 187895
Objectifs d'apprentissage
- Identifier les outils de génétique moléculaire dérivés de microorganismes
- Décrire les méthodes utilisées pour créer des molécules d'ADN recombinant
- Décrire les méthodes utilisées pour introduire de l'ADN dans les cellules procaryotes
- Énumérer les types de bibliothèques génomiques et décrire leurs utilisations
- Décrire les méthodes utilisées pour introduire de l'ADN dans les cellules eucaryotes
Orientation clinique : 1ère partie
Kayla, une ingénieure électricienne de 24 ans passionnée de course à pied, vient de déménager de l'Arizona au New Hampshire pour prendre un nouvel emploi. Pendant ses week-ends de congé, elle aime explorer son nouvel environnement et faire de longues courses dans les forêts de pins. En juillet, elle a passé une semaine à faire de la randonnée dans les montagnes. Au début du mois d'août, Kayla a développé une faible fièvre, des maux de tête et de légères douleurs musculaires, et elle s'est sentie un peu fatiguée. Sans trop y penser, elle a pris de l'ibuprofène pour combattre ses symptômes et a juré de se reposer davantage.
Exercice\(\PageIndex{1}\)
Quels types de troubles médicaux peuvent être à l'origine des symptômes de Kayla ?
La science de l'utilisation des systèmes vivants au profit de l'humanité s'appelle la biotechnologie. Techniquement parlant, la domestication des plantes et des animaux par le biais de pratiques agricoles et de sélection est un type de biotechnologie. Cependant, dans un sens contemporain, nous associons la biotechnologie à la modification directe de la génétique d'un organisme afin d'obtenir des caractères souhaitables par le biais du génie génétique. Le génie génétique implique l'utilisation de la technologie de l'ADN recombinant, le processus par lequel une séquence d'ADN est manipulée in vitro, créant ainsi des molécules d'ADN recombinant contenant de nouvelles combinaisons de matériel génétique. L'ADN recombinant est ensuite introduit dans un organisme hôte. Si l'ADN introduit provient d'une espèce différente, l'organisme hôte est désormais considéré comme transgénique.
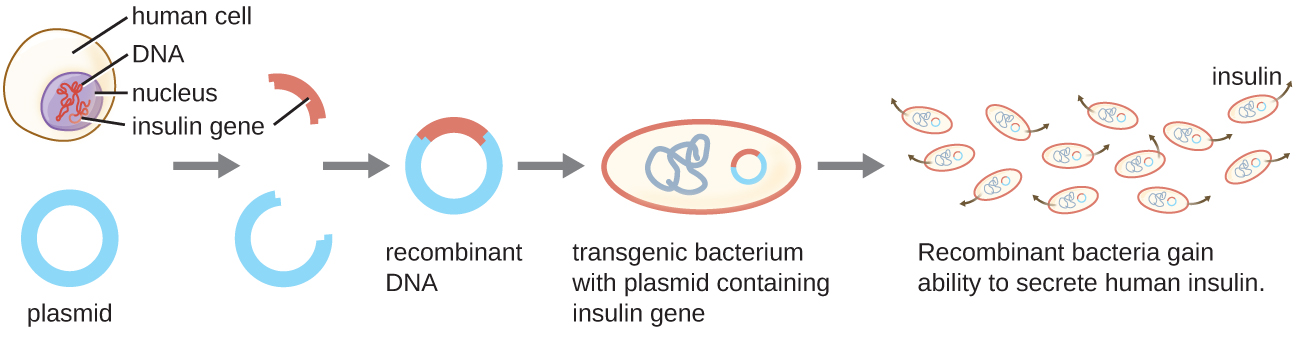
La souche bactérienne qui produit de l'insuline humaine est un exemple de microorganisme transgénique (Figure\(\PageIndex{1}\)). Le gène de l'insuline d'origine humaine a été inséré dans un plasmide. Ce plasmide d'ADN recombinant a ensuite été inséré dans des bactéries. Par conséquent, ces microbes transgéniques sont capables de produire et de sécréter de l'insuline humaine. De nombreux procaryotes sont capables d'acquérir de l'ADN étranger et d'intégrer des gènes fonctionnels dans leur propre génome par « accouplement » avec d'autres cellules (conjugaison), par infection virale (transduction) et par prélèvement d'ADN de l'environnement (transformation). Rappelons que ces mécanismes sont des exemples de transfert horizontal de gènes, c'est-à-dire le transfert de matériel génétique entre des cellules d'une même génération.
Clonage moléculaire
Herbert Boyer et Stanley Cohen ont démontré pour la première fois le processus complet de clonage moléculaire en 1973 lorsqu'ils ont cloné avec succès des gènes de la grenouille à griffes africaine (Xenopus laevis) dans un plasmide bactérien qui a ensuite été introduit dans la bactérie hôte Escherichia coli. Le clonage moléculaire est un ensemble de méthodes utilisées pour construire de l'ADN recombinant et l'intégrer dans un organisme hôte ; il fait appel à un certain nombre d'outils moléculaires dérivés de microorganismes.
Enzymes de restriction et ligases
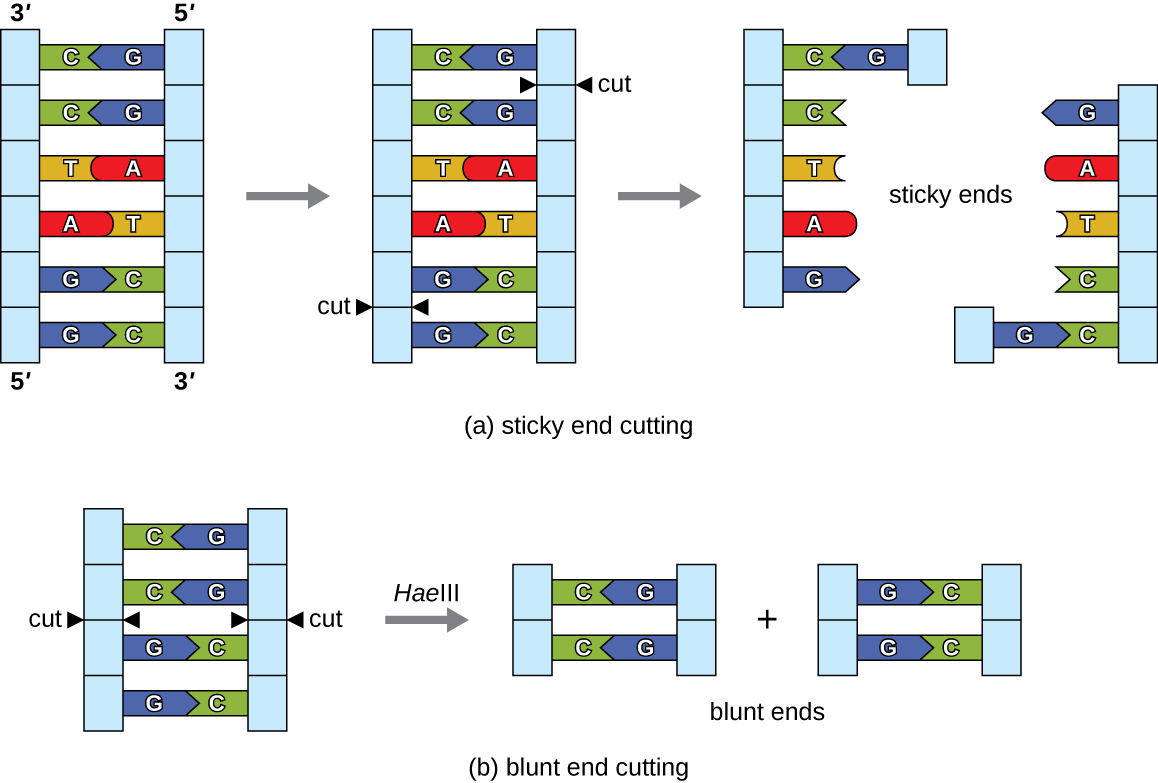
Dans la technologie de l'ADN recombinant, les molécules d'ADN sont manipulées à l'aide d'enzymes naturelles dérivées principalement de bactéries et de virus. La création de molécules d'ADN recombinantes est possible grâce à l'utilisation d'endonucléases de restriction naturelles (enzymes de restriction), des enzymes bactériennes produites comme mécanisme de protection pour couper et détruire l'ADN cytoplasmique étranger qui est le plus souvent le résultat d'une infection par un bactériophage. Stewart Linn et Werner Arber ont découvert les enzymes de restriction dans leurs études des années 1960 sur la façon dont E. coli limite la réplication des bactériophages lors d'une infection. Aujourd'hui, nous utilisons largement des enzymes de restriction pour découper des fragments d'ADN qui peuvent ensuite être épissés dans une autre molécule d'ADN pour former des molécules recombinantes. Chaque enzyme de restriction coupe l'ADN au niveau d'un site de reconnaissance caractéristique, une séquence d'ADN spécifique, généralement palindromique, généralement d'une longueur comprise entre quatre et six paires de bases. Un palindrome est une séquence de lettres qui se lit de la même manière vers l'avant et vers l'arrière. (Le mot « niveau » est un exemple de palindrome.) Les séquences d'ADN palindromique contiennent les mêmes séquences de base dans la direction 5 à 3 pieds sur un brin que dans la direction 5 à 3 pieds sur le brin complémentaire. Une enzyme de restriction reconnaît le palindrome de l'ADN et coupe chaque colonne vertébrale à des positions identiques dans le palindrome. Certaines enzymes de restriction se coupent pour produire des molécules présentant des surplombs complémentaires (extrémités collantes), tandis que d'autres coupent sans générer de tels surplombs, produisant plutôt des extrémités émoussées (Figure\(\PageIndex{2}\)).
Les molécules ayant des extrémités collantes complémentaires peuvent facilement se recuire ou former des liaisons hydrogène entre des bases complémentaires au niveau de leurs extrémités collantes. L'étape de recuit permet l'hybridation des surplombs monocaténaires. L'hybridation fait référence à l'assemblage de deux brins uniques complémentaires d'ADN. Les extrémités émoussées peuvent également se fixer ensemble, mais moins efficacement que les extrémités collantes en raison de l'absence de porte-à-faux complémentaires facilitant le processus. Dans les deux cas, la ligation par l'ADN ligase peut ensuite rejoindre les deux squelettes sucre-phosphate de l'ADN par liaison covalente, faisant de la molécule un double brin continu. En 1972, Paul Berg, biochimiste de Stanford, a été le premier à produire une molécule d'ADN recombinant à l'aide de cette technique, en combinant le virus du singe SV40 avec le bactériophage lambda d'E. coli pour créer un hybride.
Plasmides
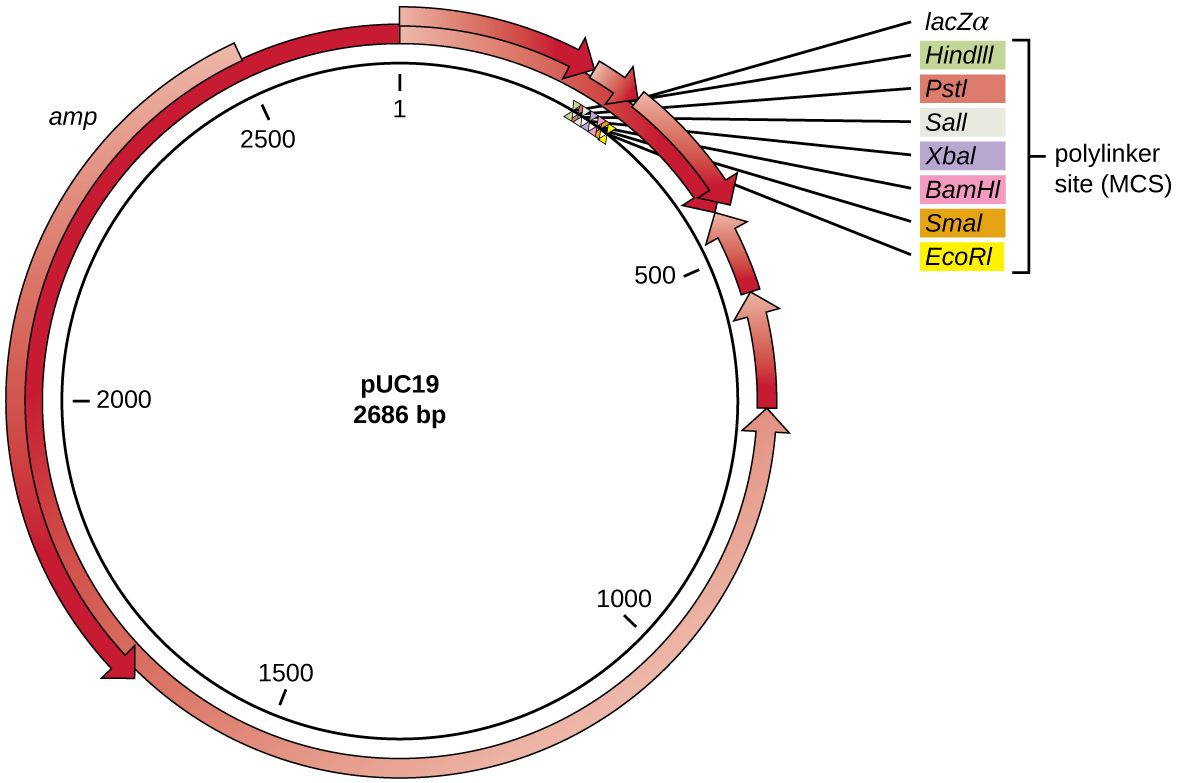
Après la digestion par restriction, les gènes d'intérêt sont généralement insérés dans des plasmides, de petits morceaux d'ADN bicaténaire généralement circulaires qui se répliquent indépendamment du chromosome bactérien (voir Caractéristiques uniques des cellules procaryotes). Dans la technologie de l'ADN recombinant, les plasmides sont souvent utilisés comme vecteurs, des molécules d'ADN qui transportent des fragments d'ADN d'un organisme à un autre. Les plasmides utilisés comme vecteurs peuvent être génétiquement modifiés par des chercheurs et des sociétés de fournitures scientifiques pour avoir des propriétés spécialisées, comme l'illustre le vecteur plasmidique pUC19 couramment utilisé (Figure\(\PageIndex{3}\)). Certains vecteurs plasmidiques contiennent des gènes qui confèrent une résistance aux antibiotiques ; ces gènes de résistance permettent aux chercheurs de trouver facilement des colonies contenant des plasmides en les plaçant sur des milieux contenant l'antibiotique correspondant. L'antibiotique tue toutes les cellules hôtes qui ne contiennent pas le vecteur plasmidique souhaité, mais celles qui contiennent le vecteur peuvent survivre et se développer.
Les vecteurs plasmidiques utilisés pour le clonage possèdent généralement un site polylinker ou un site de clonage multiple (MCS). Un site polylinker est une courte séquence contenant plusieurs sites de reconnaissance d'enzymes de restriction uniques qui sont utilisés pour insérer de l'ADN dans le plasmide après digestion par restriction de l'ADN et du plasmide. La présence de ces multiples sites de reconnaissance des enzymes de restriction au sein du site polylinker rend le vecteur plasmidique polyvalent, de sorte qu'il peut être utilisé pour de nombreuses expériences de clonage impliquant différentes enzymes de restriction.
Ce site polylinker se trouve souvent dans un gène rapporteur, une autre séquence génique modifiée artificiellement dans le plasmide qui code une protéine qui permet de visualiser l'insertion de l'ADN. Le gène rapporteur permet au chercheur de distinguer les cellules hôtes qui contiennent des plasmides recombinants avec des fragments d'ADN clonés des cellules hôtes qui ne contiennent que le vecteur plasmidique non recombinant. Le gène rapporteur le plus couramment utilisé dans les vecteurs plasmidiques est le gène lacZ bactérien codant pour la bêta-galactosidase, une enzyme qui dégrade naturellement le lactose mais qui peut également dégrader un analogue synthétique incolore X-gal, produisant ainsi des colonies bleues sur des milieux contenant du X-Gal. Le gène rapporteur LacZ est désactivé lorsque l'ADN recombinant est épissé dans le plasmide. Comme la protéine LacZ n'est pas produite lorsque le gène est désactivé, X-gal n'est pas dégradée et des colonies blanches sont produites, qui peuvent ensuite être isolées. Cette méthode de dépistage bleu-blanc est décrite plus loin et illustrée sur la figure\(\PageIndex{4}\). En plus de ces caractéristiques, certains plasmides sont prédigérés et contiennent une enzyme liée au plasmide linéarisé pour faciliter la ligation après l'insertion de fragments d'ADN étrangers.
Clonage moléculaire par transformation
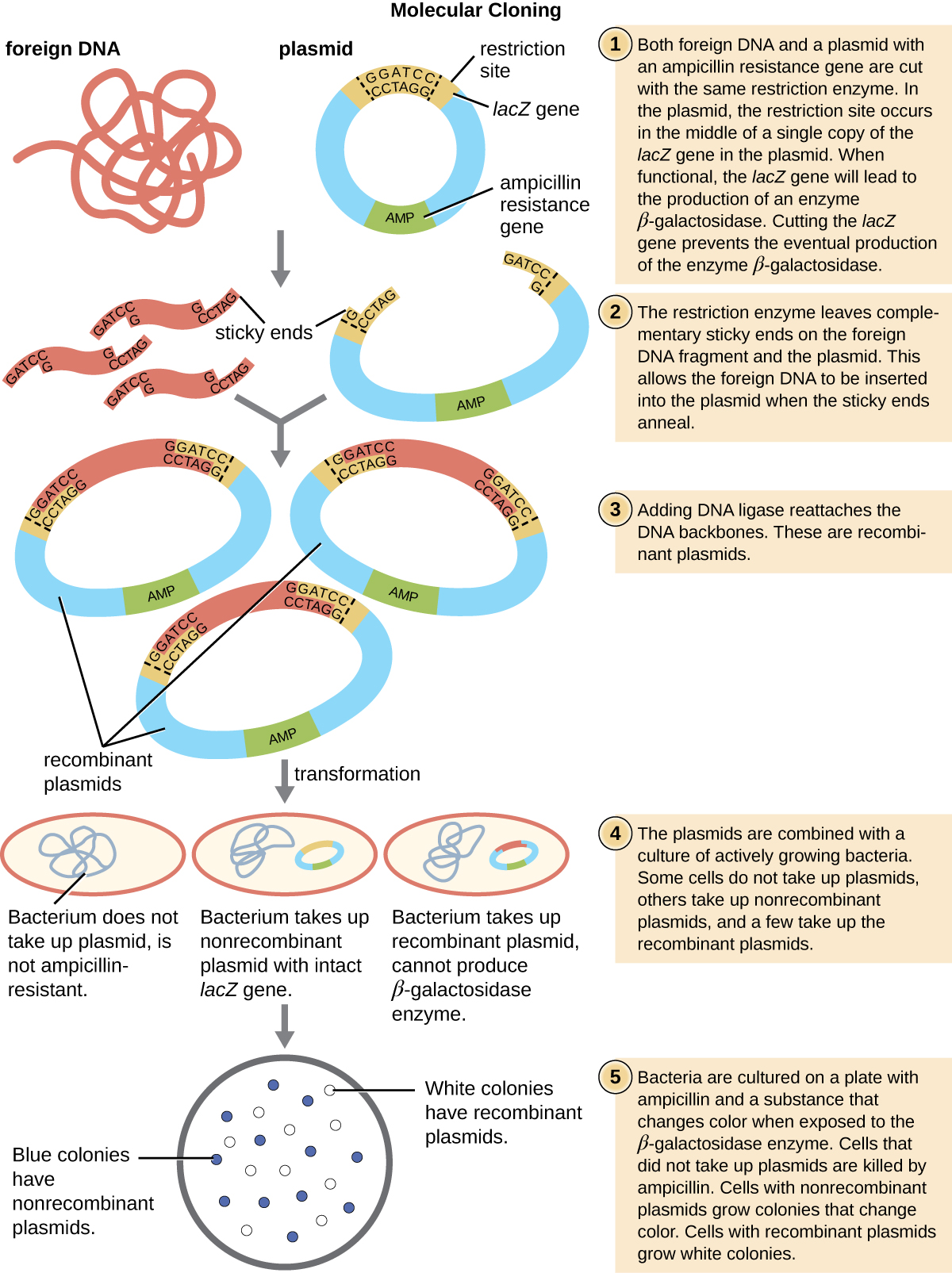
Le mécanisme le plus couramment utilisé pour introduire des plasmides modifiés dans une cellule bactérienne est la transformation, un processus par lequel les bactéries absorbent l'ADN libre de leur environnement. Dans la nature, l'ADN libre provient généralement d'autres cellules bactériennes lysées ; en laboratoire, de l'ADN libre sous forme de plasmides recombinants est introduit dans l'environnement de la cellule.
Certaines bactéries, telles que Bacillus spp., sont naturellement compétentes, ce qui signifie qu'elles sont capables d'absorber de l'ADN étranger. Cependant, toutes les bactéries ne sont pas naturellement compétentes. Dans la plupart des cas, les bactéries doivent être rendues artificiellement compétentes en laboratoire en augmentant la perméabilité de la membrane cellulaire. Cela peut être réalisé par des traitements chimiques qui neutralisent les charges sur la membrane cellulaire ou en exposant les bactéries à un champ électrique qui crée des pores microscopiques dans la membrane cellulaire. Ces méthodes produisent des bactéries chimiquement compétentes ou électrocompétentes, respectivement.
Conformément au protocole de transformation, les cellules bactériennes sont déposées sur un milieu contenant des antibiotiques afin d'inhiber la croissance des nombreuses cellules hôtes qui n'ont pas été transformées par le plasmide conférant une résistance aux antibiotiques. Une technique appelée criblage bleu-blanc est ensuite utilisée pour les vecteurs plasmidiques codant pour LacZ tels que pUC19. Les colonies bleues possèdent une enzyme bêta-galactosidase fonctionnelle parce que le gène lacZ est ininterrompu, aucun ADN étranger n'étant inséré dans le site polylinker. Ces colonies résultent généralement du fait que le plasmide digéré et linéarisé se relige à lui-même. Cependant, les colonies blanches sont dépourvues d'une enzyme bêta-galactosidase fonctionnelle, ce qui indique l'insertion d'ADN étranger dans le site polylinker du vecteur plasmidique, perturbant ainsi le gène lacZ. Ainsi, les colonies blanches résultant de ce criblage bleu-blanc contiennent des plasmides avec un insert et peuvent être criblées davantage pour caractériser l'ADN étranger. Pour s'assurer que le bon ADN a été incorporé dans le plasmide, l'insert d'ADN peut ensuite être séquencé.
Visionnez une animation sur le clonage moléculaire réalisée par le DNA Learning Center.
Exercice\(\PageIndex{2}\)
Dans le cadre du dépistage bleu-blanc, que signifie une colonie bleue et pourquoi est-elle bleue ?
Clonage moléculaire par conjugaison ou transduction
Le processus bactérien de conjugaison (voir Comment les procaryotes asexués atteignent la diversité génétique) peut également être manipulé pour le clonage moléculaire. Les plasmides F, ou plasmides de fertilité, sont transférés entre des cellules bactériennes par le biais du processus de conjugaison. L'ADN recombinant peut être transféré par conjugaison lorsque des cellules bactériennes contenant un plasmide F recombinant sont mélangées à des cellules bactériennes compatibles dépourvues de plasmide. Les plasmides F codent une structure de surface appelée pilus F qui facilite le contact entre une cellule contenant un plasmide F et une cellule dépourvue de plasmide F. Au contact, un pont cytoplasmique se forme entre les deux cellules et la cellule contenant le plasmide F réplique son plasmide, transférant une copie du plasmide F recombinant vers la cellule réceptrice. Une fois qu'elle a reçu le plasmide F recombinant, la cellule réceptrice peut produire son propre pilus F et faciliter le transfert du plasmide F recombinant vers une cellule supplémentaire. L'utilisation de la conjugaison pour transférer des plasmides F recombinants vers des cellules réceptrices est un autre moyen efficace d'introduire des molécules d'ADN recombinant dans les cellules hôtes.
Les bactériophages peuvent également être utilisés pour introduire de l'ADN recombinant dans les cellules bactériennes hôtes en manipulant le processus de transduction (voir Comment les procaryotes asexués atteignent la diversité génétique). En laboratoire, les fragments d'ADN d'intérêt peuvent être transformés en phagémides, qui sont des plasmides dont les séquences phagiques permettent de les encapsuler dans des bactériophages. Les cellules bactériennes peuvent ensuite être infectées par ces bactériophages afin que les phagémides recombinants puissent être introduits dans les cellules bactériennes. Selon le type de phage, l'ADN recombinant peut être intégré au génome de la bactérie hôte (lysogénie), ou il peut exister sous forme de plasmide dans le cytoplasme de l'hôte.
Exercice\(\PageIndex{2}\)
- Quelle est la fonction initiale d'une enzyme de restriction ?
- Quels sont les deux procédés utilisés pour introduire de l'ADN recombinant dans une cellule hôte bactérienne ?
- Distinguer les utilisations d'un gène de résistance aux antibiotiques et d'un gène rapporteur dans un vecteur plasmidique.
Création d'une bibliothèque génomique
Le clonage moléculaire peut également être utilisé pour générer une bibliothèque génomique. La bibliothèque est une copie complète (ou presque complète) du génome d'un organisme contenue sous forme de plasmides d'ADN recombinant modifiés pour former des clones uniques de bactéries. Le fait de disposer d'une telle bibliothèque permet au chercheur de créer de grandes quantités de chaque fragment en cultivant la bactérie hôte de ce fragment. Ces fragments peuvent être utilisés pour déterminer la séquence de l'ADN et la fonction de tout gène présent.
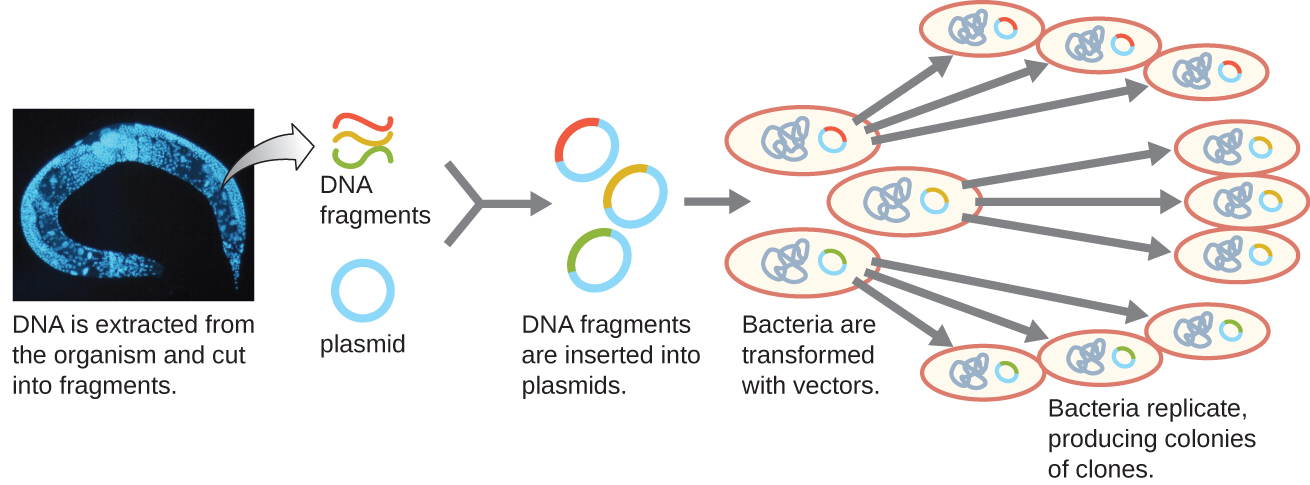
Une méthode pour générer une bibliothèque génomique consiste à ligaturer des fragments génomiques individuels digérés par une enzyme de restriction en vecteurs plasmidiques coupés avec la même enzyme de restriction (Figure\(\PageIndex{5}\)). Après transformation en hôte bactérien, chaque cellule bactérienne transformée absorbe un seul plasmide recombinant et se développe en une colonie de cellules. Toutes les cellules de cette colonie sont des clones identiques et portent le même plasmide recombinant. La bibliothèque qui en résulte est une collection de colonies, chacune contenant un fragment du génome de l'organisme d'origine, chacune étant séparée et distincte et pouvant être utilisée pour une étude plus approfondie. Cela permet aux chercheurs de sélectionner ces différents clones afin de découvrir celui qui contient un gène d'intérêt issu du génome de l'organisme d'origine.
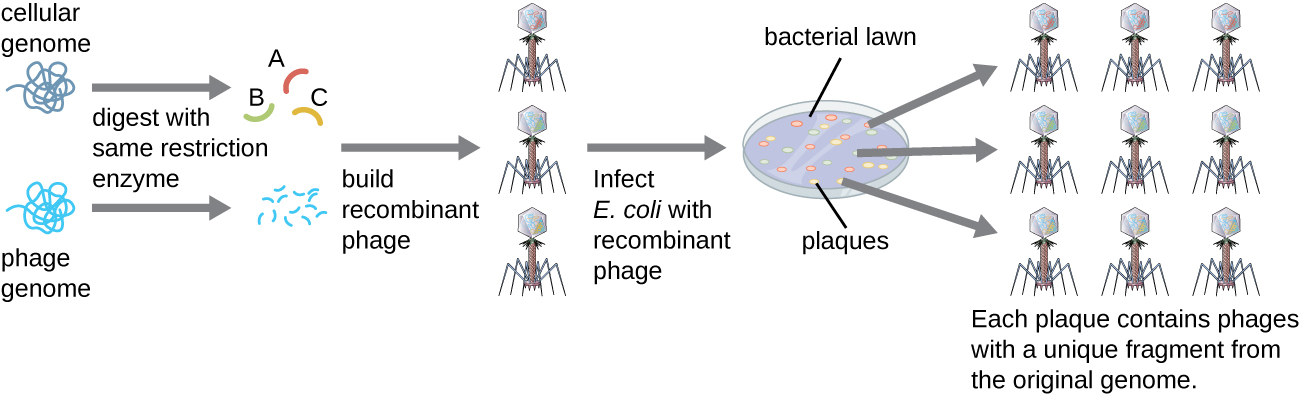
Pour construire une bibliothèque génomique à partir de plus gros fragments d'ADN génomique, un bactériophage d'E. coli, tel que le lambda, peut être utilisé comme hôte (Figure\(\PageIndex{6}\)). L'ADN génomique peut être cisaillé ou digéré par voie enzymatique et ligaturé dans un vecteur d'ADN lambda de bactériophage prédigéré. Ces molécules d'ADN phagique recombinant peuvent ensuite être conditionnées dans des particules phagiques et utilisées pour infecter les cellules hôtes d'E. coli sur une plaque. Au cours de l'infection au sein de chaque cellule, chaque phage recombinant se reproduit à de nombreuses reprises et lyse la pelouse d'E. coli pour former une plaque. Ainsi, chaque plaque d'une bibliothèque de phages représente un phage recombinant unique contenant un fragment d'ADN génomique distinct. Les plaques peuvent ensuite être examinées plus avant pour rechercher des gènes d'intérêt. L'un des avantages de la production d'une bibliothèque utilisant des phages plutôt que des plasmides est qu'une particule de phage contient un insert d'ADN étranger beaucoup plus important qu'un vecteur plasmidique, ce qui nécessite un nombre beaucoup plus petit de cultures pour représenter pleinement l'ensemble du génome de l'organisme d'origine.
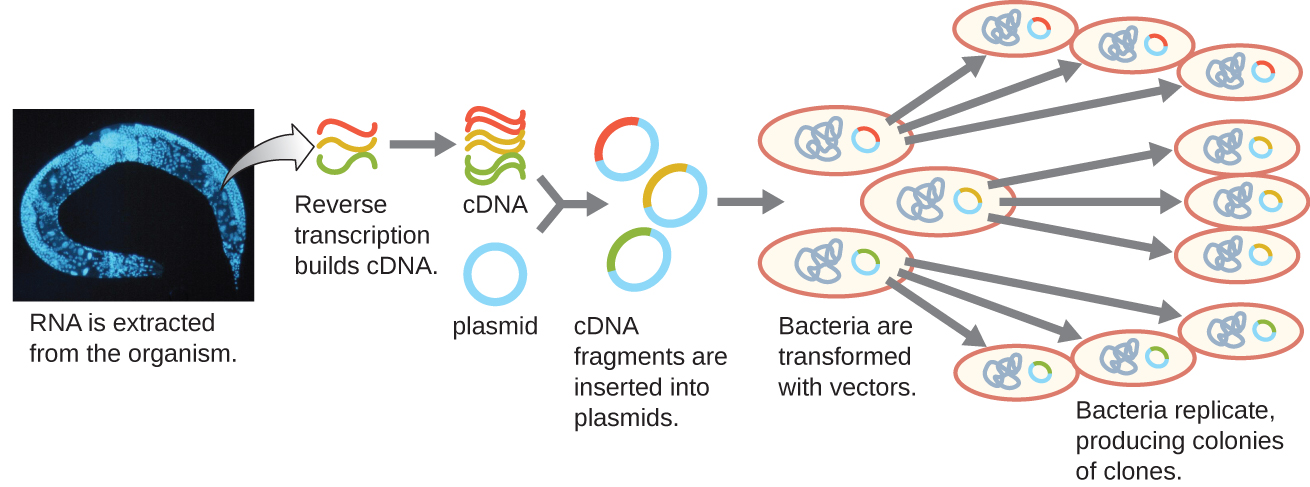
Pour se concentrer sur les gènes exprimés dans un organisme ou même un tissu, les chercheurs construisent des bibliothèques en utilisant l'ARN messager (ARNm) de l'organisme plutôt que son ADN génomique. Alors que toutes les cellules d'un même organisme auront le même ADN génomique, différents tissus expriment différents gènes, produisant différents compléments d'ARNm. Par exemple, l'ADN génomique de toutes les cellules humaines contient le gène de l'insuline, mais seules les cellules du pancréas expriment l'ARNm qui dirige la production d'insuline. Comme l'ARNm ne peut pas être cloné directement, en laboratoire, l'ARNm doit être utilisé comme matrice par l'enzyme rétrovirale transcriptase inverse pour fabriquer de l'ADN complémentaire (ADNc). L'ensemble complet d'ARNm d'une cellule peut être transcrit en sens inverse en molécules d'ADNc, qui peuvent être utilisées comme matrice pour l'ADN polymérase afin de produire des copies d'ADN double brin ; ces fragments peuvent ensuite être ligaturés soit dans des vecteurs plasmidiques, soit dans des bactériophages pour produire une banque d'ADNc. L'avantage d'une banque d'ADNc est qu'elle contient de l'ADN provenant uniquement des gènes exprimés dans la cellule. Cela signifie que les introns, les séquences de contrôle telles que les promoteurs et l'ADN qui ne sont pas destinés à être traduits en protéines ne sont pas représentés dans la bibliothèque. L'accent mis sur les séquences traduites signifie que la bibliothèque ne peut pas être utilisée pour étudier la séquence et la structure du génome dans son intégralité. La construction d'une banque génomique d'ADNc est illustrée dans la figure\(\PageIndex{7}\).
Exercice\(\PageIndex{3}\)
- Quels sont les hôtes des bibliothèques génomiques décrites ?
- Qu'est-ce que l'ADNc ?
Introduction de molécules recombinantes chez des hôtes eucaryotes
L'utilisation de bactéries hôtes à des fins de génie génétique a jeté les bases de la technologie de l'ADN recombinant ; toutefois, les chercheurs se sont également montrés très intéressés par le génie génétique des cellules eucaryotes, en particulier celles des plantes et des animaux. L'introduction de molécules d'ADN recombinant chez des hôtes eucaryotes est appelée transfection. Les plantes génétiquement modifiées, appelées plantes transgéniques, présentent un intérêt significatif à des fins agricoles et pharmaceutiques. La première plante transgénique vendue dans le commerce était la tomate à maturation retardée Flavr Savr, mise sur le marché en 1994. Des animaux génétiquement modifiés ont également été produits avec succès, ce qui a donné lieu, par exemple, à des porcs à valeur nutritive augmentée1 et à des chèvres qui sécrètent des produits pharmaceutiques dans leur lait. 2
Électroporation
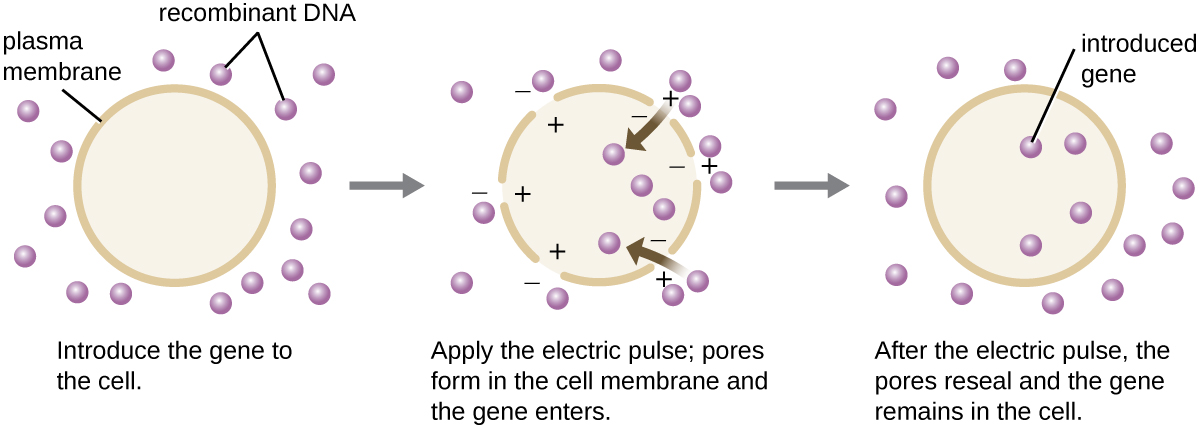
Comparativement aux cellules bactériennes, les cellules eucaryotes ont tendance à être moins aptes à héberger des molécules d'ADN recombinant. Comme les eucaryotes ne sont généralement ni capables d'absorber de l'ADN étranger ni de conserver des plasmides, la transfection d'hôtes eucaryotes est beaucoup plus difficile et nécessite des techniques plus intrusives pour réussir. L'électroporation est l'une des méthodes utilisées pour transfecter des cellules en culture cellulaire. Une brève impulsion électrique induit la formation de pores transitoires dans les bicouches de phospholipides des cellules à travers lesquelles le gène peut être introduit. Dans le même temps, l'impulsion électrique génère une charge positive de courte durée sur un côté de l'intérieur de la cellule et une charge négative sur le côté opposé ; la différence de charge attire les molécules d'ADN chargées négativement dans la cellule (Figure\(\PageIndex{8}\)).
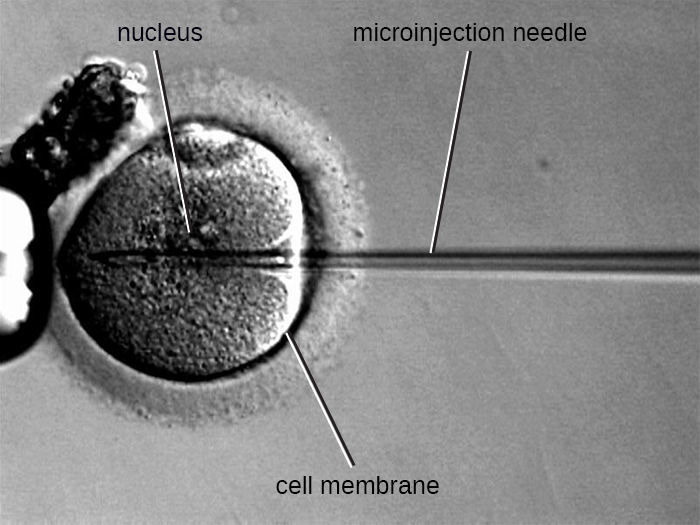
Microinjection
Une autre méthode de transfection est appelée microinjection. Les cellules eucaryotes étant généralement plus grosses que celles des procaryotes, des fragments d'ADN peuvent parfois être injectés directement dans le cytoplasme à l'aide d'une micropipette en verre, comme le montre la figure\(\PageIndex{9}\).
Armes Gene
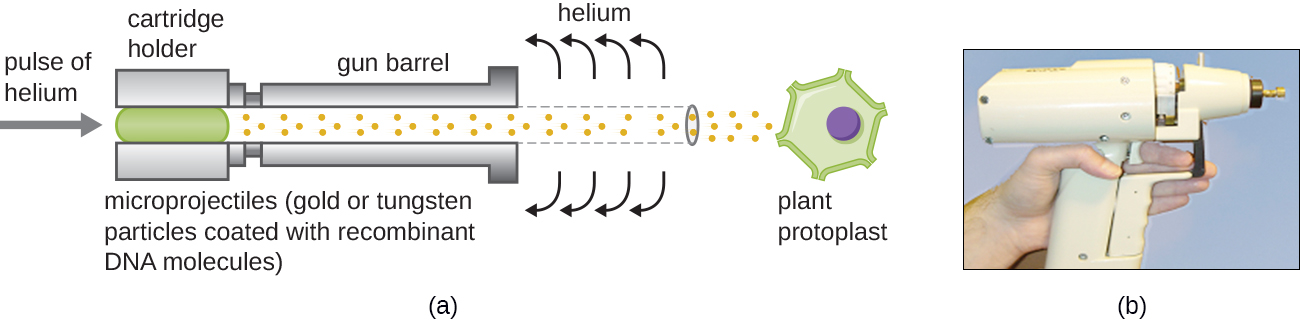
La transfection des cellules végétales peut être encore plus difficile que celle des cellules animales en raison de l'épaisseur de leurs parois cellulaires. L'une des approches consiste à traiter les cellules végétales avec des enzymes pour éliminer leurs parois cellulaires, produisant ainsi des protoplastes. Ensuite, un canon à gènes est utilisé pour lancer des particules d'or ou de tungstène recouvertes de molécules d'ADN recombinant dans les protoplastes végétaux à grande vitesse. Les cellules protoplastes réceptrices peuvent ensuite se récupérer et être utilisées pour générer de nouvelles plantes transgéniques (Figure\(\PageIndex{10}\)).
Vecteurs de navette
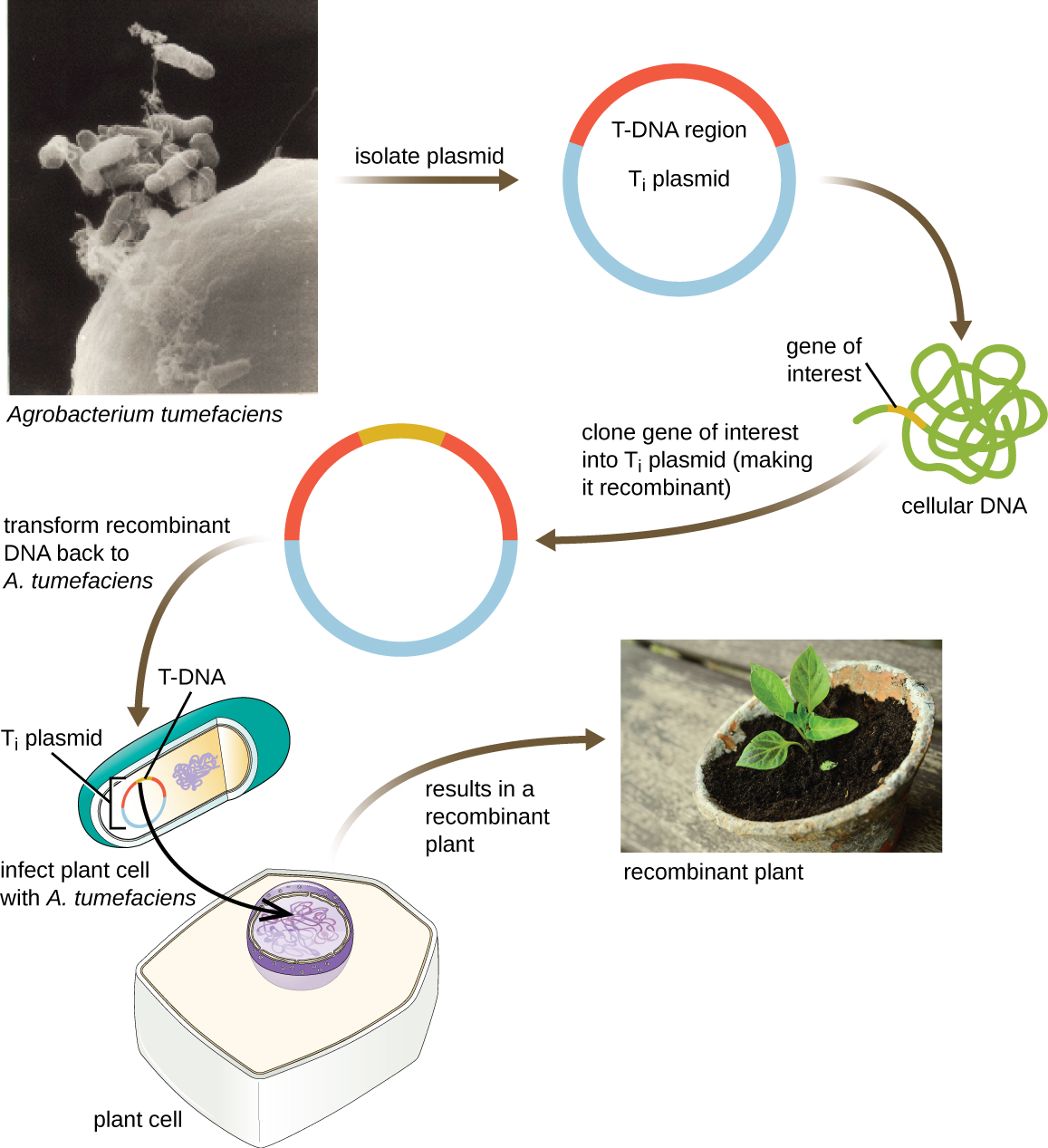
Une autre méthode de transfection de plantes fait appel à des vecteurs navettes, des plasmides qui peuvent se déplacer entre les cellules bactériennes et les cellules eucaryotes. Les plasmides induisant des tumeurs (T i) provenant de la bactérie Agrobacterium tumefaciens sont couramment utilisés comme vecteurs navettes pour l'incorporation de gènes dans les plantes (Figure\(\PageIndex{11}\)). Dans la nature, les plasmides T i d'A. tumefaciens provoquent le développement de tumeurs chez les plantes lorsqu'elles sont transférées des cellules bactériennes aux cellules végétales. Les chercheurs ont pu manipuler ces plasmides naturels pour éliminer leurs gènes responsables de tumeurs et insérer des fragments d'ADN souhaitables. Les plasmides T i recombinants qui en résultent peuvent être transférés dans le génome de la plante par transfert naturel des plasmides T i de la bactérie à la plante hôte. Une fois à l'intérieur de la cellule hôte de la plante, le gène d'intérêt se recombine dans le génome de la cellule végétale.
Vecteurs viraux
Des vecteurs viraux peuvent également être utilisés pour transfecter des cellules eucaryotes. En fait, cette méthode est souvent utilisée en thérapie génique (voir Thérapie génique) pour introduire des gènes sains chez des patients humains atteints de maladies résultant de mutations génétiques. Les gènes viraux peuvent être supprimés et remplacés par le gène à transmettre au patient3 ; le virus infecte ensuite la cellule hôte et introduit l'ADN étranger dans le génome de la cellule ciblée. Les adénovirus sont souvent utilisés à cette fin car ils peuvent être cultivés jusqu'à des titres élevés et peuvent infecter à la fois les cellules hôtes qui ne se divisent pas et qui se divisent. Cependant, l'utilisation de vecteurs viraux pour la thérapie génique peut présenter certains risques pour les patients, comme indiqué dans la section Thérapie génique.
Exercice\(\PageIndex{4}\)
- Quelles sont les méthodes utilisées pour introduire des vecteurs d'ADN recombinant dans des cellules animales ?
- Comparez et opposez les vecteurs navettes et les vecteurs viraux.
Concepts clés et résumé
- La biotechnologie est la science qui utilise les systèmes vivants au profit de l'humanité. Ces dernières années, la capacité de modifier directement le génome d'un organisme par le biais du génie génétique a été rendue possible grâce aux progrès de la technologie de l'ADN recombinant, qui permet aux chercheurs de créer des molécules d'ADN recombinant avec de nouvelles combinaisons de matériel génétique.
- Le clonage moléculaire implique des méthodes utilisées pour construire de l'ADN recombinant et faciliter sa réplication dans les organismes hôtes. Ces méthodes incluent l'utilisation d'enzymes de restriction (pour couper à la fois l'ADN étranger et les vecteurs plasmidiques), la ligature (pour coller des fragments d'ADN) et l'introduction d'ADN recombinant dans un organisme hôte (souvent des bactéries).
- Le criblage bleu-blanc permet de sélectionner des transformants bactériens contenant des plasmides recombinants en utilisant le phénotype d'un gène rapporteur désactivé par l'insertion du fragment d'ADN.
- Les banques génomiques peuvent être créées en clonant des fragments génomiques d'un organisme dans des vecteurs plasmidiques ou dans des bactériophages.
- Des banques d'ADNc peuvent être générées pour représenter les molécules d'ARNm exprimées dans une cellule à un point donné.
- La transfection d'hôtes eucaryotes peut être réalisée par diverses méthodes utilisant l'électroporation, les pistolets géniques, la microinjection, les vecteurs navettes et les vecteurs viraux.
Notes
- 1 Liangxue Lai, Jing X. Kang, Rongfeng Li, Jingdong Wang, William T. Witt, Hwan Yul Yong, Yanhong Hao et coll. « Génération de porcs transgéniques clonés riches en acides gras oméga-3. » Nature Biotechnology 24 no 4 (2006) : 435—436.
- 2 Raylene Ramos Moura, Luciana Magalhães Melo et Vicente José de Figueirêdo Freitas. « Production de protéines recombinantes dans le lait de chèvres transgéniques et non transgéniques. » Archives brésiliennes de biologie et de technologie 54 no 5 (2011) : 927—938.
- 3 William S. M. Wold et Karoly Toth. « Vecteurs d'adénovirus pour la thérapie génique, la vaccination et la thérapie génique du cancer. » Thérapie génique actuelle 13 n° 6 (2013) : 421.


