10.3 : Génomique et protéomique
- Page ID
- 186143
L'étude des acides nucléiques a débuté par la découverte de l'ADN, s'est ensuite étendue à l'étude des gènes et de petits fragments, et a maintenant explosé dans le domaine de la génomique. La génomique est l'étude de génomes entiers, y compris de l'ensemble complet des gènes, de leur séquence et de leur organisation nucléotidiques, ainsi que de leurs interactions au sein d'une espèce et avec d'autres espèces. Les progrès de la génomique ont été rendus possibles grâce à la technologie de séquençage de l'ADN. Tout comme les technologies de l'information ont conduit à Google Maps qui nous permet d'obtenir des informations détaillées sur des lieux dans le monde entier, les informations génomiques sont utilisées pour créer des cartes similaires de l'ADN de différents organismes.
Cartographie des gén
La cartographie du génome est le processus qui permet de localiser les gènes sur chaque chromosome. Les cartes créées sont comparables à celles que nous utilisons pour naviguer dans les rues. Une carte génétique est une illustration qui répertorie les gènes et leur localisation sur un chromosome. Les cartes génétiques fournissent une vue d'ensemble (similaire à une carte des autoroutes inter-États) et utilisent des marqueurs génétiques (similaires aux points de repère). Un marqueur génétique est un gène ou une séquence sur un chromosome qui présente un lien génétique avec un trait d'intérêt. Le marqueur génétique a tendance à être hérité avec le gène d'intérêt, et l'une des mesures de la distance qui les sépare est la fréquence de recombinaison au cours de la méiose. Les premiers généticiens ont appelé cette analyse des liens.
Les cartes physiques permettent d'accéder aux détails intimes de petites régions des chromosomes (comme une carte routière détaillée) (Figure\(\PageIndex{1}\)). Une carte physique est une représentation de la distance physique, en nucléotides, entre des gènes ou des marqueurs génétiques. Des cartes de couplage génétique et des cartes physiques sont nécessaires pour dresser un tableau complet du génome. Le fait de disposer d'une carte complète du génome permet aux chercheurs d'étudier plus facilement les gènes individuels. Les cartes du génome humain aident les chercheurs à identifier les gènes responsables de maladies humaines liées à des maladies telles que le cancer, les maladies cardiaques et la fibrose kystique, pour n'en nommer que quelques-unes. En outre, la cartographie du génome peut être utilisée pour aider à identifier les organismes présentant des caractéristiques bénéfiques, tels que les microbes capables d'éliminer les polluants ou même de prévenir la pollution. La recherche impliquant la cartographie du génome végétal peut mener à la mise au point de méthodes permettant d'obtenir de meilleurs rendements des cultures ou à la mise au point de plantes mieux adaptées au changement climatique.
![Schéma montrant un chromosome humain avec des bandes révélées par une coloration de Giemsa. Les bandes sont étiquetées avec Xp et un chiffre sur le bras court et Xq et un chiffre sur le bras long. Certains gènes se trouvent à l'intérieur de certaines bandes. Ces gènes sont marqués sur la droite : anémie B de Fanconi, syndrome de Wiskott-Aldrich, maladie de Pelizaeus-Merzbacher, syndrome du X fragile et déficit en G6PD [0].](https://bio.libretexts.org/@api/deki/files/9384/Figure_10_03_01.jpg)
Les cartes génétiques fournissent les grandes lignes et les cartes physiques fournissent les détails. Il est facile de comprendre pourquoi les deux types de techniques de cartographie du génome sont importants pour donner une vue d'ensemble. Les informations obtenues par chaque technique sont utilisées en combinaison pour étudier le génome. La cartographie génomique est utilisée avec différents organismes modèles utilisés pour la recherche. La cartographie du génome est toujours un processus en cours et, à mesure que des techniques plus avancées seront mises au point, d'autres avancées sont attendues. La cartographie du génome revient à compléter un casse-tête complexe en utilisant toutes les données disponibles. Les informations cartographiques générées dans les laboratoires du monde entier sont saisies dans des bases de données centrales, telles que le National Center for Biotechnology Information (NCBI). Des efforts sont déployés pour rendre les informations plus facilement accessibles aux chercheurs et au grand public. Tout comme nous utilisons des systèmes de positionnement mondial au lieu de cartes papier pour naviguer sur les routes, le NCBI nous permet d'utiliser un outil de visualisation du génome pour simplifier le processus d'exploration de données.
CONCEPT EN ACTION

L'héritage mendélien en ligne chez l'homme (OMIM) est un catalogue en ligne consultable de gènes humains et de maladies génétiques. Ce site Web présente la cartographie du génome et détaille l'histoire et la recherche de chaque trait et trouble. Cliquez sur le lien pour rechercher des traits (tels que l'aptitude à la main) et des troubles génétiques (tels que le diabète).
Séquençage du génome
Bien que des progrès importants aient été réalisés dans les sciences médicales ces dernières années, les médecins sont toujours déconcertés par de nombreuses maladies et les chercheurs utilisent le séquençage du génome entier pour aller au fond du problème. Le séquençage du génome entier est un processus qui détermine la séquence d'ADN d'un génome entier. Le séquençage du génome entier est une approche par force brute qui permet de résoudre des problèmes lorsqu'une base génétique est au cœur d'une maladie. Plusieurs laboratoires fournissent désormais des services de séquençage, d'analyse et d'interprétation de génomes entiers.
En 2010, le séquençage complet du génome a été utilisé pour sauver un jeune garçon dont les intestins présentaient de multiples abcès mystérieux. L'enfant a subi plusieurs opérations du côlon sans aucun soulagement. Enfin, une séquence génomique complète a révélé un défaut dans une voie qui contrôle l'apoptose (mort cellulaire programmée). Une greffe de moelle osseuse a été utilisée pour surmonter cette maladie génétique, permettant ainsi de guérir le garçon. Il a été la première personne à être diagnostiquée avec succès par séquençage du génome entier.
Les premiers génomes à être séquencés, tels que ceux appartenant à des virus, à des bactéries et à des levures, étaient plus petits en termes de nombre de nucléotides que les génomes des organismes multicellulaires. Les génomes d'autres organismes modèles, tels que la souris (Mus musculus), la mouche des fruits (Drosophila melanogaster) et le nématode (Caenorhabditis elegans) sont maintenant connus. Une grande partie de la recherche fondamentale est menée sur les organismes modèles, car les informations peuvent être appliquées à d'autres organismes. Un organisme modèle est une espèce étudiée comme modèle afin de comprendre les processus biologiques d'autres espèces qui peuvent être représentées par l'organisme modèle. Par exemple, les mouches des fruits sont capables de métaboliser l'alcool comme les humains. Les gènes qui influent sur la sensibilité à l'alcool ont donc été étudiés chez les mouches des fruits afin de comprendre les variations de sensibilité à l'alcool chez les humains. Le séquençage de génomes entiers contribue aux efforts de recherche sur ces organismes modèles (Figure\(\PageIndex{2}\)).

La première séquence du génome humain a été publiée en 2003. Le nombre de génomes entiers séquencés augmente régulièrement et comprend désormais des centaines d'espèces et des milliers de génomes humains individuels.
Appliquer la génomique
L'introduction du séquençage de l'ADN et de projets de séquençage du génome entier, notamment le projet du génome humain, a élargi l'applicabilité des informations sur les séquences d'ADN. La génomique est maintenant utilisée dans une grande variété de domaines, tels que la métagénomique, la pharmacogénomique et la génomique mitochondriale. L'application la plus connue de la génomique consiste à comprendre et à trouver des remèdes à des maladies.
Prédire le risque de maladie au niveau individuel
La prévision du risque de maladie implique le dépistage et l'identification de personnes actuellement en bonne santé par analyse du génome au niveau individuel. Une intervention visant à modifier le mode de vie et à prendre des médicaments peut être recommandée avant l'apparition Cependant, cette approche est particulièrement applicable lorsque le problème provient d'une seule mutation génétique. Ces défauts ne représentent qu'environ 5 pour cent des maladies détectées dans les pays développés. La plupart des maladies courantes, telles que les maladies cardiaques, sont multifactorielles ou polygéniques, ce qui fait référence à une caractéristique phénotypique déterminée par deux gènes ou plus, ainsi que par des facteurs environnementaux tels que le régime alimentaire. En avril 2010, des scientifiques de l'université de Stanford ont publié l'analyse du génome d'un individu en bonne santé (Stephen Quake, un scientifique de l'université de Stanford, dont le génome a été séquencé) ; l'analyse a prédit sa propension à contracter diverses maladies. Une évaluation des risques a été réalisée pour analyser le pourcentage de risque de Quake pour 55 affections médicales différentes. Une mutation génétique rare a été découverte qui montrait qu'il courait un risque de crise cardiaque soudaine. On prévoyait également qu'il courrait un risque de 23 pour cent de développer un cancer de la prostate et un risque de 1,4 pour cent de développer la maladie d'Alzheimer. Les scientifiques ont utilisé des bases de données et plusieurs publications pour analyser les données génomiques. Même si le séquençage génomique devient de plus en plus abordable et que les outils d'analyse sont de plus en plus fiables, les questions éthiques liées à l'analyse génomique au niveau de la population restent à résoudre. Par exemple, ces données pourraient-elles être légitimement utilisées pour facturer plus ou moins les assurances ou pour affecter les notations de crédit ?
Études d'association pangénomiques
Depuis 2005, il est possible de mener un type d'étude appelé étude d'association pangénomique, ou GWAS. Un GWAS est une méthode qui permet d'identifier les différences entre les individus en ce qui concerne les polymorphismes mononucléotidiques (SNP) qui peuvent être impliqués dans l'apparition de maladies. La méthode est particulièrement adaptée aux maladies qui peuvent être affectées par un ou plusieurs changements génétiques dans l'ensemble du génome. Il est très difficile d'identifier les gènes impliqués dans une telle maladie à l'aide des informations sur les antécédents familiaux. La méthode GWAS s'appuie sur une base de données génétiques en cours de développement depuis 2002 appelée International HapMap Project. Le projet HapMap a séquencé les génomes de plusieurs centaines d'individus du monde entier et a identifié des groupes de SNP. Les groupes comprennent des SNP situés à proximité les uns des autres sur les chromosomes, de sorte qu'ils ont tendance à rester ensemble par recombinaison. Le fait que le groupe reste uni signifie qu'il suffit d'identifier un marqueur SNP pour identifier tous les SNP du groupe. Plusieurs millions de SNP ont été identifiés, mais il est beaucoup plus facile de les identifier chez d'autres individus dont le génome complet n'a pas été séquencé, car seuls les SNP marqueurs doivent être identifiés.
Dans un plan commun pour une GWAS, deux groupes de personnes sont choisis ; l'un est atteint de la maladie et l'autre non. Les individus de chaque groupe sont appariés selon d'autres caractéristiques afin de réduire l'effet des variables confusionnelles à l'origine de différences entre les deux groupes. Par exemple, les génotypes peuvent différer parce que les deux groupes proviennent pour la plupart de différentes régions du monde. Une fois que les individus sont choisis, et qu'ils sont généralement au nombre de mille ou plus pour que l'étude fonctionne, des échantillons de leur ADN sont prélevés. L'ADN est analysé à l'aide de systèmes automatisés afin d'identifier de grandes différences dans le pourcentage de SNP particuliers entre les deux groupes. Souvent, l'étude examine un million ou plus de SNP dans l'ADN. Les résultats de la GWAS peuvent être utilisés de deux manières : les différences génétiques peuvent être utilisées comme marqueurs de la prédisposition à la maladie chez des personnes non diagnostiquées, et les gènes spécifiques identifiés peuvent servir de cibles pour la recherche sur la voie moléculaire de la maladie et les thérapies potentielles. La découverte d'associations génétiques avec les maladies a eu pour conséquence la création de sociétés qui proposent ce que l'on appelle la « génomique personnelle », qui identifiera les niveaux de risque de diverses maladies en fonction du complément de SNP d'un individu. La science qui sous-tend ces services est controversée.
Comme la GWAS recherche des associations entre gènes et maladies, ces études fournissent des données pour d'autres recherches sur les causes, plutôt que de répondre elles-mêmes à des questions spécifiques. L'association entre une différence génétique et une maladie ne signifie pas nécessairement qu'il existe une relation de cause à effet. Cependant, certaines études ont fourni des informations utiles sur les causes génétiques des maladies. Par exemple, trois études différentes menées en 2005 ont identifié le gène d'une protéine impliquée dans la régulation de l'inflammation dans l'organisme qui est associée à une cécité causant une maladie appelée dégénérescence maculaire liée à l'âge. Cela a ouvert de nouvelles possibilités de recherche sur la cause de cette maladie. Un grand nombre de gènes associés à la maladie de Crohn ont été identifiés à l'aide de la GWAS, et certains d'entre eux ont suggéré de nouveaux mécanismes hypothétiques expliquant la cause de la maladie.
Pharmacogénomique
La pharmacogénomique consiste à évaluer l'efficacité et la sécurité des médicaments sur la base d'informations provenant de la séquence génomique d'un individu. Les informations sur les séquences génomiques personnelles peuvent être utilisées pour prescrire les médicaments les plus efficaces et les moins toxiques en fonction du génotype de chaque patient. L'étude des modifications de l'expression génique pourrait fournir des informations sur le profil de transcription des gènes en présence du médicament, qui peuvent être utilisées comme indicateur précoce du potentiel d'effets toxiques. Par exemple, les gènes impliqués dans la croissance cellulaire et la mort cellulaire contrôlée, lorsqu'ils sont perturbés, peuvent entraîner la croissance de cellules cancéreuses. Des études portant sur l'ensemble du génome peuvent également aider à découvrir de nouveaux gènes impliqués dans la toxicité des médicaments. Les signatures génétiques peuvent ne pas être complètement exactes, mais elles peuvent être testées plus avant l'apparition de symptômes pathologiques.
Métagénomique
Traditionnellement, la microbiologie a été enseignée dans l'idée qu'il est préférable d'étudier les microorganismes dans des conditions de culture pure, ce qui implique d'isoler un seul type de cellule et de le cultiver en laboratoire. Comme les microorganismes peuvent traverser plusieurs générations en quelques heures, leurs profils d'expression génétique s'adaptent très rapidement au nouvel environnement de laboratoire. D'autre part, de nombreuses espèces résistent à l'élevage isolé. La plupart des microorganismes ne vivent pas comme des entités isolées, mais dans des communautés microbiennes appelées biofilms. Pour toutes ces raisons, la culture pure n'est pas toujours le meilleur moyen d'étudier les microorganismes. La métagénomique est l'étude des génomes collectifs de plusieurs espèces qui croissent et interagissent dans une niche environnementale. La métagénomique peut être utilisée pour identifier de nouvelles espèces plus rapidement et pour analyser l'effet des polluants sur l'environnement (Figure\(\PageIndex{3}\)). Les techniques de métagénomique peuvent désormais également être appliquées à des communautés d'eucaryotes supérieurs, tels que les poissons.
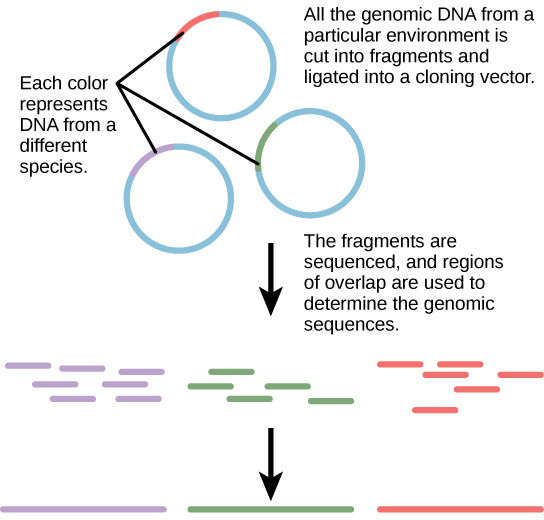
Création de nouveaux biocarburants
Les connaissances de la génomique des microorganismes sont utilisées pour trouver de meilleurs moyens d'exploiter les biocarburants issus des algues et des cyanobactéries. Les principales sources de combustible aujourd'hui sont le charbon, le pétrole, le bois et d'autres produits végétaux tels que l'éthanol. Bien que les plantes soient des ressources renouvelables, il est toujours nécessaire de trouver d'autres sources d'énergie renouvelables alternatives pour répondre aux besoins énergétiques de notre population. Le monde microbien est l'une des plus grandes ressources pour les gènes qui codent de nouvelles enzymes et produisent de nouveaux composés organiques, et il reste largement inexploité. Cette vaste ressource génétique est susceptible de fournir de nouvelles sources de biocarburants (Figure\(\PageIndex{4}\)).

Génomique mitochondriale
Les mitochondries sont des organites intracellulaires qui contiennent leur propre ADN. L'ADN mitochondrial mute rapidement et est souvent utilisé pour étudier les relations évolutives. Une autre caractéristique qui rend l'étude du génome mitochondrial intéressante est que, dans la plupart des organismes multicellulaires, l'ADN mitochondrial est transmis par la mère au cours du processus de fécondation. Pour cette raison, la génomique mitochondriale est souvent utilisée pour retracer la généalogie.
La génomique dans l'analyse judiciaire
Les informations et indices obtenus à partir d'échantillons d'ADN trouvés sur les lieux du crime ont été utilisés comme preuves dans des affaires judiciaires, et des marqueurs génétiques ont été utilisés dans des analyses médico-légales. L'analyse génomique est également devenue utile dans ce domaine. En 2001, la première utilisation de la génomique en criminalistique a été publiée. Il s'agissait d'un effort de collaboration entre des institutions de recherche universitaires et le FBI pour résoudre les mystérieux cas d'anthrax (Figure\(\PageIndex{5}\)) transportés par le service postal américain. La bactérie de l'anthrax a été transformée en poudre infectieuse et envoyée par la poste aux médias et à deux sénateurs américains. La poudre a infecté le personnel administratif et les postiers qui ouvraient ou manipulaient les lettres. Cinq personnes sont mortes et 17 sont tombées malades à cause de la bactérie. À l'aide de la génomique microbienne, les chercheurs ont déterminé qu'une souche spécifique d'anthrax était utilisée dans tous les envois ; la source a finalement été attribuée à un scientifique d'un laboratoire national de biodéfense du Maryland.
La génomique en agriculture
La génomique peut réduire dans une certaine mesure les essais et les échecs liés à la recherche scientifique, ce qui pourrait améliorer la qualité et la quantité des rendements agricoles (Figure\(\PageIndex{6}\)). Lier des caractères à des gènes ou à des signatures génétiques permet d'améliorer la sélection des cultures afin de générer des hybrides présentant les qualités les plus recherchées. Les scientifiques utilisent les données génomiques pour identifier les caractères souhaitables, puis les transfèrent à un autre organisme pour créer un nouvel organisme génétiquement modifié, comme décrit dans le module précédent. Les scientifiques découvrent comment la génomique peut améliorer la qualité et la quantité de la production agricole. Par exemple, les scientifiques pourraient utiliser des caractéristiques souhaitables pour créer un produit utile ou améliorer un produit existant, par exemple pour rendre une culture sensible à la sécheresse plus tolérante à la saison sèche.

Protéomique
Les protéines sont les produits finaux des gènes qui remplissent la fonction codée par le gène. Les protéines sont composées d'acides aminés et jouent un rôle important dans la cellule. Toutes les enzymes (sauf les ribozymes) sont des protéines et agissent comme des catalyseurs qui influent sur la vitesse des réactions. Les protéines sont également des molécules régulatrices et certaines sont des hormones. Les protéines de transport, telles que l'hémoglobine, aident à transporter l'oxygène vers divers organes. Les anticorps qui se défendent contre les particules étrangères sont également des protéines. À l'état malade, la fonction des protéines peut être altérée en raison de modifications au niveau génétique ou en raison d'un impact direct sur une protéine spécifique.
Un protéome est l'ensemble des protéines produites par un type de cellule. Les protéomes peuvent être étudiés à l'aide de la connaissance des génomes, car les gènes codent pour les ARNm et les ARNm codent pour les protéines. L'étude de la fonction des protéomes s'appelle la protéomique. La protéomique complète la génomique et est utile lorsque les scientifiques souhaitent tester leurs hypothèses basées sur des gènes. Même si toutes les cellules d'un organisme multicellulaire possèdent le même ensemble de gènes, l'ensemble des protéines produites dans les différents tissus est différent et dépend de l'expression des gènes. Ainsi, le génome est constant, mais le protéome varie et est dynamique au sein d'un organisme. De plus, les ARN peuvent être épissés alternativement (coupés-collés pour créer de nouvelles combinaisons et de nouvelles protéines), et de nombreuses protéines sont modifiées après traduction. Bien que le génome fournisse un schéma directeur, l'architecture finale dépend de plusieurs facteurs qui peuvent modifier la progression des événements qui génèrent le protéome.
Les génomes et les protéomes de patients atteints de maladies spécifiques sont étudiés afin de comprendre les bases génétiques de la maladie. La maladie la plus importante étudiée à l'aide d'approches protéomiques est le cancer (Figure\(\PageIndex{7}\)). Des approches protéomiques sont utilisées pour améliorer le dépistage et la détection précoce du cancer ; ceci est réalisé en identifiant les protéines dont l'expression est affectée par le processus pathologique. Une protéine individuelle est appelée biomarqueur, tandis qu'un ensemble de protéines dont les niveaux d'expression sont modifiés est appelé signature protéique. Pour qu'un biomarqueur ou une signature protéique soit utile en tant que candidat au dépistage et à la détection précoces d'un cancer, il doit être sécrété dans des fluides corporels tels que la sueur, le sang ou l'urine, afin que des dépistages à grande échelle puissent être réalisés de manière non invasive. Le problème actuel lié à l'utilisation de biomarqueurs pour la détection précoce du cancer est le taux élevé de résultats faussement négatifs. Un résultat faussement négatif est un résultat de test négatif qui aurait dû être positif. En d'autres termes, de nombreux cas de cancer ne sont pas détectés, ce qui rend les biomarqueurs peu fiables. Quelques exemples de biomarqueurs protéiques utilisés pour la détection du cancer sont le CA-125 pour le cancer de l'ovaire et le PSA pour le cancer de la prostate. Les signatures protéiques peuvent être plus fiables que les biomarqueurs pour détecter les cellules cancéreuses. La protéomique est également utilisée pour élaborer des plans de traitement individualisés, qui consistent à prédire si une personne répondra ou non à des médicaments spécifiques et aux effets secondaires que cette personne peut avoir. La protéomique est également utilisée pour prédire la possibilité d'une récurrence de la maladie.

Le National Cancer Institute a développé des programmes visant à améliorer la détection et le traitement du cancer. Les technologies protéomiques cliniques pour le cancer et le réseau de recherche sur la détection précoce visent à identifier les signatures protéiques spécifiques à différents types de cancers. Le programme de protéomique biomédicale est conçu pour identifier les signatures protéiques et concevoir des thérapies efficaces pour les patients atteints de cancer.
Résumé
La cartographie du génome revient à résoudre un grand casse-tête complexe avec des informations provenant de laboratoires du monde entier. Les cartes génétiques fournissent un aperçu de la localisation des gènes au sein d'un génome et elles estiment la distance entre les gènes et les marqueurs génétiques sur la base de la fréquence de recombinaison pendant la méiose. Les cartes physiques fournissent des informations détaillées sur la distance physique entre les gènes. Les informations les plus détaillées sont disponibles via le mappage de séquences. Les informations provenant de toutes les sources de cartographie et de séquençage sont combinées pour étudier un génome entier.
Le séquençage du génome entier est la dernière ressource disponible pour traiter les maladies génétiques. Certains médecins utilisent le séquençage complet du génome pour sauver des vies. La génomique a de nombreuses applications industrielles, notamment le développement de biocarburants, l'agriculture, les produits pharmaceutiques et le contrôle de la pollution.
L'imagination est le seul obstacle à l'applicabilité de la génomique. La génomique est appliquée à la plupart des domaines de la biologie ; elle peut être utilisée pour la médecine personnalisée, la prédiction des risques de maladie au niveau individuel, l'étude des interactions médicamenteuses avant la réalisation d'essais cliniques et l'étude des microorganismes présents dans l'environnement plutôt qu'en laboratoire. Il est également appliqué à la production de nouveaux biocarburants, à l'évaluation généalogique à l'aide de mitochondries, aux progrès de la criminalistique et à l'amélioration de l'agriculture.
La protéomique est l'étude de l'ensemble des protéines exprimées par un type de cellule donné dans certaines conditions environnementales. Dans un organisme multicellulaire, les différents types de cellules possèdent des protéomes différents, qui varient en fonction de l'évolution de l'environnement. Contrairement à un génome, un protéome est dynamique et en constante évolution, ce qui le rend plus complexe et plus utile que la seule connaissance des génomes.
Lexique
- biomarqueur
- une protéine individuelle produite uniquement à l'état malade
- carte génétique
- un aperçu des gènes et de leur localisation sur un chromosome basé sur les fréquences de recombinaison entre les marqueurs
- génomique
- l'étude de génomes entiers, y compris de l'ensemble complet des gènes, de leur séquence et de leur organisation nucléotidiques, ainsi que de leurs interactions au sein d'une espèce et avec d'autres espèces
- métagénomique
- l'étude des génomes collectifs de plusieurs espèces qui croissent et interagissent dans une niche environnementale
- organisme modèle
- espèce étudiée et utilisée comme modèle pour comprendre les processus biologiques chez d'autres espèces représentées par l'organisme modèle
- pharmacogénomique
- l'étude des interactions médicamenteuses avec le génome ou le protéome ; également appelée toxicogénomique
- carte physique
- une représentation de la distance physique entre des gènes ou des marqueurs génétiques
- signature protéique
- ensemble de protéines surexprimées ou sous-exprimées caractéristiques des cellules d'un tissu malade particulier
- protéomique
- étude de la fonction des protéomes
- séquençage du génome entier
- processus qui détermine la séquence nucléotidique d'un génome entier


