17.1 : Biotechnologie
- Page ID
- 189706
Compétences à développer
- Décrire l'électrophorèse sur gel
- Expliquer le clonage moléculaire et reproductif
- Décrire les utilisations de la biotechnologie en médecine et en agriculture
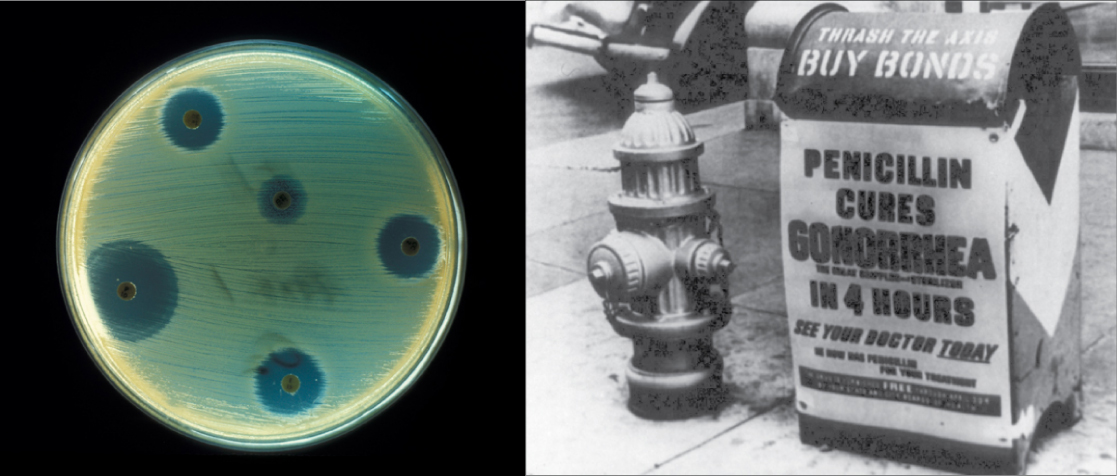
La biotechnologie est l'utilisation d'agents biologiques pour le progrès technologique. La biotechnologie a été utilisée pour l'élevage et les cultures bien avant que les bases scientifiques de ces techniques ne soient comprises. Depuis la découverte de la structure de l'ADN en 1953, le domaine de la biotechnologie s'est développé rapidement grâce à la recherche universitaire et aux entreprises privées. Les principales applications de cette technologie sont la médecine (production de vaccins et d'antibiotiques) et l'agriculture (modification génétique des cultures, par exemple pour augmenter les rendements). La biotechnologie a également de nombreuses applications industrielles, telles que la fermentation, le traitement des déversements d'hydrocarbures et la production de biocarburants (Figure\(\PageIndex{1}\)).
Techniques de base pour manipuler le matériel génétique (ADN et ARN)
Pour comprendre les techniques de base utilisées pour travailler avec les acides nucléiques, rappelez-vous que les acides nucléiques sont des macromolécules constituées de nucléotides (un sucre, un phosphate et une base azotée) liés par des liaisons phosphodiesters. Les groupes phosphates de ces molécules ont chacun une charge négative nette. Un ensemble complet de molécules d'ADN présentes dans le noyau s'appelle le génome. L'ADN possède deux brins complémentaires liés par des liaisons hydrogène entre les bases appariées. Les deux brins peuvent être séparés par exposition à des températures élevées (dénaturation de l'ADN) et peuvent être recuits par refroidissement. L'ADN peut être répliqué par l'enzyme ADN polymérase. Contrairement à l'ADN, qui se trouve dans le noyau des cellules eucaryotes, les molécules d'ARN quittent le noyau. Le type d'ARN le plus couramment analysé est l'ARN messager (ARNm), car il représente les gènes codant pour les protéines qui sont activement exprimés. Cependant, les molécules d'ARN présentent d'autres défis en matière d'analyse, car elles sont souvent moins stables que l'ADN.
Extraction d'ADN et d'ARN
Pour étudier ou manipuler les acides nucléiques, l'ADN ou l'ARN doit d'abord être isolé ou extrait des cellules. Différentes techniques sont utilisées pour extraire différents types d'ADN (Figure\(\PageIndex{2}\)). La plupart des techniques d'extraction d'acides nucléiques impliquent des étapes visant à ouvrir la cellule et à utiliser des réactions enzymatiques pour détruire toutes les macromolécules indésirables (telles que la dégradation des molécules indésirables et la séparation de l'échantillon d'ADN). Les cellules sont brisées à l'aide d'un tampon de lyse (une solution qui est principalement un détergent) ; la lyse signifie « se séparer ». Ces enzymes décomposent les molécules lipidiques des membranes cellulaires et des membranes nucléaires. Les macromolécules sont inactivées à l'aide d'enzymes telles que les protéases qui décomposent les protéines et les ribonucléases (RNases) qui décomposent l'ARN. L'ADN est ensuite précipité à l'aide d'alcool. L'ADN génomique humain est généralement visible sous la forme d'une masse gélatineuse blanche. Les échantillons d'ADN peuvent être conservés congelés à —80 °C pendant plusieurs années.
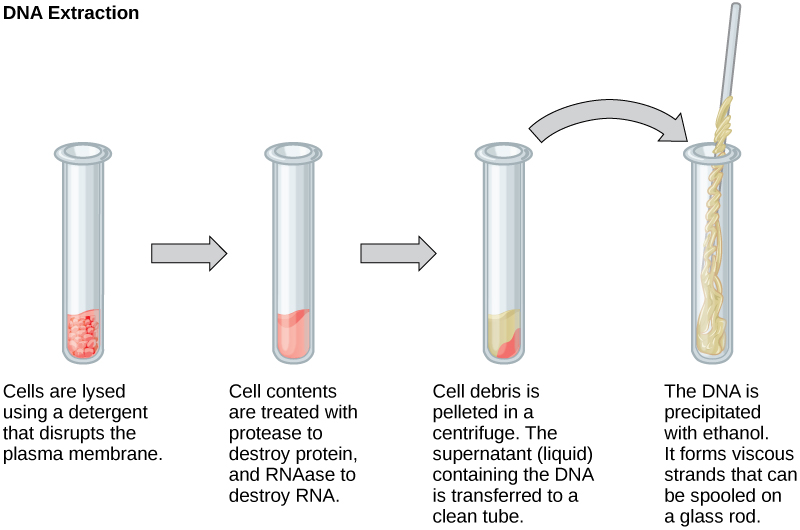
L'analyse de l'ARN est réalisée pour étudier les modèles d'expression génique dans les cellules. L'ARN est naturellement très instable car les RNases sont couramment présentes dans la nature et très difficiles à inactiver. À l'instar de l'ADN, l'extraction de l'ARN implique l'utilisation de divers tampons et enzymes pour inactiver les macromolécules et préserver l'ARN.
Électrophorèse sur gel
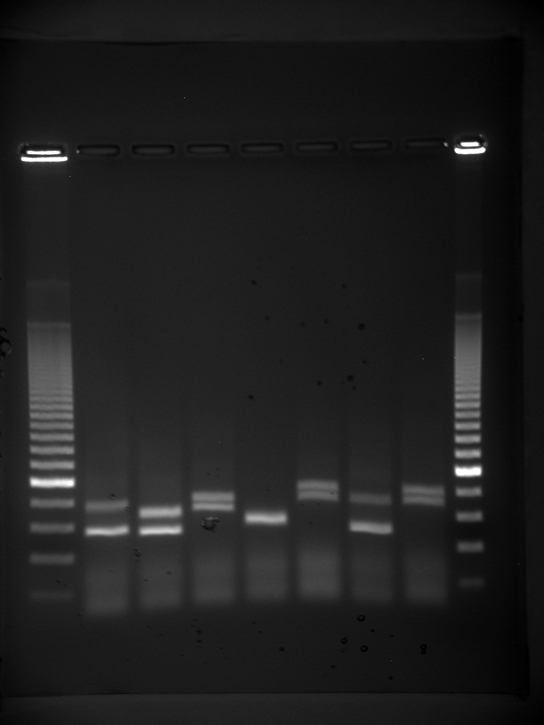
Comme les acides nucléiques sont des ions chargés négativement à un pH neutre ou basique dans un environnement aqueux, ils peuvent être mobilisés par un champ électrique. L'électrophorèse sur gel est une technique utilisée pour séparer les molécules en fonction de leur taille, en utilisant cette charge. Les acides nucléiques peuvent être séparés sous forme de chromosomes entiers ou de fragments. Les acides nucléiques sont chargés dans une fente située à proximité de l'électrode négative d'une matrice de gel poreuse semi-solide et tirés vers l'électrode positive située à l'extrémité opposée du gel. Les petites molécules se déplacent à travers les pores du gel plus rapidement que les molécules plus grosses ; cette différence de vitesse de migration sépare les fragments en fonction de leur taille. Il existe des échantillons standard de poids moléculaire qui peuvent être analysés en même temps que les molécules pour fournir une comparaison de taille. Les acides nucléiques d'une matrice de gel peuvent être observés à l'aide de divers colorants fluorescents ou colorés. Des fragments d'acides nucléiques distincts apparaissent sous forme de bandes à des distances spécifiques du sommet du gel (extrémité de l'électrode négative) en fonction de leur taille (Figure\(\PageIndex{3}\)). Un mélange de fragments d'ADN génomique de différentes tailles apparaît sous la forme d'un long frottis, alors que l'ADN génomique non découpé est généralement trop gros pour traverser le gel et forme une seule grande bande au sommet du gel.
Amplification de fragments d'acides nucléiques par réaction en chaîne par polymér
Bien que l'ADN génomique soit visible à l'œil nu lorsqu'il est extrait en vrac, l'analyse de l'ADN nécessite souvent de se concentrer sur une ou plusieurs régions spécifiques du génome. La réaction en chaîne par polymérase (PCR) est une technique utilisée pour amplifier des régions spécifiques de l'ADN en vue d'une analyse plus approfondie (Figure\(\PageIndex{4}\)). La PCR est utilisée à de nombreuses fins en laboratoire, telles que le clonage de fragments de gènes pour analyser des maladies génétiques, l'identification de l'ADN étranger contaminant dans un échantillon et l'amplification de l'ADN pour le séquençage. Des applications plus pratiques incluent la détermination de la paternité et la détection de maladies génétiques.
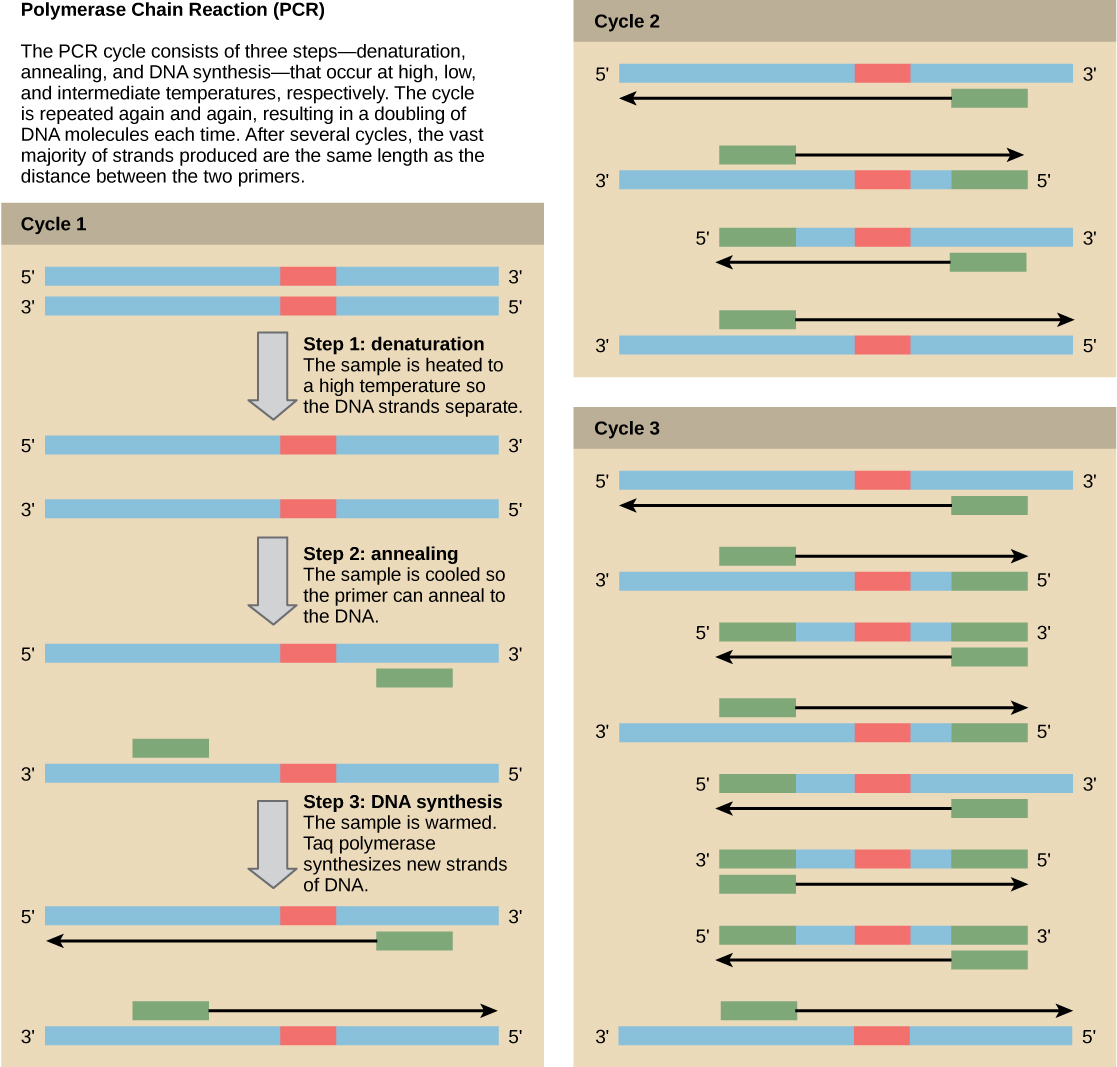
Les fragments d'ADN peuvent également être amplifiés à partir d'une matrice d'ARN dans le cadre d'un processus appelé PCR par transcriptase inverse (RT-PCR). La première étape consiste à recréer le brin modèle d'ADN original (appelé ADNc) en appliquant des nucléotides d'ADN à l'ARNm. Ce processus s'appelle la transcription inverse. Cela nécessite la présence d'une enzyme appelée transcriptase inverse. Une fois l'ADNc fabriqué, une PCR régulière peut être utilisée pour l'amplifier.
Lien vers l'apprentissage
Approfondissez votre compréhension de la réaction en chaîne par polymérase en suivant cet exercice interactif.
Hybridation, Southern Blotting et Northern Blotting
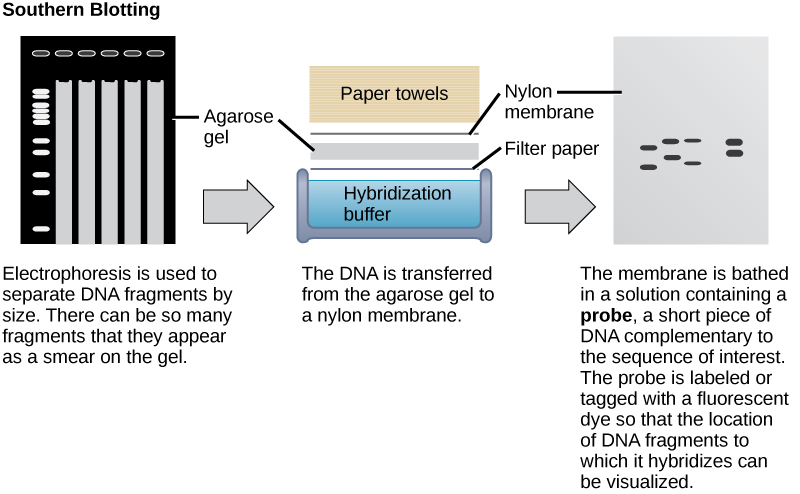
Les échantillons d'acides nucléiques, tels que l'ADN génomique fragmenté et les extraits d'ARN, peuvent être sondés pour détecter la présence de certaines séquences. De courts fragments d'ADN appelés sondes sont conçus et marqués avec des colorants radioactifs ou fluorescents pour faciliter la détection. L'électrophorèse sur gel sépare les fragments d'acides nucléiques en fonction de leur taille. Les fragments du gel sont ensuite transférés sur une membrane de nylon dans le cadre d'une procédure appelée « transfert » (Figure\(\PageIndex{5}\)). Les fragments d'acides nucléiques qui sont liés à la surface de la membrane peuvent ensuite être sondés avec des séquences de sonde spécifiques marquées par radioactivité ou par fluorescence. Lorsque l'ADN est transféré sur une membrane de nylon, la technique est appelée transfert Southern, et lorsque l'ARN est transféré sur une membrane de nylon, on parle de transfert nordique. Les Southern blots sont utilisés pour détecter la présence de certaines séquences d'ADN dans un génome donné, et les Northern Blot sont utilisés pour détecter l'expression génique.
Clonage moléculaire
En général, le mot « clonage » signifie la création d'une réplique parfaite ; toutefois, en biologie, la recréation d'un organisme entier est appelée « clonage reproductif ». Bien avant de tenter de cloner un organisme entier, les chercheurs ont appris à reproduire des régions ou des fragments souhaités du génome, un processus connu sous le nom de clonage moléculaire.
Le clonage de petits fragments du génome permet de manipuler et d'étudier des gènes spécifiques (et leurs produits protéiques) ou des régions non codantes de manière isolée. Un plasmide (également appelé vecteur) est une petite molécule d'ADN circulaire qui se réplique indépendamment de l'ADN chromosomique. Lors du clonage, les molécules plasmidiques peuvent être utilisées pour fournir un « dossier » dans lequel insérer le fragment d'ADN souhaité. Les plasmides sont généralement introduits dans un hôte bactérien à des fins de prolifération. Dans le contexte bactérien, le fragment d'ADN du génome humain (ou du génome d'un autre organisme étudié) est appelé ADN étranger, ou transgène, afin de le différencier de l'ADN de la bactérie, appelé ADN de l'hôte.
Les plasmides sont naturellement présents dans les populations bactériennes (comme Escherichia coli) et possèdent des gènes qui peuvent apporter des caractéristiques favorables à l'organisme, telles que la résistance aux antibiotiques (capacité de ne pas être affectés par les antibiotiques). Les plasmides ont été réutilisés et modifiés en tant que vecteurs pour le clonage moléculaire et la production à grande échelle de réactifs importants, tels que l'insuline et l'hormone de croissance humaine. Une caractéristique importante des vecteurs plasmidiques est la facilité avec laquelle un fragment d'ADN étranger peut être introduit via le site de clonage multiple (MCS). Le MCS est une courte séquence d'ADN contenant de multiples sites qui peuvent être coupés avec différentes endonucléases de restriction couramment disponibles. Les endonucléases de restriction reconnaissent des séquences d'ADN spécifiques et les coupent de manière prévisible ; elles sont produites naturellement par les bactéries comme mécanisme de défense contre l'ADN étranger. De nombreuses endonucléases de restriction effectuent des coupes échelonnées dans les deux brins d'ADN, de telle sorte que les extrémités coupées présentent un surplomb monocaténaire à 2 ou 4 bases. Comme ces surplombs peuvent être recuits avec des surplombs complémentaires, on les appelle « extrémités collantes ». L'ajout d'une enzyme appelée ADN ligase permet de joindre de façon permanente les fragments d'ADN via des liaisons phosphodiester. Ainsi, tout fragment d'ADN généré par le clivage de l'endonucléase de restriction peut être épissé entre les deux extrémités d'un ADN plasmidique découpé avec la même endonucléase de restriction (Figure\(\PageIndex{6}\)).
Molécules d'ADN recombinant
Les plasmides dans lesquels de l'ADN étranger est inséré sont appelés molécules d'ADN recombinant parce qu'ils sont créés artificiellement et ne se trouvent pas dans la nature. Elles sont également appelées molécules chimériques car l'origine de différentes parties des molécules peut être attribuée à différentes espèces d'organismes biologiques ou même à la synthèse chimique. Les protéines exprimées à partir de molécules d'ADN recombinant sont appelées protéines recombinantes. Tous les plasmides recombinants ne sont pas capables d'exprimer des gènes. L'ADN recombinant peut avoir besoin d'être déplacé vers un autre vecteur (ou hôte) mieux conçu pour l'expression génique. Les plasmides peuvent également être modifiés pour exprimer des protéines uniquement lorsqu'ils sont stimulés par certains facteurs environnementaux, afin que les scientifiques puissent contrôler l'expression des protéines recombinantes.
Art Connection
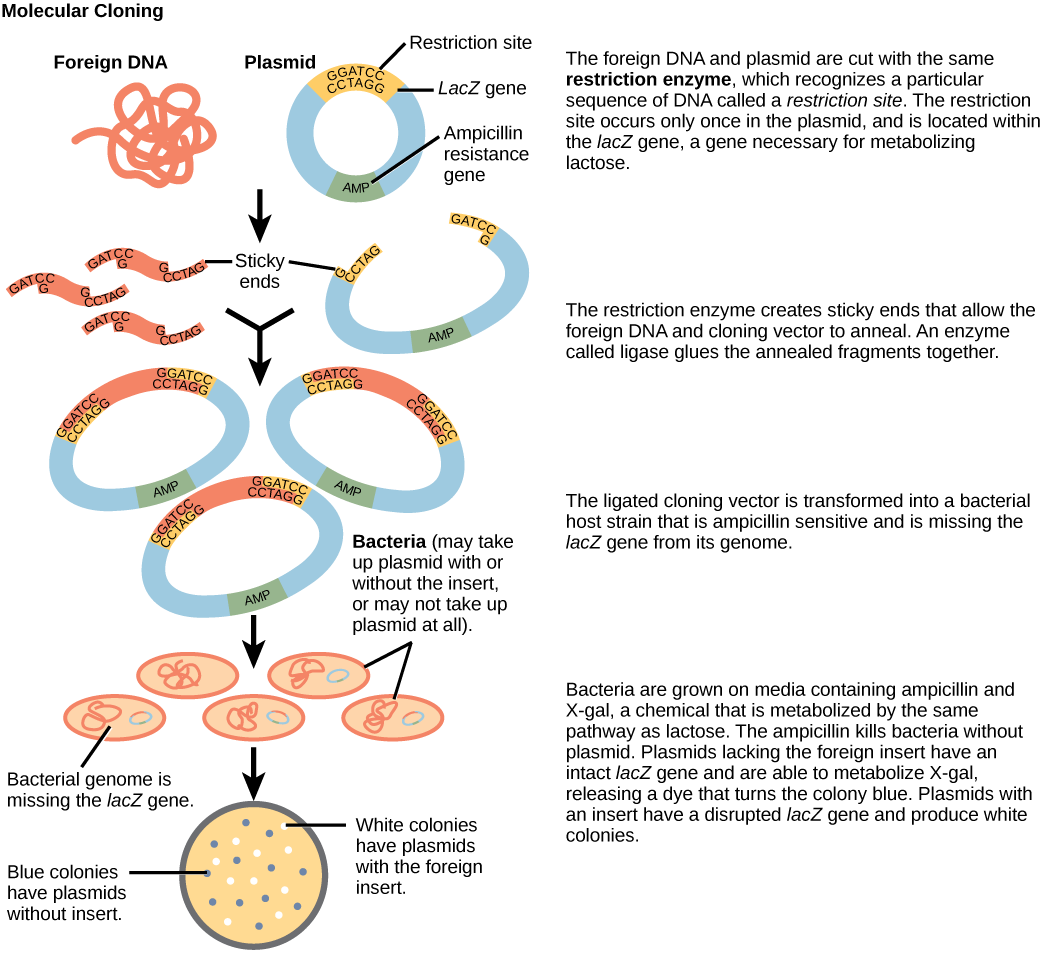
Vous travaillez dans un laboratoire de biologie moléculaire et, à votre insu, votre partenaire de laboratoire a laissé l'ADN génomique étranger que vous prévoyez de cloner sur le banc de laboratoire pendant la nuit au lieu de le conserver au congélateur. En conséquence, il a été dégradé par les nucléases, mais il a quand même été utilisé dans l'expérience. Le plasmide, en revanche, est bon. Quels résultats attendriez-vous de votre expérience de clonage moléculaire ?
- Il n'y aura pas de colonies sur la plaque bactérienne.
- Il n'y aura que des colonies bleues.
- Il y aura des colonies bleues et blanches.
- Ce ne seront que des colonies blanches.
Lien vers l'apprentissage
Visionnez une animation sur la recombinaison lors du clonage depuis le DNA Learning Center.
Clonage cellulaire
Les organismes unicellulaires, tels que les bactéries et les levures, produisent naturellement des clones d'eux-mêmes lorsqu'ils se répliquent de manière asexuée par fission binaire ; c'est ce que l'on appelle le clonage cellulaire. L'ADN nucléaire se duplique par le processus de mitose, ce qui crée une réplique exacte du matériel génétique.
Clonage reproductif
Le clonage reproductif est une méthode utilisée pour créer un clone ou une copie identique d'un organisme multicellulaire entier. La plupart des organismes multicellulaires se reproduisent par voie sexuée, ce qui implique l'hybridation génétique de deux individus (parents), rendant impossible la génération d'une copie identique ou d'un clone de l'un ou l'autre des parents. Les récents progrès de la biotechnologie ont permis d'induire artificiellement la reproduction asexuée de mammifères en laboratoire.
La parthénogenèse, ou « naissance vierge », se produit lorsqu'un embryon grandit et se développe sans que l'ovule ne soit fécondé ; il s'agit d'une forme de reproduction asexuée. Un exemple de parthénogenèse se produit chez les espèces chez lesquelles la femelle pond un œuf et, si l'ovule est fécondé, il s'agit d'un ovule diploïde et l'individu devient une femelle ; si l'ovule n'est pas fécondé, il reste un œuf haploïde et devient un mâle. L'œuf non fécondé est appelé œuf parthénogène, ou œuf vierge. Certains insectes et reptiles pondent des œufs parthénogènes qui peuvent devenir des adultes.
La reproduction sexuée nécessite deux cellules ; lorsque l'ovule haploïde et les spermatozoïdes fusionnent, il en résulte un zygote diploïde. Le noyau du zygote contient les informations génétiques nécessaires à la production d'un nouvel individu. Cependant, le développement embryonnaire précoce nécessite le matériel cytoplasmique contenu dans l'ovule. Cette idée est à la base du clonage reproductif. Par conséquent, si le noyau haploïde d'un ovule est remplacé par un noyau diploïde provenant de la cellule d'un individu de la même espèce (appelé donneur), il deviendra un zygote génétiquement identique au donneur. Le transfert de noyau de cellules somatiques est la technique qui consiste à transférer un noyau diploïde dans un ovule énucléé. Il peut être utilisé soit pour le clonage thérapeutique, soit pour le clonage reproductif.
Le premier animal cloné était Dolly, une brebis née en 1996. Le taux de réussite du clonage reproductif à l'époque était très faible. Dolly a vécu sept ans et est décédée des suites de complications respiratoires (Figure 17.1.7). Certains pensent que, comme l'ADN de la cellule appartient à un individu plus âgé, l'âge de l'ADN peut affecter l'espérance de vie d'un individu cloné. Depuis Dolly, plusieurs animaux tels que des chevaux, des taureaux et des chèvres ont été clonés avec succès, bien que ces individus présentent souvent des anomalies du visage, des membres et du cœur. Des tentatives ont été faites pour produire des embryons humains clonés en tant que sources de cellules souches embryonnaires, parfois appelées clonage à des fins thérapeutiques. Le clonage thérapeutique produit des cellules souches pour tenter de remédier à des maladies ou à des défauts nocifs (contrairement au clonage reproductif, qui vise à reproduire un organisme). Néanmoins, les efforts de clonage thérapeutique se sont heurtés à des résistances en raison de considérations bioéthiques.
Art Connection
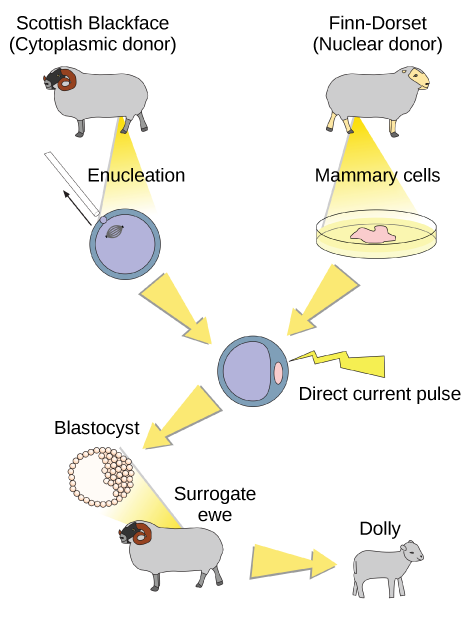
Pensez-vous que Dolly était un mouton Finn-Dorset ou un mouton Blackface écossais ?
Génie génétique
Le génie génétique est la modification du génotype d'un organisme à l'aide de la technologie de l'ADN recombinant pour modifier l'ADN d'un organisme afin d'obtenir les caractères souhaités. L'ajout d'ADN étranger sous forme de vecteurs d'ADN recombinant générés par clonage moléculaire est la méthode la plus courante de génie génétique. L'organisme qui reçoit l'ADN recombinant est appelé organisme génétiquement modifié (OGM). Si l'ADN étranger introduit provient d'une espèce différente, l'organisme hôte est dit transgénique. Les bactéries, les plantes et les animaux ont été génétiquement modifiés depuis le début des années 1970 à des fins universitaires, médicales, agricoles et industrielles. Aux États-Unis, les OGM tels que le soja Roundup Ready et le maïs résistant à la pyrale font partie de nombreux aliments transformés courants.
Ciblage génétique
Bien que les méthodes classiques d'étude de la fonction des gènes aient commencé par un phénotype donné et aient déterminé la base génétique de ce phénotype, les techniques modernes permettent aux chercheurs de commencer par le niveau de la séquence d'ADN et de se demander : « À quoi sert ce gène ou cet élément d'ADN ? » Cette technique, appelée génétique inverse, a permis d'inverser la méthodologie génétique classique. Cette méthode serait similaire à l'endommagement d'une partie du corps pour déterminer sa fonction. Un insecte qui perd une aile ne peut pas voler, ce qui signifie que la fonction de l'aile est de voler. La méthode génétique classique consiste à comparer des insectes incapables de voler à des insectes capables de voler et à observer que les insectes non volants ont perdu des ailes. De même, la mutation ou la suppression de gènes fournissent aux chercheurs des indices sur le fonctionnement des gènes. Les méthodes utilisées pour désactiver la fonction des gènes sont collectivement appelées ciblage génique. Le ciblage génique est l'utilisation de vecteurs d'ADN recombinant pour modifier l'expression d'un gène particulier, soit en introduisant des mutations dans un gène, soit en éliminant l'expression d'un certain gène en supprimant une partie ou la totalité de la séquence génique du génome d'un organisme.
Biotechnologie en médecine et en agriculture
Il est facile de voir comment la biotechnologie peut être utilisée à des fins médicales. La connaissance de la constitution génétique de notre espèce, de la base génétique des maladies héréditaires et de l'invention de technologies permettant de manipuler et de fixer des gènes mutants fournit des méthodes de traitement de la maladie. La biotechnologie en agriculture peut renforcer la résistance aux maladies, aux ravageurs et au stress environnemental, et améliorer à la fois le rendement et la qualité des cultures.
Diagnostic génétique et thérapie génique
Le processus de dépistage des anomalies génétiques présumées avant l'administration du traitement est appelé diagnostic génétique par tests génétiques. En fonction des modèles de transmission du gène responsable de la maladie, il est conseillé aux membres de la famille de se soumettre à des tests génétiques. Par exemple, il est généralement conseillé aux femmes ayant reçu un diagnostic de cancer du sein de subir une biopsie afin que l'équipe médicale puisse déterminer les bases génétiques du développement du cancer. Les plans de traitement sont basés sur les résultats de tests génétiques qui permettent de déterminer le type de cancer. Si le cancer est causé par des mutations génétiques héréditaires, il est également conseillé aux autres femmes de la famille de se soumettre à des tests génétiques et à un dépistage périodique du cancer du sein. Des tests génétiques sont également proposés pour les fœtus (ou les embryons fécondés in vitro) afin de déterminer la présence ou l'absence de gènes pathogènes dans les familles atteintes de maladies débilitantes spécifiques.
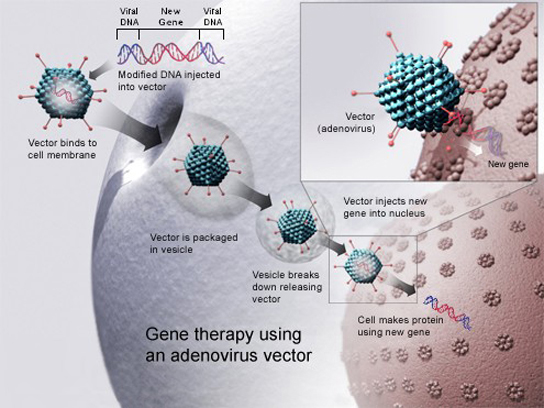
La thérapie génique est une technique de génie génétique utilisée pour soigner des maladies. Dans sa forme la plus simple, elle implique l'introduction d'un bon gène à un endroit aléatoire du génome pour aider à guérir une maladie causée par un gène muté. Le bon gène est généralement introduit dans les cellules malades en tant que partie d'un vecteur transmis par un virus qui peut infecter la cellule hôte et délivrer l'ADN étranger (Figure\(\PageIndex{8}\)). Des formes plus avancées de thérapie génique tentent de corriger la mutation au niveau du site d'origine du génome, comme c'est le cas pour le traitement de l'immunodéficience combinée sévère (SCID).
Production de vaccins, d'antibiotiques et d'hormones
Les stratégies de vaccination traditionnelles utilisent des formes affaiblies ou inactives de microorganismes pour augmenter la réponse immunitaire initiale. Les techniques modernes utilisent les gènes de microorganismes clonés dans des vecteurs pour produire en masse l'antigène souhaité. L'antigène est ensuite introduit dans l'organisme pour stimuler la réponse immunitaire primaire et déclencher la mémoire immunitaire. Des gènes clonés à partir du virus de la grippe ont été utilisés pour combattre les souches changeantes de ce virus.
Les antibiotiques sont des produits biotechnologiques. Elles sont produites naturellement par des microorganismes, tels que des champignons, afin d'obtenir un avantage sur les populations bactériennes. Les antibiotiques sont produits à grande échelle en cultivant et en manipulant des cellules fongiques.
La technologie de l'ADN recombinant a été utilisée pour produire de grandes quantités d'insuline humaine chez E. coli dès 1978. Auparavant, il n'était possible de traiter le diabète qu'avec de l'insuline porcine, qui provoquait des réactions allergiques chez l'homme en raison de différences dans le produit génique. De plus, l'hormone de croissance humaine (HGH) est utilisée pour traiter les troubles de croissance chez les enfants. Le gène HGH a été cloné à partir d'une banque d'ADNc et inséré dans des cellules d'E. coli par clonage dans un vecteur bactérien.
Animaux transgéniques
Bien que plusieurs protéines recombinantes utilisées en médecine soient produites avec succès par des bactéries, certaines protéines nécessitent un hôte animal eucaryote pour être traitées correctement. Pour cette raison, les gènes souhaités sont clonés et exprimés chez des animaux tels que des moutons, des chèvres, des poulets et des souris. Les animaux qui ont été modifiés pour exprimer l'ADN recombinant sont appelés animaux transgéniques. Plusieurs protéines humaines sont exprimées dans le lait de brebis et de chèvre transgéniques, et certaines sont exprimées dans les œufs de poulets. Les souris ont été largement utilisées pour exprimer et étudier les effets de gènes et de mutations recombinants.
Plantes transgéniques
La manipulation de l'ADN des plantes (c'est-à-dire la création d'OGM) a contribué à créer des caractéristiques souhaitables, telles que la résistance aux maladies, aux herbicides et aux pesticides, une meilleure valeur nutritive et une meilleure durée de conservation (Figure\(\PageIndex{9}\)). Les plantes sont la principale source de nourriture pour la population humaine. Les agriculteurs ont développé des méthodes de sélection des variétés végétales présentant des caractéristiques souhaitables bien avant que les pratiques biotechnologiques modernes ne soient mises en place.

Les plantes qui ont reçu de l'ADN recombinant d'autres espèces sont appelées plantes transgéniques. Comme ils ne sont pas naturels, les plantes transgéniques et autres OGM sont étroitement surveillés par les agences gouvernementales afin de s'assurer qu'ils sont propres à la consommation humaine et qu'ils ne mettent pas en danger d'autres formes de vie végétale et animale. Comme les gènes étrangers peuvent se propager à d'autres espèces présentes dans l'environnement, des tests approfondis sont nécessaires pour garantir la stabilité écologique. Les produits de base tels que le maïs, les pommes de terre et les tomates ont été les premières plantes cultivées à être génétiquement modifiées.
Transformation de plantes à l'aide d'Agrobacterium tumefaciens
Le transfert de gènes se produit naturellement entre les espèces des populations microbiennes. De nombreux virus responsables de maladies humaines, telles que le cancer, agissent en incorporant leur ADN dans le génome humain. Chez les plantes, les tumeurs causées par la bactérie Agrobacterium tumefaciens se produisent par transfert d'ADN de la bactérie à la plante. Bien que les tumeurs ne tuent pas les plantes, elles les rendent rabougries et les rendent plus vulnérables aux conditions environnementales difficiles. De nombreuses plantes, telles que les noix, les raisins, les arbres à noix et les betteraves, sont touchées par A. tumefaciens. L'introduction artificielle d'ADN dans les cellules végétales est plus difficile que dans les cellules animales en raison de l'épaisseur de la paroi cellulaire végétale.
Les chercheurs ont utilisé le transfert naturel de l'ADN d'Agrobacterium à une plante hôte pour introduire des fragments d'ADN de leur choix dans des plantes hôtes. Dans la nature, les A. tumefaciens responsables de la maladie possèdent un ensemble de plasmides, appelés plasmides Ti (plasmides induisant des tumeurs), qui contiennent des gènes permettant la production de tumeurs chez les plantes. L'ADN du plasmide Ti s'intègre dans le génome de la cellule végétale infectée. Les chercheurs manipulent les plasmides Ti pour éliminer les gènes responsables des tumeurs et insérer le fragment d'ADN souhaité pour le transfert dans le génome de la plante. Les plasmides Ti sont porteurs de gènes de résistance aux antibiotiques qui facilitent la sélection et peuvent également être propagés dans les cellules d'E. coli.
L'insecticide biologique Bacillus thuringiensis
Le Bacillus thuringiensis (Bt) est une bactérie qui produit des cristaux de protéines pendant la sporulation qui sont toxiques pour de nombreuses espèces d'insectes qui affectent les plantes. La toxine Bt doit être ingérée par les insectes pour qu'elle soit activée. Les insectes qui ont mangé de la toxine Bt cessent de se nourrir des plantes en quelques heures. Une fois la toxine activée dans les intestins des insectes, la mort survient en quelques jours. La biotechnologie moderne a permis aux plantes de coder leur propre toxine cristalline Bt qui agit contre les insectes. Les gènes de la toxine cristalline ont été clonés à partir du Bt et introduits dans des plantes. La toxine Bt s'est révélée sans danger pour l'environnement, non toxique pour les humains et les autres mammifères, et son utilisation par les agriculteurs biologiques est approuvée comme insecticide naturel.
Tomate Flavr Savr
La première culture génétiquement modifiée à être introduite sur le marché a été la tomate Flavr Savr produite en 1994. La technologie antisense ARN a été utilisée pour ralentir le processus de ramollissement et de pourriture causé par des infections fongiques, ce qui a entraîné une augmentation de la durée de conservation des tomates GM. Des modifications génétiques supplémentaires ont amélioré la saveur de cette tomate. La tomate Flavr Savr n'a pas réussi à se maintenir sur le marché en raison de problèmes de conservation et d'expédition de la récolte.
Résumé
Les acides nucléiques peuvent être isolés des cellules à des fins d'analyse plus approfondie en brisant les cellules et en détruisant enzymatiquement toutes les autres macromolécules majeures. Les chromosomes fragmentés ou entiers peuvent être séparés en fonction de leur taille par électrophorèse sur gel. De courts segments d'ADN ou d'ARN peuvent être amplifiés par PCR. Les blots Southern et Northern peuvent être utilisés pour détecter la présence de courtes séquences spécifiques dans un échantillon d'ADN ou d'ARN. Le terme « clonage » peut désigner le clonage de petits fragments d'ADN (clonage moléculaire), le clonage de populations cellulaires (clonage cellulaire) ou le clonage d'organismes entiers (clonage reproductif). Des tests génétiques sont effectués pour identifier les gènes responsables de maladies, et la thérapie génique est utilisée pour guérir une maladie héréditaire.
Les organismes transgéniques possèdent de l'ADN d'une espèce différente, généralement généré par des techniques de clonage moléculaire. Les vaccins, les antibiotiques et les hormones sont des exemples de produits obtenus par la technologie de l'ADN recombinant. Les plantes transgéniques sont généralement créées pour améliorer les caractéristiques des plantes cultivées.
Connexions artistiques
Figure\(\PageIndex{6}\) : Vous travaillez dans un laboratoire de biologie moléculaire et, à votre insu, votre partenaire de laboratoire a laissé l'ADN génomique étranger que vous prévoyez de cloner sur le banc pendant la nuit au lieu de le conserver au congélateur. En conséquence, il a été dégradé par les nucléases, mais il a quand même été utilisé dans l'expérience. Le plasmide, en revanche, est bon. Quels résultats attendriez-vous de votre expérience de clonage moléculaire ?
- Il n'y aura pas de colonies sur la plaque bactérienne.
- Il n'y aura que des colonies bleues.
- Il y aura des colonies bleues et blanches.
- Ce ne seront que des colonies blanches.
- Réponse
-
B. L'expérience ne produirait que des colonies bleues.
Figure\(\PageIndex{7}\) : Pensez-vous que Dolly était une brebis finlandaise du Dorset ou une brebis noire écossaise ?
- Réponse
-
Dolly était un mouton Finn-Dorset car même si la cellule d'origine provenait d'un mouton à tête noire écossais et que la mère porteuse était une tête noire écossaise, l'ADN provenait d'un Finn-Dorset.
Lexique
- résistance aux antibiotiques
- capacité d'un organisme à ne pas être affecté par les actions d'un antibiotique
- biotechnologie
- utilisation d'agents biologiques pour le progrès technologique
- clonage cellulaire
- production de populations cellulaires identiques par fission binaire
- clone
- réplique exacte
- ADN étranger
- ADN appartenant à une espèce différente ou ADN synthétisé artificiellement
- électrophorèse sur gel
- technique utilisée pour séparer les molécules en fonction de leur taille à l'aide d'une charge électrique
- ciblage génétique
- procédé pour modifier la séquence d'un gène spécifique en introduisant la version modifiée sur un vecteur
- thérapie génique
- technique utilisée pour soigner les maladies héréditaires en remplaçant les gènes mutants par de bons gènes
- diagnostic génétique
- diagnostic du potentiel de développement de la maladie en analysant les gènes à l'origine de la maladie
- génie génétique
- modification de la constitution génétique d'un organisme
- tests génétiques
- processus de dépistage de la présence de gènes responsables de maladies
- organisme génétiquement modifié (OGM)
- organisme dont le génome a été modifié artificiellement
- ADN de l'hôte
- ADN présent dans le génome de l'organisme d'intérêt
- tampon de lyse
- solution utilisée pour briser la membrane cellulaire et libérer le contenu cellulaire
- clonage moléculaire
- clonage de fragments d'ADN
- site de clonage multiple (MCS)
- site reconnaissable par de multiples endonucléases de restriction
- Northern Blotting
- transfert d'ARN d'un gel vers une membrane en nylon
- réaction en chaîne par polymérase (PCR)
- technique utilisée pour amplifier l'ADN
- sonde
- petit fragment d'ADN utilisé pour déterminer si la séquence complémentaire est présente dans un échantillon d'ADN
- protéase
- enzyme qui décompose les protéines
- ADN recombinant
- combinaison de fragments d'ADN générés par clonage moléculaire qui n'existe pas dans la nature ; également connue sous le nom de molécule chimérique
- protéine recombinante
- produit protéique d'un gène dérivé par clonage moléculaire
- clonage reproductif
- clonage d'organismes entiers
- endonucléase de restriction
- enzyme capable de reconnaître et de cliver des séquences d'ADN spécifiques
- génétique inversée
- méthode pour déterminer la fonction d'un gène en commençant par le gène lui-même au lieu de partir du produit génique
- PCR par transcriptase inverse (RT-PCR)
- Technique de PCR qui consiste à convertir l'ARN en ADN par transcriptase inverse
- ribonucléase
- enzyme qui décompose l'ARN
- Southern Blot
- transfert d'ADN d'un gel vers une membrane en nylon
- Plasmide Ti
- système plasmidique dérivé d'Agrobacterium tumifaciens utilisé par les scientifiques pour introduire de l'ADN étranger dans des cellules végétales
- transgénique
- organisme qui reçoit de l'ADN d'une espèce différente


