16.5 : Régulation des gènes post-transcriptionnels eucaryotes
- Page ID
- 189775
Compétences à développer
- Comprendre l'épissage de l'ARN et expliquer son rôle dans la régulation de l'expression génique
- Décrire l'importance de la stabilité de l'ARN dans la régulation génique
L'ARN est transcrit, mais doit être transformé en une forme mature avant que la traduction puisse commencer. Ce traitement après la transcription d'une molécule d'ARN, mais avant qu'elle ne soit traduite en protéine, est appelé modification post-transcriptionnelle. Comme pour les étapes épigénétiques et transcriptionnelles du traitement, cette étape post-transcriptionnelle peut également être régulée pour contrôler l'expression génique dans la cellule. Si l'ARN n'est pas traité, transporté ou traduit, aucune protéine ne sera synthétisée.
L'épissage de l'ARN, première étape du contrôle post-transcriptionnel
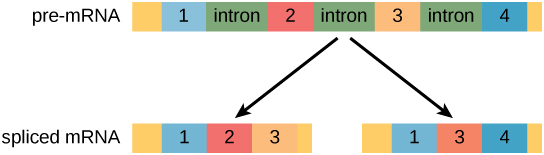
Dans les cellules eucaryotes, le transcrit d'ARN contient souvent des régions, appelées introns, qui sont éliminées avant la traduction. Les régions de l'ARN qui codent pour les protéines sont appelées exons (Figure\(\PageIndex{1}\)). Une fois qu'une molécule d'ARN a été transcrite, mais avant qu'elle ne quitte le noyau à traduire, l'ARN est traité et les introns sont retirés par épissage.
Evolution Connection : épissage alternatif de l'ARN
Dans les années 1970, des gènes présentant un épissage alternatif de l'ARN ont été observés pour la première fois. L'épissage alternatif de l'ARN est un mécanisme qui permet de produire différents produits protéiques à partir d'un gène lorsque différentes combinaisons d'introns, et parfois d'exons, sont retirées du transcrit (Figure\(\PageIndex{2}\)). Cet épissage alternatif peut être aléatoire, mais le plus souvent, il est contrôlé et agit comme un mécanisme de régulation génique, la fréquence des différentes alternatives d'épissage étant contrôlée par la cellule afin de contrôler la production de différents produits protéiques dans différentes cellules ou à différents stades de développement. L'épissage alternatif est maintenant considéré comme un mécanisme courant de régulation génique chez les eucaryotes ; selon une estimation, 70 % des gènes chez l'homme sont exprimés sous forme de protéines multiples par épissage alternatif.
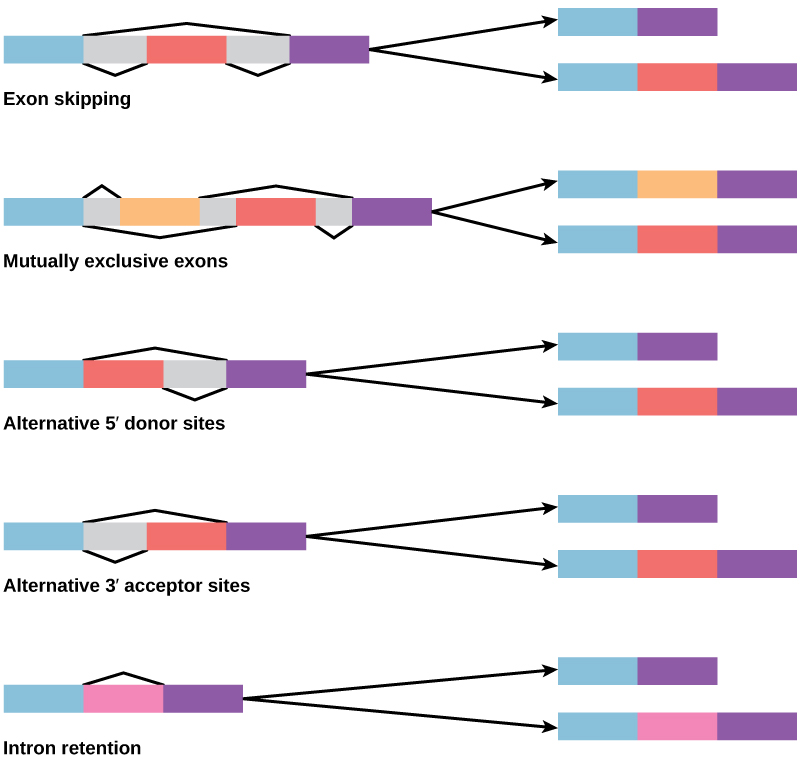
Comment l'épissage alternatif pourrait-il évoluer ? Les introns ont une séquence de reconnaissance de début et de fin ; il est facile d'imaginer l'échec du mécanisme d'épissage à identifier l'extrémité d'un intron et à trouver la fin de l'intron suivant, éliminant ainsi deux introns et l'exon intermédiaire. En fait, des mécanismes sont en place pour empêcher ce type de saut d'intron, mais des mutations sont susceptibles d'entraîner leur échec. De telles « erreurs » produiraient probablement une protéine non fonctionnelle. En effet, la cause de nombreuses maladies génétiques est l'épissage alternatif plutôt que les mutations d'une séquence. Cependant, un épissage alternatif créerait un variant protéique sans perte de la protéine d'origine, ouvrant ainsi des possibilités d'adaptation du nouveau variant à de nouvelles fonctions. La duplication de gènes a joué un rôle important dans l'évolution de nouvelles fonctions de la même manière en fournissant des gènes qui peuvent évoluer sans éliminer la protéine fonctionnelle d'origine.
Lien vers l'apprentissage
Visualisez comment se produit l'épissage de l'ARNm en regardant le processus en action dans cette vidéo. Animation du projet NDSU Virtual Cell Animations « ArNm Splicing ».
Contrôle de la stabilité de l'ARN
Avant que l'ARNm ne quitte le noyau, il reçoit deux « capuchons » protecteurs qui empêchent l'extrémité du brin de se dégrader au cours de son voyage. Le capuchon 5', qui est placé à l'extrémité 5' de l'ARNm, est généralement composé d'une molécule de guanosine triphosphate (GTP) méthylée. La queue en poly-A, qui est attachée à l'extrémité 3', est généralement composée d'une série de nucléotides adénines. Une fois que l'ARN est transporté vers le cytoplasme, la durée pendant laquelle l'ARN y réside peut être contrôlée. Chaque molécule d'ARN a une durée de vie définie et se désintègre à une vitesse spécifique. Ce taux de décomposition peut influencer la quantité de protéines présentes dans la cellule. Si le taux de désintégration augmente, l'ARN n'existera pas dans le cytoplasme aussi longtemps, ce qui raccourcira le temps de traduction. Inversement, si le taux de désintégration diminue, la molécule d'ARN restera plus longtemps dans le cytoplasme et une plus grande quantité de protéines pourra être traduite. Ce taux de désintégration est appelé stabilité de l'ARN. Si l'ARN est stable, il sera détecté pendant de plus longues périodes dans le cytoplasme.
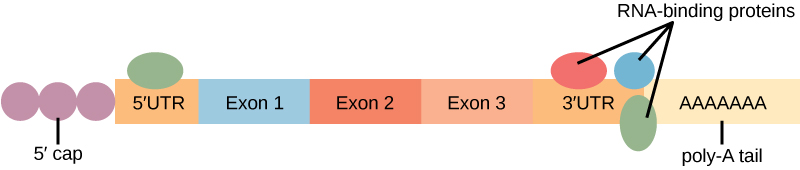
La liaison des protéines à l'ARN peut influencer sa stabilité. Les protéines, appelées protéines liant l'ARN, ou RBP, peuvent se lier aux régions de l'ARN situées juste en amont ou en aval de la région codant pour les protéines. Les régions de l'ARN qui ne sont pas traduites en protéines sont appelées régions non traduites, ou UTR. Ce ne sont pas des introns (ils ont été retirés dans le noyau). Il s'agit plutôt de régions qui régulent la localisation, la stabilité et la traduction des protéines de l'ARNm. La région située juste avant la région codant pour les protéines est appelée UTR 5', tandis que la région située après la région codante est appelée UTR 3' (Figure\(\PageIndex{3}\)). La liaison des RBP à ces régions peut augmenter ou diminuer la stabilité d'une molécule d'ARN, en fonction de la RBP spécifique qui se lie.
Stabilité de l'ARN et microARN
Outre les RBP qui se lient à l'ARN et contrôlent (augmentent ou diminuent) la stabilité de l'ARN, d'autres éléments appelés microARN peuvent se lier à la molécule d'ARN. Ces microARN, ou miARN, sont de courtes molécules d'ARN qui ne mesurent que 21 à 24 nucléotides. Les miRNA sont fabriqués dans le noyau sous forme de pré-miARN plus longs. Ces pré-miARN sont découpés en miARN matures par une protéine appelée découpage en dés. Comme les facteurs de transcription et les RBP, les miARN matures reconnaissent une séquence spécifique et se lient à l'ARN ; toutefois, les miARN s'associent également à un complexe ribonucléoprotéique appelé complexe de silençage induit par l'ARN (RISC). Le RISC se lie au miRNA pour dégrader l'ARNm cible. Ensemble, les miRNA et le complexe RISC détruisent rapidement la molécule d'ARN.
Résumé
Le contrôle post-transcriptionnel peut se produire à n'importe quel stade après la transcription, y compris lors de l'épissage de l'ARN, de la navette nucléaire et de la stabilité de l'ARN. Une fois que l'ARN est transcrit, il doit être traité pour créer un ARN mature prêt à être traduit. Cela implique l'élimination des introns qui ne codent pas pour les protéines. Les épissages se lient aux signaux qui marquent la frontière exon/intron pour éliminer les introns et ligaturer les exons ensemble. Une fois que cela se produit, l'ARN est mature et peut être traduit. L'ARN est créé et épissé dans le noyau, mais doit être transporté vers le cytoplasme pour être traduit. L'ARN est transporté vers le cytoplasme à travers le complexe de pores nucléaires. Une fois que l'ARN est dans le cytoplasme, la durée pendant laquelle il y réside avant d'être dégradé, appelée stabilité de l'ARN, peut également être modifiée pour contrôler la quantité globale de protéines synthétisées. La stabilité de l'ARN peut être augmentée, entraînant un temps de séjour plus long dans le cytoplasme, ou diminuée, ce qui réduit le temps et réduit la synthèse des protéines. La stabilité de l'ARN est contrôlée par les protéines liant l'ARN (RPB) et les microARN (miARN). Ces RPB et microARN se lient à l'UTR 5' ou à l'UTR 3' de l'ARN pour augmenter ou diminuer la stabilité de l'ARN. En fonction de la pression artérielle, la stabilité peut être augmentée ou diminuée de manière significative ; cependant, les miRNA diminuent toujours la stabilité et favorisent la décomposition.
Lexique
- 3 PIEDS D'URNE
- Région 3' non traduite ; région située juste en aval de la région codant pour les protéines dans une molécule d'ARN qui n'est pas traduite
- Casquette de 5 pieds
- une molécule de guanosine triphosphate (GTP) méthylée fixée à l'extrémité 5' d'un ARN messager pour protéger cette extrémité de la dégradation
- BUR DE 5 PIEDS
- Région 5' non traduite ; région située juste en amont de la région codant pour les protéines dans une molécule d'ARN qui n'est pas traduite
- découpeur
- enzyme qui découpe le pré-miARN pour en faire la forme mature du miARN
- MicroARN (miARN)
- petites molécules d'ARN (d'une longueur d'environ 21 nucléotides) qui se lient aux molécules d'ARN pour les dégrader
- Queue en poly-A
- une série de nucléotides d'adénine qui sont attachés à l'extrémité 3' d'un mRNA pour protéger l'extrémité de la dégradation
- Protéine liant l'ARN (RBP)
- protéine qui se lie à l'UTR 3' ou 5' pour augmenter ou diminuer la stabilité de l'ARN
- stabilité de l'ARN
- combien de temps une molécule d'ARN restera intacte dans le cytoplasme
- région non traduite
- segment de la molécule d'ARN qui n'est pas traduit en protéine. Ces régions se situent avant (en amont ou 5') et après (en aval ou 3') la région codant pour les protéines
- RISC
- complexe protéique qui se lie avec le miARN à l'ARN pour le dégrader


