15.5 : Ribosomes et synthèse des protéines
- Page ID
- 189826
Compétences à développer
- Décrire les différentes étapes de la synthèse des protéines
- Discuter du rôle des ribosomes dans la synthèse des protéines
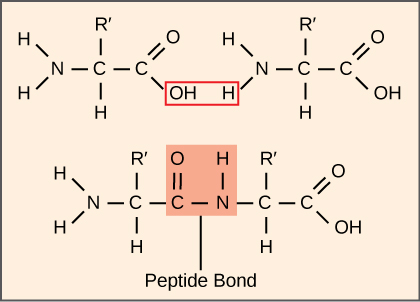
La synthèse des protéines consomme plus d'énergie cellulaire que tout autre processus métabolique. À leur tour, les protéines représentent plus de masse que tout autre composant des organismes vivants (à l'exception de l'eau), et les protéines remplissent pratiquement toutes les fonctions d'une cellule. Le processus de traduction, ou synthèse des protéines, implique le décodage d'un message d'ARNm en un produit polypeptidique. Les acides aminés sont liés entre eux de manière covalente en liant des liaisons peptidiques sur des longueurs allant d'environ 50 résidus d'acides aminés à plus de 1 000. Chaque acide aminé individuel possède un groupe amino (NH 2) et un groupe carboxyle (COOH). Les polypeptides se forment lorsque le groupe amino d'un acide aminé forme une liaison amide (c'est-à-dire un peptide) avec le groupe carboxyle d'un autre acide aminé (Figure\(\PageIndex{1}\)). Cette réaction est catalysée par les ribosomes et génère une molécule d'eau.
La machine de synthèse des protéines
Outre le modèle d'ARNm, de nombreuses molécules et macromolécules contribuent au processus de traduction. La composition de chaque composant peut varier d'une espèce à l'autre ; par exemple, les ribosomes peuvent contenir un nombre différent d'ARNr et de polypeptides selon l'organisme. Cependant, les structures générales et les fonctions du mécanisme de synthèse des protéines sont comparables des bactéries aux cellules humaines. La traduction nécessite la saisie d'un modèle d'ARNm, de ribosomes, d'ARNt et de divers facteurs enzymatiques.
Lien vers l'apprentissage

Cliquez sur les étapes de cette page interactive PBS pour voir la synthèse des protéines en action.
Ribosomes
Avant même qu'un ARNm ne soit traduit, une cellule doit investir de l'énergie pour construire chacun de ses ribosomes. Chez E. coli, entre 10 000 et 70 000 ribosomes sont présents dans chaque cellule à un moment donné. Un ribosome est une macromolécule complexe composée d'ARNr structuraux et catalytiques et de nombreux polypeptides distincts. Chez les eucaryotes, le nucléole est complètement spécialisé dans la synthèse et l'assemblage des ARNr.
Les ribosomes existent dans le cytoplasme des procaryotes et dans le cytoplasme et le réticulum endoplasmique rugueux chez les eucaryotes. Les mitochondries et les chloroplastes possèdent également leurs propres ribosomes dans la matrice et le stroma, qui ressemblent davantage aux ribosomes procaryotes (et présentent une sensibilité aux médicaments similaire) que les ribosomes situés juste à l'extérieur de leurs membranes externes dans le cytoplasme. Les ribosomes se dissocient en grandes et petites sous-unités lorsqu'ils ne synthétisent pas de protéines et se réassocient au début de la traduction. Chez E. coli, la petite sous-unité est décrite comme 30S et la grande sous-unité est 50S, pour un total de 70S (rappelons que les unités de Svedberg ne sont pas additives). Les ribosomes de mammifères ont une petite sous-unité 40S et une grande sous-unité 60S, pour un total de 80S. La petite sous-unité est responsable de la liaison à la matrice d'ARNm, tandis que la grande sous-unité se lie séquentiellement aux ARNt. Chaque molécule d'ARNm est traduite simultanément par de nombreux ribosomes, tous synthétisant des protéines dans la même direction : lecture de l'ARNm de 5' à 3' et synthèse du polypeptide de l'extrémité N à l'extrémité C. La structure complète de l'ARNM/du poly-ribosome est appelée polysome.
ARNt
Les ARNt sont des molécules d'ARN structurales qui ont été transcrites à partir de gènes par l'ARN polymérase III. Selon les espèces, 40 à 60 types d'ARNt existent dans le cytoplasme. En tant qu'adaptateurs, des ARNt spécifiques se lient aux séquences de la matrice d'ARNm et ajoutent l'acide aminé correspondant à la chaîne polypeptidique. Par conséquent, les ARNt sont les molécules qui « traduisent » réellement le langage de l'ARN dans le langage des protéines.
Parmi les 64 codons d'ARNm possibles, ou combinaisons triplet de A, U, G et C, trois indiquent la fin de la synthèse des protéines et 61 indiquent l'ajout d'acides aminés à la chaîne polypeptidique. Parmi ces 61, un codon (AUG) code également le début de la traduction. Chaque anticodon d'ARNt peut s'apparier à l'un des codons d'ARNm et ajouter un acide aminé ou terminer la traduction, selon le code génétique. Par exemple, si la séquence CUA apparaissait sur un modèle d'ARNm dans le cadre de lecture approprié, elle lierait un ARNt exprimant la séquence complémentaire, GAU, qui serait liée à l'acide aminé leucine.
En tant que molécules adaptatrices de la traduction, il est surprenant que les ARNt puissent intégrer autant de spécificité dans un boîtier aussi petit. Considérez que les ARNt doivent interagir avec trois facteurs : 1) ils doivent être reconnus par la bonne aminoacyl synthétase (voir ci-dessous) ; 2) ils doivent être reconnus par les ribosomes ; et 3) ils doivent se lier à la séquence correcte de l'ARNm.
Synthétases d'aminoacyl tRNA
Le processus de pré-synthèse de l'ARNt par l'ARN polymérase III crée uniquement la partie ARN de la molécule adaptatrice. L'acide aminé correspondant doit être ajouté ultérieurement, une fois que l'ARNt est traité et exporté vers le cytoplasme. Grâce au processus de « chargement » de l'ARNt, chaque molécule d'ARNt est liée à son acide aminé approprié par un groupe d'enzymes appelées aminoacyl tRNA synthétases. Au moins un type d'aminoacyl tRNA synthétase existe pour chacun des 20 acides aminés ; le nombre exact d'aminoacyl tRNA synthétases varie selon les espèces. Ces enzymes se lient et hydrolysent d'abord l'ATP pour catalyser une liaison à haute énergie entre un acide aminé et l'adénosine monophosphate (AMP) ; une molécule de pyrophosphate est expulsée lors de cette réaction. L'acide aminé activé est ensuite transféré vers l'ARNt, et l'AMP est libéré.
Le mécanisme de synthèse des protéines
Comme pour la synthèse des ARNm, la synthèse des protéines peut être divisée en trois phases : initiation, élongation et terminaison. Le processus de traduction est similaire chez les procaryotes et les eucaryotes. Nous allons explorer ici comment se produit la traduction chez E. coli, un procaryote représentatif, et préciser les différences entre la traduction procaryote et la traduction eucaryote.
Initiation de la traduction
La synthèse des protéines commence par la formation d'un complexe initiatique. Chez E. coli, ce complexe implique le petit ribosome 30S, la matrice d'ARNm, trois facteurs d'initiation (IF-1, IF-2 et IF-3) et un ARNt initiateur spécial, appelé\(\text{tRNA}_\text{f}^\text{Met}\). The initiator tRNA interacts with the start codon AUG (or rarely, GUG), links to a formylated methionine called fMet, and can also bind IF-2. Formylated methionine is inserted by \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) at the beginning of every polypeptide chain synthesized by E. coli, but it is usually clipped off after translation is complete. When an in-frame AUG is encountered during translation elongation, a non-formylated methionine is inserted by a regular Met-tRNAMet.
In E. coli mRNA, a sequence upstream of the first AUG codon, called the Shine-Dalgarno sequence (AGGAGG), interacts with the rRNA molecules that compose the ribosome. This interaction anchors the 30S ribosomal subunit at the correct location on the mRNA template. Guanosine triphosphate (GTP), which is a purine nucleotide triphosphate, acts as an energy source during translation—both at the start of elongation and during the ribosome’s translocation.
In eukaryotes, a similar initiation complex forms, comprising mRNA, the 40S small ribosomal subunit, IFs, and nucleoside triphosphates (GTP and ATP). The charged initiator tRNA, called Met-tRNAi, does not bind fMet in eukaryotes, but is distinct from other Met-tRNAs in that it can bind IFs.
Instead of depositing at the Shine-Dalgarno sequence, the eukaryotic initiation complex recognizes the 7-methylguanosine cap at the 5' end of the mRNA. A cap-binding protein (CBP) and several other IFs assist the movement of the ribosome to the 5' cap. Once at the cap, the initiation complex tracks along the mRNA in the 5' to 3' direction, searching for the AUG start codon. Many eukaryotic mRNAs are translated from the first AUG, but this is not always the case. According to Kozak’s rules, the nucleotides around the AUG indicate whether it is the correct start codon. Kozak’s rules state that the following consensus sequence must appear around the AUG of vertebrate genes: 5'-gccRccAUGG-3'. The R (for purine) indicates a site that can be either A or G, but cannot be C or U. Essentially, the closer the sequence is to this consensus, the higher the efficiency of translation.
Once the appropriate AUG is identified, the other proteins and CBP dissociate, and the 60S subunit binds to the complex of Met-tRNAi, mRNA, and the 40S subunit. This step completes the initiation of translation in eukaryotes.
Translation, Elongation, and Termination
In prokaryotes and eukaryotes, the basics of elongation are the same, so we will review elongation from the perspective of E. coli. The 50S ribosomal subunit of E. coli consists of three compartments: the A (aminoacyl) site binds incoming charged aminoacyl tRNAs. The P (peptidyl) site binds charged tRNAs carrying amino acids that have formed peptide bonds with the growing polypeptide chain but have not yet dissociated from their corresponding tRNA. The E (exit) site releases dissociated tRNAs so that they can be recharged with free amino acids. There is one exception to this assembly line of tRNAs: in E. coli, \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) is capable of entering the P site directly without first entering the A site. Similarly, the eukaryotic Met-tRNAi, with help from other proteins of the initiation complex, binds directly to the P site. In both cases, this creates an initiation complex with a free A site ready to accept the tRNA corresponding to the first codon after the AUG.
During translation elongation, the mRNA template provides specificity. As the ribosome moves along the mRNA, each mRNA codon comes into register, and specific binding with the corresponding charged tRNA anticodon is ensured. If mRNA were not present in the elongation complex, the ribosome would bind tRNAs nonspecifically.
Elongation proceeds with charged tRNAs entering the A site and then shifting to the P site followed by the E site with each single-codon “step” of the ribosome. Ribosomal steps are induced by conformational changes that advance the ribosome by three bases in the 3' direction. The energy for each step of the ribosome is donated by an elongation factor that hydrolyzes GTP. Peptide bonds form between the amino group of the amino acid attached to the A-site tRNA and the carboxyl group of the amino acid attached to the P-site tRNA. The formation of each peptide bond is catalyzed by peptidyl transferase, an RNA-based enzyme that is integrated into the 50S ribosomal subunit. The energy for each peptide bond formation is derived from GTP hydrolysis, which is catalyzed by a separate elongation factor. The amino acid bound to the P-site tRNA is also linked to the growing polypeptide chain. As the ribosome steps across the mRNA, the former P-site tRNA enters the E site, detaches from the amino acid, and is expelled (Figure \(\PageIndex{2}\)). Amazingly, the E. coli translation apparatus takes only 0.05 seconds to add each amino acid, meaning that a 200-amino acid protein can be translated in just 10 seconds.
Art Connection
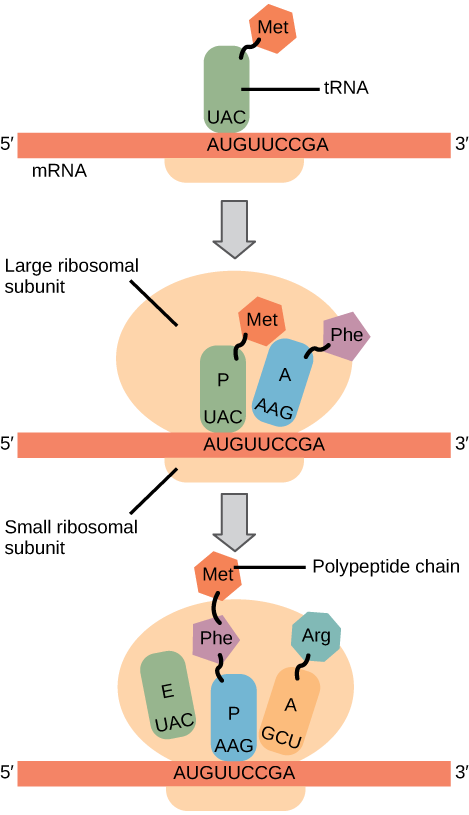
Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Termination of translation occurs when a nonsense codon (UAA, UAG, or UGA) is encountered. Upon aligning with the A site, these nonsense codons are recognized by release factors in prokaryotes and eukaryotes that instruct peptidyl transferase to add a water molecule to the carboxyl end of the P-site amino acid. This reaction forces the P-site amino acid to detach from its tRNA, and the newly made protein is released. The small and large ribosomal subunits dissociate from the mRNA and from each other; they are recruited almost immediately into another translation initiation complex. After many ribosomes have completed translation, the mRNA is degraded so the nucleotides can be reused in another transcription reaction.
Protein Folding, Modification, and Targeting
During and after translation, individual amino acids may be chemically modified, signal sequences may be appended, and the new protein “folds” into a distinct three-dimensional structure as a result of intramolecular interactions. A signal sequence is a short tail of amino acids that directs a protein to a specific cellular compartment. These sequences at the amino end or the carboxyl end of the protein can be thought of as the protein’s “train ticket” to its ultimate destination. Other cellular factors recognize each signal sequence and help transport the protein from the cytoplasm to its correct compartment. For instance, a specific sequence at the amino terminus will direct a protein to the mitochondria or chloroplasts (in plants). Once the protein reaches its cellular destination, the signal sequence is usually clipped off.
Many proteins fold spontaneously, but some proteins require helper molecules, called chaperones, to prevent them from aggregating during the complicated process of folding. Even if a protein is properly specified by its corresponding mRNA, it could take on a completely dysfunctional shape if abnormal temperature or pH conditions prevent it from folding correctly.
Summary
The players in translation include the mRNA template, ribosomes, tRNAs, and various enzymatic factors. The small ribosomal subunit forms on the mRNA template either at the Shine-Dalgarno sequence (prokaryotes) or the 5' cap (eukaryotes). Translation begins at the initiating AUG on the mRNA, specifying methionine. The formation of peptide bonds occurs between sequential amino acids specified by the mRNA template according to the genetic code. Charged tRNAs enter the ribosomal A site, and their amino acid bonds with the amino acid at the P site. The entire mRNA is translated in three-nucleotide “steps” of the ribosome. When a nonsense codon is encountered, a release factor binds and dissociates the components and frees the new protein. Folding of the protein occurs during and after translation.
Art Connections
Figure \(\PageIndex{2}\): Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
- Answer
-
Tetracycline: a; Chloramphenicol: c.
Glossary
- aminoacyl tRNA synthetase
- enzyme that “charges” tRNA molecules by catalyzing a bond between the tRNA and a corresponding amino acid
- initiator tRNA
- in prokaryotes, called \(\text{tRNA}_\text{f}^\text{Met}\); in eukaryotes, called tRNAi; a tRNA that interacts with a start codon, binds directly to the ribosome P site, and links to a special methionine to begin a polypeptide chain
- Kozak’s rules
- determines the correct initiation AUG in a eukaryotic mRNA; the following consensus sequence must appear around the AUG: 5’-GCC(purine)CCAUGG-3’; the bolded bases are most important
- peptidyl transferase
- RNA-based enzyme that is integrated into the 50S ribosomal subunit and catalyzes the formation of peptide bonds
- polysome
- mRNA molecule simultaneously being translated by many ribosomes all going in the same direction
- Shine-Dalgarno sequence
- (AGGAGG); initiates prokaryotic translation by interacting with rRNA molecules comprising the 30S ribosome
- signal sequence
- short tail of amino acids that directs a protein to a specific cellular compartment
- start codon
- AUG (or rarely, GUG) on an mRNA from which translation begins; always specifies methionine


