14.4 : Réplication de l'ADN chez les procaryotes
- Page ID
- 189761
Compétences à développer
- Expliquer le processus de réplication de l'ADN chez les procaryotes
- Discutez du rôle des différentes enzymes et protéines dans le soutien de ce processus
La réplication de l'ADN a été extrêmement bien étudiée chez les procaryotes, principalement en raison de la petite taille du génome et des mutants disponibles. E. coli possède 4,6 millions de paires de bases dans un seul chromosome circulaire et toutes se répliquent en 42 minutes environ, en commençant par une seule origine de réplication et en faisant le tour du cercle dans les deux sens. Cela signifie qu'environ 1000 nucléotides sont ajoutés par seconde. Le processus est assez rapide et se déroule sans trop d'erreurs.
La réplication de l'ADN fait appel à un grand nombre de protéines et d'enzymes, chacune jouant un rôle essentiel au cours du processus. L'un des principaux acteurs est l'enzyme ADN polymérase, également connue sous le nom d'ADN pol, qui ajoute des nucléotides un par un à la chaîne d'ADN en croissance qui sont complémentaires au brin matrice. L'ajout de nucléotides nécessite de l'énergie ; cette énergie est obtenue à partir des nucléotides auxquels sont attachés trois phosphates, comme l'ATP auquel sont attachés trois groupes phosphates. Lorsque la liaison entre les phosphates est rompue, l'énergie libérée est utilisée pour former la liaison phosphodiester entre le nucléotide entrant et la chaîne en croissance. Chez les procaryotes, trois principaux types de polymérases sont connus : l'ADN pol I, l'ADN pol II et l'ADN pol III. On sait maintenant que l'ADN pol III est l'enzyme nécessaire à la synthèse de l'ADN ; l'ADN pol I et l'ADN pol II sont principalement nécessaires à la réparation.
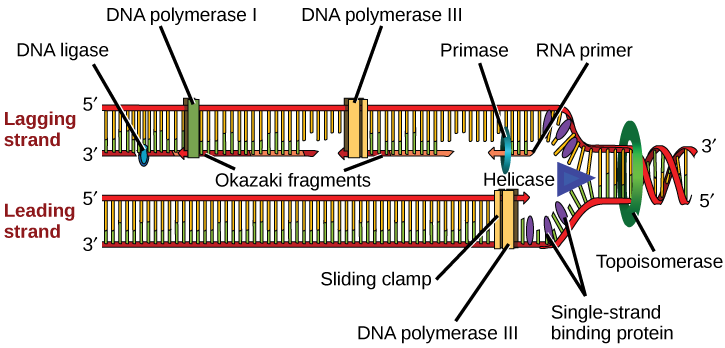
Comment la machine de réplication sait-elle par où commencer ? Il s'avère qu'il existe des séquences nucléotidiques spécifiques appelées origines de réplication où commence la réplication. Chez E. coli, qui a une origine de réplication unique sur son seul chromosome (comme c'est le cas pour la plupart des procaryotes), elle mesure environ 245 paires de bases et est riche en séquences AT. L'origine de la réplication est reconnue par certaines protéines qui se lient à ce site. Une enzyme appelée hélicase détend l'ADN en rompant les liaisons hydrogène entre les paires de bases azotées. L'hydrolyse de l'ATP est requise pour ce processus. Lorsque l'ADN s'ouvre, des structures en forme de Y appelées fourches de réplication se forment. Deux fourches de réplication sont formées à l'origine de la réplication et s'étendent de manière bidirectionnelle au fur et à mesure de la réplication. Les protéines de liaison monocaténaires recouvrent les brins simples de l'ADN à proximité de la fourche de réplication pour empêcher l'ADN monocaténaire de redevenir une double hélice. L'ADN polymérase est capable d'ajouter des nucléotides uniquement dans la direction 5' à 3' (un nouveau brin d'ADN ne peut être étendu que dans cette direction). Il a également besoin d'un groupe 3'-OH libre auquel il peut ajouter des nucléotides en formant une liaison phosphodiester entre l'extrémité 3'-OH et le phosphate 5' du nucléotide suivant. Cela signifie essentiellement qu'il ne peut pas ajouter de nucléotides si aucun groupe 3'-OH libre n'est disponible. Alors, comment ajoute-t-il le premier nucléotide ? Le problème est résolu à l'aide d'une amorce qui fournit l'extrémité 3'-OH libre. Une autre enzyme, l'ARN primase, synthétise une amorce d'ARN longue d'environ cinq à dix nucléotides et complémentaire à l'ADN. Comme cette séquence amorce la synthèse de l'ADN, elle est appelée à juste titre amorce. L'ADN polymérase peut désormais étendre cette amorce d'ARN, en ajoutant un par un des nucléotides complémentaires au brin matrice (Figure\(\PageIndex{1}\)).
Exercice\(\PageIndex{1}\)
Vous isolez une souche cellulaire dans laquelle la liaison entre des fragments d'Okazaki est altérée et vous soupçonnez qu'une mutation s'est produite dans une enzyme présente au niveau de la fourche de réplication. Quelle enzyme est la plus susceptible d'être mutée ?
- Réponse
-
ADN ligase, car cette enzyme réunit des fragments d'Okazaki.
La fourche de réplication se déplace au rythme de 1000 nucléotides par seconde. L'ADN polymérase ne peut s'étendre que dans la direction 5' à 3', ce qui pose un léger problème au niveau de la fourche de réplication. Comme nous le savons, la double hélice de l'ADN est antiparallèle, c'est-à-dire qu'un brin est orienté dans la direction 5' à 3' et l'autre dans la direction 3' à 5'. Un brin, complémentaire du brin d'ADN parental 3' à 5', est synthétisé en continu vers la fourche de réplication car la polymérase peut ajouter des nucléotides dans cette direction. Ce brin synthétisé en continu est connu sous le nom de brin principal. L'autre brin, complémentaire de l'ADN parental de 5' à 3', est étendu loin de la fourche de réplication, en petits fragments appelés fragments d'Okazaki, chacun nécessitant une amorce pour démarrer la synthèse. Les fragments d'Okazaki portent le nom du scientifique japonais qui les a découverts pour la première fois. Le fil contenant les fragments d'Okazaki est connu sous le nom de fil en retard.
Le brin principal peut être prolongé par une seule amorce, tandis que le brin en retard a besoin d'un nouvel apprêt pour chacun des courts fragments d'Okazaki. La direction générale du brin en retard sera de 3' à 5', et celle du brin avant de 5' à 3'. Une protéine appelée pince coulissante maintient l'ADN polymérase en place alors qu'elle continue à ajouter des nucléotides. La pince coulissante est une protéine en forme d'anneau qui se lie à l'ADN et maintient la polymérase en place. La topoisomérase empêche le débordement de la double hélice de l'ADN avant la fourche de réplication lorsque l'ADN s'ouvre ; elle le fait en provoquant des entailles temporaires dans l'hélice d'ADN, puis en la refermant. Au fur et à mesure de la synthèse, les amorces d'ARN sont remplacées par l'ADN. Les amorces sont éliminées par l'activité exonucléasique de l'ADN pol I, et les lacunes sont comblées par des désoxyribonucléotides. Les entailles qui subsistent entre l'ADN nouvellement synthétisé (qui a remplacé l'amorce d'ARN) et l'ADN précédemment synthétisé sont scellées par l'enzyme ADN ligase qui catalyse la formation d'une liaison phosphodiester entre l'extrémité 3'-OH d'un nucléotide et l'extrémité phosphate 5' de l'autre fragment.
Une fois que le chromosome a été complètement répliqué, les deux copies d'ADN se déplacent dans deux cellules différentes au cours de la division cellulaire. Le processus de réplication de l'ADN peut être résumé comme suit.
Étapes de réplication de
- L'ADN se déroule à l'origine de la réplication.
- L'hélicase ouvre les fourches de réplication formant l'ADN ; celles-ci sont étendues de manière bidirectionnelle.
- Des protéines de liaison à brin unique recouvrent l'ADN autour de la fourche de réplication pour empêcher le rembobinage de l'ADN
- La topoisomérase se lie à la région située en avant de la fourche de réplication pour empêcher le superenroulement.
- La primase synthétise des amorces d'ARN complémentaires au brin d'ADN.
- L'ADN polymérase commence à ajouter des nucléotides à l'extrémité 3'-OH de l'amorce.
- L'allongement du brin en retard et du brin avant se poursuit.
- Les amorces à ARN sont éliminées par activité exonucléasique.
- Les lacunes sont comblées par l'ADN pol en ajoutant des dNTP.
- L'espace entre les deux fragments d'ADN est scellé par l'ADN ligase, qui contribue à la formation de liaisons phosphodiesters.
\(\PageIndex{1}\)Le tableau résume les enzymes impliquées dans la réplication de l'ADN procaryote et les fonctions de chacune.
| Enzyme/protéine | Fonction spécifique |
|---|---|
| Pôle ADN II | L'activité de l'exonucléase élimine l'amorce de l'ARN et la remplace par de l'ADN |
| ADN pol II | Fonction de réparation |
| ADN pol III | Enzyme principale qui ajoute des nucléotides dans la direction 5'-3' |
| Hélicase | Ouvre l'hélice de l'ADN en rompant les liaisons hydrogène entre les bases azotées |
| Ligase | Comble les espaces entre les fragments d'Okazaki pour créer un brin d'ADN continu |
| Primase | Synthétise les amorces d'ARN nécessaires au démarrage de la réplication |
| Pince coulissante | Aide à maintenir l'ADN polymérase en place lorsque des nucléotides sont ajoutés |
| Topoisomérase | Aide à soulager le stress sur l'ADN lors du déroulement en provoquant des cassures puis en refermant l'ADN |
| Protéines de liaison à brin unique (SSB) | Se lie à l'ADN monocaténaire pour éviter le retour en arrière de l'ADN. |
Lien vers l'apprentissage
Passez en revue le processus complet de réplication de l'ADN ici.
Résumé
La réplication chez les procaryotes commence à partir d'une séquence trouvée sur le chromosome appelée origine de la réplication, c'est-à-dire le point où l'ADN s'ouvre. L'hélicase ouvre la double hélice de l'ADN, ce qui entraîne la formation de la fourche de réplication. Les protéines de liaison monocaténaires se lient à l'ADN monocaténaire à proximité de la fourche de réplication pour maintenir celle-ci ouverte. La primase synthétise une amorce d'ARN pour initier la synthèse par l'ADN polymérase, qui ne peut ajouter des nucléotides que dans la direction 5' à 3'. Un brin est synthétisé en continu en direction de la fourche de réplication ; c'est ce que l'on appelle le brin principal. L'autre brin est synthétisé dans une direction opposée à la fourche de réplication, dans de courtes portions d'ADN appelées fragments d'Okazaki. Ce brin est connu sous le nom de brin en retard. Une fois la réplication terminée, les amorces d'ARN sont remplacées par des nucléotides d'ADN et l'ADN est scellé avec de l'ADN ligase, qui crée des liaisons phosphodiester entre le 3'-OH d'une extrémité et le phosphate 5' de l'autre brin.
Lexique
- hélicase
- lors de la réplication, cette enzyme aide à ouvrir l'hélice de l'ADN en rompant les liaisons hydrogène
- brin en retard
- pendant la réplication, le brin qui est répliqué en fragments courts et loin de la fourchette de réplication
- brin principal
- brin synthétisé en continu dans la direction 5'-3' qui est synthétisé dans la direction de la fourche de réplication
- ligase
- enzyme qui catalyse la formation d'une liaison phosphodiester entre les extrémités 3' OH et 5' phosphate de l'ADN
- Fragment d'Okazaki
- Fragment d'ADN synthétisé sur de courtes distances sur le brin en retard
- primase
- enzyme qui synthétise l'amorce d'ARN ; l'amorce est nécessaire à l'ADN pol pour démarrer la synthèse d'un nouveau brin d'ADN
- apprêt
- courte portion de nucléotides nécessaire pour initier la réplication ; dans le cas de la réplication, l'amorce contient des nucléotides d'ARN
- fourche de réplication
- Structure en Y formée lors du début de la réplication
- protéine de liaison à brin unique
- lors de la réplication, protéine qui se lie à l'ADN monocaténaire ; cela aide à séparer les deux brins d'ADN afin qu'ils puissent servir de modèles
- pince coulissante
- protéine en forme d'anneau qui maintient le pôle d'ADN sur le brin d'ADN
- topoisomérase
- enzyme qui provoque un enroulement ou un retournement de l'ADN lors de la réplication de l'ADN


