15.5: الريبوسومات وتخليق البروتين
- Page ID
- 196622
المهارات اللازمة للتطوير
- وصف الخطوات المختلفة في تخليق البروتين
- ناقش دور الريبوسومات في تخليق البروتين
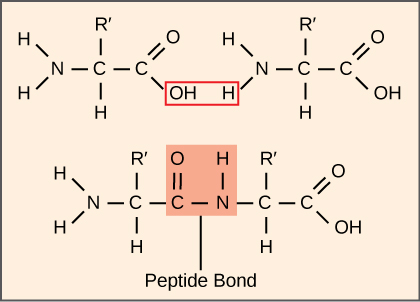
يستهلك تخليق البروتينات طاقة الخلية أكثر من أي عملية أيضية أخرى. في المقابل، تمثل البروتينات كتلة أكبر من أي مكون آخر من الكائنات الحية (باستثناء الماء)، وتؤدي البروتينات تقريبًا كل وظيفة من وظائف الخلية. تتضمن عملية الترجمة، أو تخليق البروتين، فك تشفير رسالة mRNA إلى منتج متعدد الببتيد. يتم ربط الأحماض الأمينية معًا بشكل تساهمي عن طريق ربط روابط الببتيد بأطوال تتراوح من حوالي 50 من بقايا الأحماض الأمينية إلى أكثر من 1000. يحتوي كل حمض أميني فردي على مجموعة أمينية (NH 2) ومجموعة كربوكسيل (COOH). تتشكل الببتيدات المتعددة عندما تشكل المجموعة الأمينية لأحد الأحماض الأمينية رابطة أميد (أي الببتيد) مع مجموعة الكربوكسيل لحمض أميني آخر (الشكل\(\PageIndex{1}\)). يتم تحفيز هذا التفاعل بواسطة الريبوسومات ويولد جزيء ماء واحد.
آلات تخليق البروتين
بالإضافة إلى قالب mRNA، تساهم العديد من الجزيئات والجزيئات الكبيرة في عملية الترجمة. قد يختلف تكوين كل مكون باختلاف الأنواع؛ على سبيل المثال، قد تتكون الريبوسومات من أعداد مختلفة من الرنا الريباسي والببتيدات المتعددة اعتمادًا على الكائن الحي. ومع ذلك، فإن الهياكل والوظائف العامة لآلات تخليق البروتين قابلة للمقارنة من البكتيريا إلى الخلايا البشرية. تتطلب الترجمة إدخال قالب mRNA، والريبوسومات، والترانا، والعوامل الأنزيمية المختلفة.
الريبوسومات
حتى قبل ترجمة mRNA، يجب أن تستثمر الخلية الطاقة لبناء كل من الريبوسومات الخاصة بها. في E. coli، يوجد ما بين 10000 و 70000 ريبوسوم في كل خلية في أي وقت. الريبوسوم عبارة عن جزيء كبير معقد يتكون من الحمض الريبي الريبوزي الهيكلي والتحفيزي والعديد من الببتيدات المتعددة المتميزة. في حقيقيات النوى، تكون النواة متخصصة تمامًا في تخليق وتجميع الحمض الريبي الريبي.
توجد الريبوسومات في السيتوبلازم في بدائيات النواة وفي السيتوبلازم والشبكة الإندوبلازمية الخشنة في حقيقيات النوى. تحتوي الميتوكوندريا والكلوروبلاستيدات أيضًا على ريبوسومات خاصة بها في المصفوفة والسدى، والتي تبدو أكثر تشابهًا مع الريبوسومات بدائية النواة (ولديها حساسيات دوائية مماثلة) من الريبوسومات الموجودة خارج أغشيتها الخارجية في السيتوبلازم. تتفكك الريبوسومات إلى وحدات فرعية كبيرة وصغيرة عندما لا تقوم بتجميع البروتينات وإعادة الارتباط أثناء بدء الترجمة. في E. coli، توصف الوحدة الفرعية الصغيرة بـ 30S، والوحدة الفرعية الكبيرة هي 50S، ليصبح المجموع 70S (تذكر أن وحدات Svedberg ليست مضافة). تحتوي ريبوسومات الثدييات على وحدة فرعية صغيرة من 40S ووحدة فرعية كبيرة من 60S، ليصبح المجموع 80S. الوحدة الفرعية الصغيرة مسؤولة عن ربط قالب mRNA، في حين أن الوحدة الفرعية الكبيرة تربط TrNAs بالتتابع. تتم ترجمة كل جزيء من mRNA في وقت واحد بواسطة العديد من الريبوسومات، وكلها تقوم بتجميع البروتين في نفس الاتجاه: قراءة mRNA من 5 إلى 3 «وتوليف البولي ببتيد من نهاية N إلى نهاية C. يسمى هيكل MRNA/Poly-ribosome الكامل بالبوليسوم.
الحمض النووي الريبي
الحمض النووي الريبي الريبي هو جزيئات RNA هيكلية تم نسخها من الجينات بواسطة بوليميراز RNA III. اعتمادًا على الأنواع، يوجد 40 إلى 60 نوعًا من الحمض النووي الريبي في السيتوبلازم. تعمل كمحولات، وترتبط الحمض الريبي النووي الريبي المحدد بالتسلسلات الموجودة على قالب mRNA وتضيف الأحماض الأمينية المقابلة إلى سلسلة البولي ببتيد. لذلك، فإن الحمض النووي الريبي هو الجزيئات التي «تترجم» بالفعل لغة الحمض النووي الريبي إلى لغة البروتينات.
من بين 64 كودون mRNA ممكنًا - أو تركيبات ثلاثية من A و U و G و C - تحدد ثلاثة منها إنهاء تخليق البروتين وتحدد 61 إضافة الأحماض الأمينية إلى سلسلة البولي ببتيد. من بين هذه الـ 61، يقوم رمز واحد (AUG) أيضًا بتشفير بدء الترجمة. يمكن لكل مضاد من مضادات الحمض النووي الريبي أن يستند إلى أحد أكواد mRNA وإضافة حمض أميني أو إنهاء الترجمة، وفقًا للشفرة الجينية. على سبيل المثال، إذا حدث تسلسل CUA على قالب mRNA في إطار القراءة المناسب، فسيؤدي ذلك إلى ربط الحمض الريبي الريبي الذي يعبر عن التسلسل التكميلي، GAU، والذي سيتم ربطه بلوسين الأحماض الأمينية.
كجزيئات محول للترجمة، من المدهش أن الحمض النووي الريبي يمكن أن يلائم الكثير من الخصوصية في مثل هذه الحزمة الصغيرة. ضع في اعتبارك أن الحمض النووي الريبي يحتاج إلى التفاعل مع ثلاثة عوامل: 1) يجب التعرف عليها من خلال سينثيتاز الأمينوسيل الصحيح (انظر أدناه)؛ 2) يجب التعرف عليها بواسطة الريبوسومات؛ و 3) يجب أن ترتبط بالتسلسل الصحيح في mRNA.
تخليق الحمض النووي الريبي أمينوسيل
إن عملية تخليق الحمض النووي الريبي قبل الحمض النووي الريبي بواسطة بوليميراز الحمض النووي الريبي III تخلق فقط جزء RNA من جزيء المحول. يجب إضافة الحمض الأميني المقابل لاحقًا، بمجرد معالجة الحمض الريبي النووي الريبي وتصديره إلى السيتوبلازم. من خلال عملية «شحن» الحمض النووي الريبي، يتم ربط كل جزيء من الحمض الأميني الصحيح الخاص به من خلال مجموعة من الإنزيمات تسمى تخليق الحمض النووي الريبي الأميني. يوجد نوع واحد على الأقل من تخليق الحمض النووي الريبي الأميني لكل من الأحماض الأمينية العشرين؛ ويختلف العدد الدقيق لمركب الحمض الريبي الريبي الأميني حسب الأنواع. تقوم هذه الإنزيمات أولاً بربط ATP وتحلله لتحفيز رابطة عالية الطاقة بين الأحماض الأمينية ومونوزين أحادي الفوسفات (AMP)؛ يتم طرد جزيء البيروفوسفات في هذا التفاعل. ثم يتم نقل الحمض الأميني المنشط إلى الحمض النووي الريبي، ويتم إطلاق AMP.
آلية تخليق البروتين
كما هو الحال مع تخليق mRNA، يمكن تقسيم تخليق البروتين إلى ثلاث مراحل: البدء والاستطالة والإنهاء. تتشابه عملية الترجمة في بدائيات النواة وحقائق النوى. سنستكشف هنا كيفية حدوث الترجمة في E. coli، وهي مقدمة نموذجية، ونحدد أي اختلافات بين الترجمة بدائية النواة وترجمة حقيقية النواة.
بدء الترجمة
يبدأ تخليق البروتين بتكوين مجمع البدء. في الإشريكية القولونية، يتضمن هذا المركب ريبوسوم 30S الصغير، ونموذج mRNA، وثلاثة عوامل بدء (IFs؛ IF-1، IF-2، و IF-3)، وبادئ خاص بـ trNA، يسمى\(\text{tRNA}_\text{f}^\text{Met}\). The initiator tRNA interacts with the start codon AUG (or rarely, GUG), links to a formylated methionine called fMet, and can also bind IF-2. Formylated methionine is inserted by \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) at the beginning of every polypeptide chain synthesized by E. coli, but it is usually clipped off after translation is complete. When an in-frame AUG is encountered during translation elongation, a non-formylated methionine is inserted by a regular Met-tRNAMet.
In E. coli mRNA, a sequence upstream of the first AUG codon, called the Shine-Dalgarno sequence (AGGAGG), interacts with the rRNA molecules that compose the ribosome. This interaction anchors the 30S ribosomal subunit at the correct location on the mRNA template. Guanosine triphosphate (GTP), which is a purine nucleotide triphosphate, acts as an energy source during translation—both at the start of elongation and during the ribosome’s translocation.
In eukaryotes, a similar initiation complex forms, comprising mRNA, the 40S small ribosomal subunit, IFs, and nucleoside triphosphates (GTP and ATP). The charged initiator tRNA, called Met-tRNAi, does not bind fMet in eukaryotes, but is distinct from other Met-tRNAs in that it can bind IFs.
Instead of depositing at the Shine-Dalgarno sequence, the eukaryotic initiation complex recognizes the 7-methylguanosine cap at the 5' end of the mRNA. A cap-binding protein (CBP) and several other IFs assist the movement of the ribosome to the 5' cap. Once at the cap, the initiation complex tracks along the mRNA in the 5' to 3' direction, searching for the AUG start codon. Many eukaryotic mRNAs are translated from the first AUG, but this is not always the case. According to Kozak’s rules, the nucleotides around the AUG indicate whether it is the correct start codon. Kozak’s rules state that the following consensus sequence must appear around the AUG of vertebrate genes: 5'-gccRccAUGG-3'. The R (for purine) indicates a site that can be either A or G, but cannot be C or U. Essentially, the closer the sequence is to this consensus, the higher the efficiency of translation.
Once the appropriate AUG is identified, the other proteins and CBP dissociate, and the 60S subunit binds to the complex of Met-tRNAi, mRNA, and the 40S subunit. This step completes the initiation of translation in eukaryotes.
Translation, Elongation, and Termination
In prokaryotes and eukaryotes, the basics of elongation are the same, so we will review elongation from the perspective of E. coli. The 50S ribosomal subunit of E. coli consists of three compartments: the A (aminoacyl) site binds incoming charged aminoacyl tRNAs. The P (peptidyl) site binds charged tRNAs carrying amino acids that have formed peptide bonds with the growing polypeptide chain but have not yet dissociated from their corresponding tRNA. The E (exit) site releases dissociated tRNAs so that they can be recharged with free amino acids. There is one exception to this assembly line of tRNAs: in E. coli, \(\text{fMet} - \text{tRNA}_\text{f}^\text{Met}\) is capable of entering the P site directly without first entering the A site. Similarly, the eukaryotic Met-tRNAi, with help from other proteins of the initiation complex, binds directly to the P site. In both cases, this creates an initiation complex with a free A site ready to accept the tRNA corresponding to the first codon after the AUG.
During translation elongation, the mRNA template provides specificity. As the ribosome moves along the mRNA, each mRNA codon comes into register, and specific binding with the corresponding charged tRNA anticodon is ensured. If mRNA were not present in the elongation complex, the ribosome would bind tRNAs nonspecifically.
Elongation proceeds with charged tRNAs entering the A site and then shifting to the P site followed by the E site with each single-codon “step” of the ribosome. Ribosomal steps are induced by conformational changes that advance the ribosome by three bases in the 3' direction. The energy for each step of the ribosome is donated by an elongation factor that hydrolyzes GTP. Peptide bonds form between the amino group of the amino acid attached to the A-site tRNA and the carboxyl group of the amino acid attached to the P-site tRNA. The formation of each peptide bond is catalyzed by peptidyl transferase, an RNA-based enzyme that is integrated into the 50S ribosomal subunit. The energy for each peptide bond formation is derived from GTP hydrolysis, which is catalyzed by a separate elongation factor. The amino acid bound to the P-site tRNA is also linked to the growing polypeptide chain. As the ribosome steps across the mRNA, the former P-site tRNA enters the E site, detaches from the amino acid, and is expelled (Figure \(\PageIndex{2}\)). Amazingly, the E. coli translation apparatus takes only 0.05 seconds to add each amino acid, meaning that a 200-amino acid protein can be translated in just 10 seconds.
Art Connection
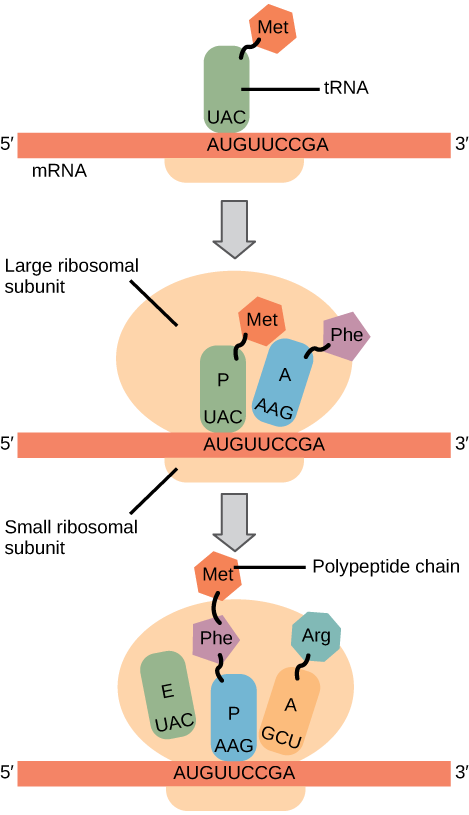
Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Termination of translation occurs when a nonsense codon (UAA, UAG, or UGA) is encountered. Upon aligning with the A site, these nonsense codons are recognized by release factors in prokaryotes and eukaryotes that instruct peptidyl transferase to add a water molecule to the carboxyl end of the P-site amino acid. This reaction forces the P-site amino acid to detach from its tRNA, and the newly made protein is released. The small and large ribosomal subunits dissociate from the mRNA and from each other; they are recruited almost immediately into another translation initiation complex. After many ribosomes have completed translation, the mRNA is degraded so the nucleotides can be reused in another transcription reaction.
Protein Folding, Modification, and Targeting
During and after translation, individual amino acids may be chemically modified, signal sequences may be appended, and the new protein “folds” into a distinct three-dimensional structure as a result of intramolecular interactions. A signal sequence is a short tail of amino acids that directs a protein to a specific cellular compartment. These sequences at the amino end or the carboxyl end of the protein can be thought of as the protein’s “train ticket” to its ultimate destination. Other cellular factors recognize each signal sequence and help transport the protein from the cytoplasm to its correct compartment. For instance, a specific sequence at the amino terminus will direct a protein to the mitochondria or chloroplasts (in plants). Once the protein reaches its cellular destination, the signal sequence is usually clipped off.
Many proteins fold spontaneously, but some proteins require helper molecules, called chaperones, to prevent them from aggregating during the complicated process of folding. Even if a protein is properly specified by its corresponding mRNA, it could take on a completely dysfunctional shape if abnormal temperature or pH conditions prevent it from folding correctly.
Summary
The players in translation include the mRNA template, ribosomes, tRNAs, and various enzymatic factors. The small ribosomal subunit forms on the mRNA template either at the Shine-Dalgarno sequence (prokaryotes) or the 5' cap (eukaryotes). Translation begins at the initiating AUG on the mRNA, specifying methionine. The formation of peptide bonds occurs between sequential amino acids specified by the mRNA template according to the genetic code. Charged tRNAs enter the ribosomal A site, and their amino acid bonds with the amino acid at the P site. The entire mRNA is translated in three-nucleotide “steps” of the ribosome. When a nonsense codon is encountered, a release factor binds and dissociates the components and frees the new protein. Folding of the protein occurs during and after translation.
Art Connections
Figure \(\PageIndex{2}\): Many antibiotics inhibit bacterial protein synthesis. For example, tetracycline blocks the A site on the bacterial ribosome, and chloramphenicol blocks peptidyl transfer. What specific effect would you expect each of these antibiotics to have on protein synthesis?
Tetracycline would directly affect:
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
Chloramphenicol would directly affect
- tRNA binding to the ribosome
- ribosome assembly
- growth of the protein chain
- Answer
-
Tetracycline: a; Chloramphenicol: c.
Glossary
- aminoacyl tRNA synthetase
- enzyme that “charges” tRNA molecules by catalyzing a bond between the tRNA and a corresponding amino acid
- initiator tRNA
- in prokaryotes, called \(\text{tRNA}_\text{f}^\text{Met}\); in eukaryotes, called tRNAi; a tRNA that interacts with a start codon, binds directly to the ribosome P site, and links to a special methionine to begin a polypeptide chain
- Kozak’s rules
- determines the correct initiation AUG in a eukaryotic mRNA; the following consensus sequence must appear around the AUG: 5’-GCC(purine)CCAUGG-3’; the bolded bases are most important
- peptidyl transferase
- RNA-based enzyme that is integrated into the 50S ribosomal subunit and catalyzes the formation of peptide bonds
- polysome
- mRNA molecule simultaneously being translated by many ribosomes all going in the same direction
- Shine-Dalgarno sequence
- (AGGAGG); initiates prokaryotic translation by interacting with rRNA molecules comprising the 30S ribosome
- signal sequence
- short tail of amino acids that directs a protein to a specific cellular compartment
- start codon
- AUG (or rarely, GUG) on an mRNA from which translation begins; always specifies methionine



